RIBO-seq En Bacterias: Una Colección de Muestras Y Protocolo De Preparación De La Biblioteca Para La Secuenciación NGS
Summary
Aquí se describen las etapas de la recolección de muestras y la preparación de RIBO-seq en bacterias. La secuenciación de las bibliotecas preparadas de acuerdo con estas directrices da como resultado datos suficientes para un análisis bioinformático exhaustivo. El protocolo que presentamos es simple, utiliza equipos de laboratorio estándar y tarda siete días desde la lisis hasta la obtención de bibliotecas.
Abstract
La técnica de perfilado de ribosomas (RIBO-seq) es actualmente la herramienta más eficaz para estudiar el proceso de síntesis de proteínas in vivo. La ventaja de este método, en comparación con otros enfoques, es su capacidad para monitorear la traducción mediante el mapeo preciso de la posición y el número de ribosomas en una transcripción de ARNm.
En este artículo, se describen las etapas consecutivas de la recolección de muestras y la preparación para el método RIBO-seq en bacterias, destacando los detalles relevantes para la planificación y ejecución del experimento.
Puesto que el RIBO-seq confía en los ribosomas intactos y los mRNAs relacionados, el paso dominante es la inhibición rápida de la traducción y la desintegración adecuada de células. Por lo tanto, sugerimos la filtración y la congelación flash en nitrógeno líquido para la recolección celular con un pretratamiento opcional con cloranfenicol para detener la traducción en bacterias. Para la desintegración, proponemos moler las células congeladas con mortero y pestle en presencia de óxido de aluminio para alterar mecánicamente la pared celular. En este protocolo, no se requiere un cojín de sacarosa o una ultracentrifugación de gradiente de sacarosa para la purificación del monosoma. En lugar, la separación del mRNA usando la electroforesis del gel de la poliacrilamida (PÁGINA) seguida por la supresión ribosómica de la huella (venda de 28-30 nt) se aplica y proporciona resultados satisfactorios. Esto simplifica en gran medida el método, así como reduce los requisitos de tiempo y equipo para el procedimiento. Para la preparación de la biblioteca, recomendamos usar el pequeño kit de ARN disponible comercialmente para la secuenciación de Illumina de New England Biolabs, siguiendo las pautas del fabricante con cierto grado de optimización.
Las bibliotecas de ADNc resultantes presentan la cantidad y calidad adecuadas requeridas para la secuenciación de próxima generación (NGS). La secuenciación de las bibliotecas preparadas de acuerdo con el protocolo descrito da como resultado de 2 a 10 mln lecturas mapeadas de forma única por muestra, lo que proporciona datos suficientes para un análisis bioinformático exhaustivo. El protocolo que presentamos es rápido y relativamente fácil y se puede realizar con equipos de laboratorio estándar.
Introduction
La técnica de perfilado de ribosomas (RIBO-seq) fue desarrollada en el laboratorio de Jonathan Weissman de la Universidad de California, San Francisco1. En comparación con otros métodos utilizados para estudiar la expresión génica a nivel traslacional, RIBO-seq se centra en cada ribosoma que se une al ARNm y proporciona información sobre su ubicación y el número relativo de ribosomas en una transcripción. Permite monitorizar el proceso de síntesis de proteínas in vivo y puede proporcionar resolución y precisión de un solo codón permitiendo la medición de la densidad del ribosoma tanto en el ARNm individual como a lo largo de todo el transcriptoma de la célula. En la fundación de la técnica RIBO-seq se encuentra el hecho de que durante la traducción el ribosoma se une a la molécula de ARNm y por lo tanto protege el fragmento enterrado de la transcripción de una digestión ribonucleasa. Sobre la adición de la ribonucleasa, el mRNA desprotegido se digiere y los fragmentos encerrados por los ribosomas – típicamente de ~28-30 nt de largo – permanecen intactos. Estos fragmentos, llamados huellas ribosómicas (RF), pueden ser aislados, secuenciados y mapeados en la transcripción de la que se originaron, lo que resulta en la detección de la posición exacta de los ribosomas. De hecho, la capacidad del ribosoma para proteger los fragmentos de ARNm se ha utilizado desde la década de 1960 para estudiar la unión ribosómica y los sitios de iniciación a la traducción (TIS)2,3,4. Sin embargo, con el avance en la tecnología de secuenciación profunda, RIBO-seq se ha convertido en un estándar de oro para el monitoreo de la traducción5 que, a través del compromiso del ribosoma, puede proporcionar una información de todo el genoma sobre la síntesis de proteínas6. El perfilado de ribosomas llenó el vacío tecnológico que existía entre la cuantificación del transcriptoma y elproteoma 6.
Para llevar a cabo el perfilado de ribosomas necesitamos obtener el lisoto celular del organismo que había crecido en las condiciones investigadas. La interrupción de estas condiciones durante la recolección y lisis celular puede proporcionar datos poco confiables. Para prevenir esto, los inhibidores de la traducción, la cosecha rápida y la congelación repentina en nitrógeno líquido se utilizan comúnmente. Las células pueden ser lisadas por molienda criogénica en un homogeneizador mecánico como un molino mezclador7,8 o un batidor de perlas9,y por trituración a través de una pipeta10 o con una aguja11. El tampón de lisis se puede agregar justo antes o poco después de la pulverización de las células. En nuestro protocolo utilizamos nitrógeno líquido para preenfriar mortero y pestle, así como óxido de aluminio como un enfoque más suave para la interrupción de la pared celular bacteriana, lo que evita la cizalladura de ARN que a menudo se encuentra cuando se aplican métodos como la sonificación. Después de la pulverización, añadimos un tampón de lisis helada en el contenido enfriado del mortero. La selección de un tampón de lisis adecuado es importante para obtener la mejor resolución de las huellas ribosómicas. Dado que la resistencia iónica afecta tanto al tamaño de RF como a la precisión del marco de lectura, actualmente se recomienda utilizar tampones de lisis con baja resistencia iónica y capacidad de tampón, incluso si parece que la composición del tampón no afecta a la ocupación ribosómica en arns11,12. Los componentes importantes del tampón de lisis son los iones de magnesio, cuya presencia impide la disociación de las subunidades ribosómicas e inhibe los cambios conformacionales espontáneos en los ribosomas bacterianos11,13. Los iones de calcio también juegan un papel importante y son esenciales para la actividad de la nucleasa microcócica (MNase) utilizada en el método de perfilado de ribosomas bacterianos14. La adición de guanosina 5′-[β,γ-imido]trifosfato (GMP-PNP), un análogo no hidrolizable de GTP, junto con cloranfenicol inhibe la traducción durante la lisis15.
Cuando se obtiene el lisato, se clarifica por centrifugación y se divide en dos porciones, cada una para un RIBO-seq y una secuenciación de ARNm total de alto rendimiento (ARN-seq) ya que se realizan simultáneamente (Figura 1). El ARN-seq proporciona un punto de referencia que permite la comparación de datos tanto de RIBO-seq como de ARN-seq durante el análisis de datos. El translatoma investigado se define por la normalización de las huellas ribosómicas a la abundancia deARNm 16. Los datos de RNA-seq también pueden ayudar a identificar artefactos de clonación o secuenciación17.
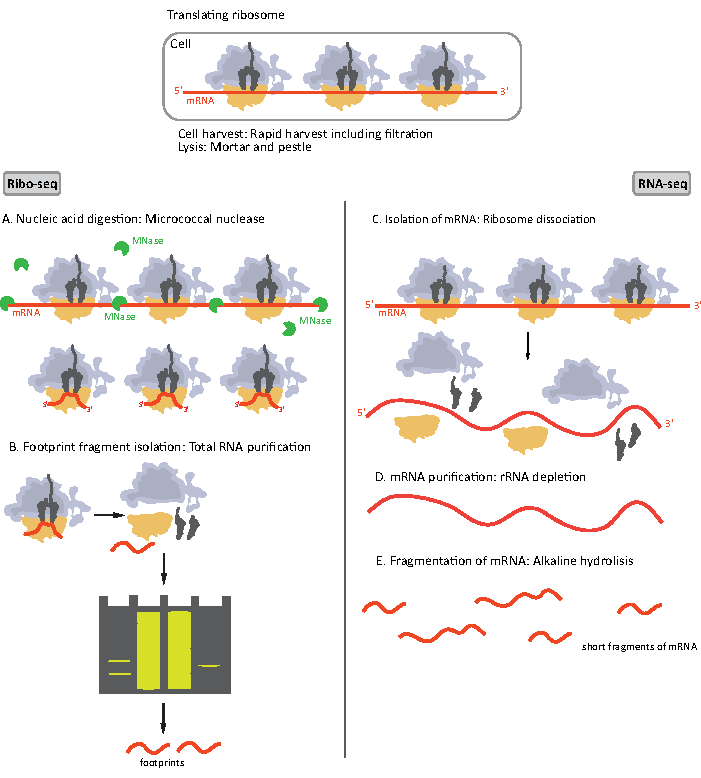
Figura 1. Schematics of mRNA sample preparation for RIBO-seq and RNA-seq. Para la preparación de la biblioteca RIBO-seq, el ARN se digiere con MNase (A), seguido por la selección del tamaño de RF de ~28-30 nt de longitud (B); para el ARN-seq el ARN se aísla (c), agotado del rRNA (d), y el mRNA resultante se hace fragmentos aleatoriamente en los fragmentos de longitudes diversas (e). Haga clic aquí para ver una versión más amplia de esta figura.
Los pasos iniciales del procedimiento de preparación de muestras para RIBO-seq y ARN-seq difieren ligeramente (Figura 1). Para el perfilación ribosómica, el lisoto necesita ser digerido por una endonucleasa específica para degradar las moléculas de ARNm no protegidas por los ribosomas. En los protocolos estándar, los monosomas obtenidos son recuperados mediante una ultracentrifugación de cojín de sacarosa o una ultracentrifugación de gradiente de sacarosa8,14. En este artículo, mostramos que este paso no es necesario para aislar la RF requerida para el RIBO-seq en bacterias, así mismo para las células eucariotas18,y que la selección del tamaño de los fragmentos de ARNm de longitud adecuada del gel de poliacrilamida es suficiente.
Para el ARN-seq, el mRNA es obtenido por el agotamiento del rRNA del ARN total – las moléculas del rRNA hibridan a las puntas de prueba biotinylated del oligonucleótido que atan a los perlas magnéticos streptavidin-revestidos. Los complejos rRNA-oligonucleótido-perlas se eliminan de la muestra con un imán dando como resultado una muestra agotada de ARNr19,20. Las moléculas purificadas del mRNA entonces son fragmentadas aleatoriamente por hidrólisis alcalina. Los fragmentos obtenidos de ARNm, así como las huellas ribosómicas se convierten en bibliotecas de ADNc y se preparan para la secuenciación profunda (Figura 2). Esto implica la reparación de los extremos necesarios después de la hidrólisis alcalina (para el mRNA) y de la digestión enzimática (para el RF): dephosphorylation de los extremos 3′ seguidos por la fosforilación de los extremos de 5′. Los siguientes pasos son la ligadura de adaptadores y la transcripción inversa para crear insertos de ADNc enmarcados por secuencias requeridas para la secuenciación de próxima generación (NGS) utilizando la plataforma Illumina. La última fase de la preparación de la biblioteca es una reacción de PCR en la que las construcciones se amplifican y etiquetan con códigos de barras específicos de la muestra para permitir la multiplexación y secuenciación de varias muestras en un canal. Antes de la secuenciación, la calidad y la cantidad de las bibliotecas se evalúan mediante la electroforesis en chip de ADN de alta sensibilidad. Las bibliotecas de ADNc con los parámetros adecuados se pueden agrupar y secuenciar. La secuenciación se puede realizar en diferentes plataformas Illumina, como MiSeq, NextSeq o HighSeq, dependiendo del número de bibliotecas, la longitud de lectura requerida y la profundidad de secuenciación. Después de la secuenciación, se realiza el análisis bioinformático.

Figura 2. Preparación de la biblioteca. La preparación de la biblioteca incluye la reparación de los extremos, la ligadura de los adaptadores, la transcripción inversa y la amplificación con código de barras. Haga clic aquí para ver una versión más amplia de esta figura.
El perfilado de ribosomas es un método universal que puede modificarse y ajustarse fácilmente de acuerdo con la pregunta científica. Originalmente se utilizaba en la levadura1,pero poco después se aplicó a las células bacterianas21, así como a organismos modelo eucariotas como el ratón10,el pez cebra22,la mosca de la fruta23 y Arabidopsis thaliana24. También se utilizó para el estudio de diferentes tipos de ribosomas: citoplasmático, mitocondrial25,26 y cloroplasto27,28. En eucariotas RIBO-seq es comúnmente adaptado y refinado para investigar aspectos específicos de la traducción, incluyendo la iniciación10,11,29,30,31,32,el alargamiento1,10,11,31, 33,ribosoma estancamiento33 y cambio de conformación33. La mayoría de las modificaciones implican el uso de diferentes inhibidores de la traducción. En bacterias sin embargo, estudios análogos han sido difíciles de realizar debido a la escasez de inhibidores con el mecanismo de acción requerido34. El inhibidor de la traducción más comúnmente utilizado en bacterias es el cloranfenicol (CAM) que se une al centro de la peptidil transferasa (PTC) e impide la correcta colocación del aminoacil-ARNt en el sitio A. Como resultado, cam previene la formación de un enlace peptídico que conduce a la detención de los ribosomas alargados35. Otros ejemplos de inhibidores de la traducción en bacterias son la tetraciclina (TET)36,la retapamulina (RET)34 y la Onc11237, que se han utilizado para investigar los sitios de iniciación a la traducción. La TET, que impide la entrega de ARNt al ribosoma mediante la superposición directa con el tallo-as del anticodón del ARNt en el sitio A, se aplicó originalmente para verificar los resultados obtenidos del tratamiento con CAM, ya que ambos son antibióticos que inhiben la elongación de la traducción38. Se encontró que TET detectaba TIS primario, sin embargo, no pudo revelar TIS36interno. Ret se une en el PTC del ribosoma bacteriano, y previene la formación del primer enlace peptídico interfiriendo con un aminoacil-ARNt elongador en el sitio A. La aplicación de RET da lugar a la detención de los ribosomas tanto en el TISs primario como en el interno34. Onc112, un péptido antimicrobiano prolina-rico, ata en el túnel de la salida y bloquea el atascamiento aminoacyl-tRNA en el sitio ribosomal de A. Como resultado, Onc112 evita que los complejos de iniciación entren en la fase de elongación37.
La principal información que proporciona el perfilado de ribosomas es la densidad de los ribosomas y su posición sobre el ARNm. Se aplicó con éxito para investigar la expresión génica diferencial a nivel de traslación en diversas condiciones de crecimiento1,6,medir la eficiencia traslacional1,38,39 y detectar eventos de regulación de la traducción como la pausa ribosómica10. RIBO-seq también permite descubrir la traducción de ncRNA anotado, pseudogenes y pequeños marcos de lectura abiertos no anotados (ORF) que conducen a la identificación de genes codificantes de proteínas nuevos y /o muy cortos10,12,22,30,37. En tales casos, RIBO-seq puede afinar y mejorar la anotación del genoma. Con su alta sensibilidad para la identificación de ORFs traducidos y su naturaleza cuantitativa, el perfilado de ribosomas también puede servir como un proxy para la determinación del proteoma o en la ayuda a los estudios de proteómica31,34,39. Mediante el mapeo de TIS, el perfilado de ribosomas revela isoformas N-terminalmente extendidas y truncadas de proteínas conocidas10,32. RIBO-seq también se adaptó para estudiar el plegamiento co-traslacional de proteínas14,21,24. Este método permite medir las tasas de elongación1,10,39 o velocidades de decodificación de codones individuales6 y ayuda en el desarrollo de modelos cuantitativos de traslación17. El método de perfilado de ribosomas también es capaz de proporcionar información mecanicista sobre la pausa del ribosoma en bacterias7,15,17,cambio de marco40,lectura de codones de parada21,defectos de terminación /reciclaje41,42 y cambios de conformación ribosómica33 en eucariotas. RIBO-seq también se adaptó para examinar el impacto de factores específicos de acción trans en la traducción, como los miRNAs6 y las proteínas de unión a ARN en eucariotas16,43. Sin embargo, es importante reconocer que el diseño experimental y la resolución obtenida de RIBO-seq determinan la cantidad de información que puede ser extraída de los datos de secuenciaciónresultantes 12.
Protocol
Representative Results
Discussion
El desafío técnico clave del perfilado de ribosomas es la necesidad de inhibir rápidamente la traducción para capturar una instantánea de ribosomas en ARNm en un estado fisiológico particular de interés. Para lograr esto, los inhibidores de la traducción, la cosecha rápida y la congelación flash en nitrógeno líquido se utilizan comúnmente. La aplicación de antibióticos es opcional, ya que pueden causar artefactos. El cloranfenicol es un fármaco de uso común para detener los ribosomas alargados en ribo-se…
Disclosures
The authors have nothing to disclose.
Acknowledgements
ALS desea agradecer el apoyo financiero de EMBO Installation Grants IG 3914 y POIR. 04.04.00-00-3E9C/17-00 realizado en el marco del programa First TEAM de la Fundación para la Ciencia Polaca cofinanciado por la Unión Europea con cargo al Fondo Europeo de Desarrollo Regional.
Materials
| 10X TBE (powder) | Invitrogen | AM9864 | |
| 2-Mercaptoethanol, 99%, pure | Acros Organics | 125472500 | |
| Adenosine 5'-Triphosphate (ATP) | New England Biolabs | P0756S | |
| Aluminium oxide calcinated pure p.a. | Chempur | 114560600 | |
| Calcium chloride dihydrate | Sigma-Aldrich | C3881-500G | |
| Chloramphenicol | MP Biomedicals | 190321 | |
| DNA Clean & Concentrator -5 | Zymo Research | D4004 | |
| Dnase I recombinant, Rnase-free | Roche | 4716728001 | |
| EDTA disodium salt | Fisher Scientific | E/P140/48 | |
| Ethyl Alcohol Absolut 99,8% Pure-P.A.-Basic | POCH Avantor Performance Materials Poland S.A | BA6480111 | |
| Filtration apparatus | VWR Collection | 511-0265 | all-glass filtration apparatus, with funnel, fritted base, cap, 47 mm Ø spring clamp and ground joint flask |
| Gel 40 (19:1) | Rotiphorese | 3030.1 | |
| Gel Loading Dye, Blue, 6X | New England Biolabs | E6138G | |
| Guanosine 5′-[β,γ-imido]triphosphate trisodium salt hydrate | Sigma-Aldrich | G0635-25MG | |
| labZAP | A&A Biotechnology | 040-500 | |
| Magnesium acetate tetrahydrate | Sigma-Aldrich | M5661-250G | |
| MCE membrane fiter | Alfatec Technology | M47MCE45GWS | pore size: 0.45um |
| MICROBExpress Bacterial mRNA Purification | Invitrogen | AM1905 | |
| Multiplex Small RNA Library Prep Set for Illumina | New England Biolabs | E7300S | |
| Nuclease-Free Water | Ambion | AM9937 | |
| Potassium Acetate Anhydrous Pure P.A. | POCH Avantor Performance Materials Poland S.A | 744330113 | |
| Quick-Load pBR322 DNA-MspI Digest | New England Biolabs | E7323A | |
| RNA Clean & Concentrator -25 | Zymo Research | R1018 | |
| Sodium acetate | Sigma-Aldrich | S2889-250G | |
| Sodium carbonate | Sigma-Aldrich | 223530-500G | |
| Sodium hydrogen carbonate pure p.a. | POCH Avantor Performance Materials Poland S.A | 810530115 | |
| SYBR Gold nucleic acid gel stain | Life Technologies | S11494 | |
| T4 Polynucleotide Kinase | New England Biolabs | M0201L | |
| T4 Polynucleotide Kinase Reaction Buffer | New England Biolabs | B0201S | |
| TBE-Urea Sample Buffer (2x) | Invitrogen | LC6876 | |
| Tris(hydroxymethyl)amino-methane, ultrapure, 99,9% | AlfaAesar | J65594 | |
| Triton X-100, 98% | Acros Organics | 327371000 | |
| Urea G.R. | lach:ner | 40096-AP0 |
References
- Ingolia, N. T., Ghaemmaghami, S., Newman, J. R., Weissman, J. S. Genome-wide analysis in vivo of translation with nucleotide resolution using ribosome profiling. Science. 324 (5924), 218-223 (2009).
- Takanami, M., Yan, Y., Jukes, T. H. Studies on the site of ribosomal binding of f2 bacteriophage RNA. Journal of Molecular Biology. 12 (3), 761-773 (1965).
- Steitz, J. A. Polypeptide Chain Initiation: Nucleotide Sequences of the Three Ribosomal Binding Sites in Bacteriophage R17 RNA. Nature. 224 (5223), 957-964 (1969).
- Wolin, S. L., Walter, P. Ribosome pausing and stacking during translation of a eukaryotic mRNA. The EMBO journal. 7 (11), 3559-3569 (1988).
- Argüello, R. J., et al. SunRiSE – measuring translation elongation at single-cell resolution by means of flow cytometry. Journal of Cell Science. 131 (10), 214346 (2018).
- Michel, A. M., Baranov, P. V. Ribosome profiling: a Hi-Def monitor for protein synthesis at the genome-wide scale. Wiley Interdisciplinary Reviews: RNA. 4 (5), 473-490 (2013).
- Woolstenhulme, C. J., Guydosh, N. R., Green, R., Buskirk, A. R. High-precision analysis of translational pausing by ribosome profiling in bacteria lacking EFP. Cell reports. 11 (1), 13-21 (2015).
- McGlincy, N. J., Ingolia, N. T. Transcriptome-wide measurement of translation by ribosome profiling. Methods (San Diego, Calif). 126, 112-129 (2017).
- Gerashchenko, M. V., Gladyshev, V. N. Translation inhibitors cause abnormalities in ribosome profiling experiments. Nucleic acids research. 42 (17), 134 (2014).
- Ingolia, N. T., Lareau, L. F., Weissman, J. S. Ribosome Profiling of Mouse Embryonic Stem Cells Reveals the Complexity of Mammalian Proteomes. Cell. 147 (4), 789-802 (2011).
- Ingolia, N. T., Brar, G. A., Rouskin, S., McGeachy, A. M., Weissman, J. S. The ribosome profiling strategy for monitoring translation in vivo by deep sequencing of ribosome-protected mRNA fragments. Nature protocols. 7 (8), 1534-1550 (2012).
- Hsu, P. Y., et al. Super-resolution ribosome profiling reveals unannotated translation events in Arabidopsis. Proceedings of the National Academy of Sciences of the United States of America. 113 (45), 7126-7135 (2016).
- Blanchard, S. C., Kim, H. D., Gonzalez, R. L., Puglisi, J. D., Chu, S. tRNA dynamics on the ribosome during translation. Proceedings of the National Academy of Sciences of the United States of America. 101 (35), 12893-12898 (2004).
- Becker, A. H., Oh, E., Weissman, J. S., Kramer, G., Bukau, B. Selective ribosome profiling as a tool for studying the interaction of chaperones and targeting factors with nascent polypeptide chains and ribosomes. Nature protocols. 8 (11), 2212-2239 (2013).
- Li, G. W., Oh, E., Weissman, J. S. The anti-Shine-Dalgarno sequence drives translational pausing and codon choice in bacteria. Nature. 484 (7395), 538-541 (2012).
- King, H. A., Gerber, A. P. Translatome profiling: methods for genome-scale analysis of mRNA translation. Briefings in functional genomics. 15 (1), 22-31 (2014).
- Mohammad, F., Woolstenhulme, C. J., Green, R., Buskirk, A. R. Clarifying the Translational Pausing Landscape in Bacteria by Ribosome Profiling. Cell reports. 14 (4), 686-694 (2016).
- Reid, D. W., Shenolikar, S., Nicchitta, C. V. Simple and inexpensive ribosome profiling analysis of mRNA translation. Methods (San Diego, Calif). 91, 69-74 (2015).
- Sanz, E., Yang, L., Su, T., Morris, D. R., McKnight, G. S., Amieux, P. S. Cell-type-specific isolation of ribosome-associated mRNA from complex tissues. Proceedings of the National Academy of Sciences of the United States of America. 106 (33), 13939-13944 (2009).
- Petrova, O. E., Garcia-Alcalde, F., Zampaloni, C., Sauer, K. Comparative evaluation of rRNA depletion procedures for the improved analysis of bacterialbiofilm and mixed pathogen culture transcriptomes. Scientific Reports. 7, 41114 (2017).
- Oh, E. Selective ribosome profiling reveals the cotranslational chaperone action of trigger factor in vivo. Cell. 147 (6), 1295-1308 (2011).
- Chew, G. L. Ribosome profiling reveals resemblance between long non-coding RNAs and 5′ leaders of coding RNAs. Development. 140 (13), 2828-2834 (2013).
- Dunn, J. G., Foo, C. K., Belletier, N. G., Gavis, E. R., Weissman, J. S. Ribosome profiling reveals pervasive and regulated stop codon readthrough in Drosophila melanogaster. eLife. 2, 01179 (2013).
- Liu, M. J., et al. Translational landscape of photomorphogenic Arabidopsis. The Plant cell. 25 (10), 3699-3710 (2013).
- Rooijers, K., Loayza-Puch, F., Nijtmans, L. G., Agami, R. Ribosome profiling reveals features of normal and disease-associated mitochondrial translation. Nature Communications. 4, 2886 (2013).
- Danielle, L., et al. Fidelity of translation initiation is required for coordinated respiratory complex assembly. Science Advances. 5 (12), 2118 (2019).
- Zoschke, R., Watkins, K. P., Barkan, A. A rapid ribosome profiling method elucidates chloroplast ribosome behavior in vivo. The Plant cell. 25 (6), 2265-2275 (2013).
- Chotewutmontri, P., Barkan, A. Dynamics of chloroplast translation during chloroplast differentiation in maize. PLoS genetics. 12 (7), 1006106 (2016).
- Lee, S., Liu, B., Huang, S. X., Shen, B., Qian, S. B. Global mapping of translation initiation sites in mammalian cells at single-nucleotide resolution. Proceedings of the National Academy of Sciences of the United States of America. 109 (37), 2424-2432 (2012).
- Martinez, T. F., Chu, Q., Donaldson, C., Tan, D., Shokhirev, M. N., Saghatelian, A. Accurate annotation of human protein-coding small open reading frames. Nature chemical biology. 16 (4), 458-468 (2020).
- Simsek, D., et al. The Mammalian Ribo-interactome Reveals Ribosome Functional Diversity and Heterogeneity. Cell. 169 (6), 1051-1065 (2017).
- Fritsch, C., et al. Genome-wide search for novel human uORFs and N-terminal protein extensions using ribosomal footprinting. Genome research. 22 (11), 2208-2218 (2012).
- Lareau, L. F., Hite, D. H., Hogan, G. J., Brown, P. O. Distinct stages of the translation elongation cycle revealed by sequencing ribosome-protected mRNA fragments. eLife. 3, 01257 (2014).
- Meydan, S., et al. Retapamulin-Assisted Ribosome Profiling Reveals the Alternative Bacterial Proteome. Molecular Cell. 74 (3), 481-493 (2019).
- Wilson, D. N. The A-Z of bacterial translation inhibitors. Critical Reviews in Biochemistry and Molecular Biology. 44, 393-433 (2009).
- Nakahigashi, K. Comprehensive identification of translation start sites by tetracycline-inhibited ribosome profiling. DNA research : an international journal for rapid publication of reports on genes and genomes. 23 (3), 193-201 (2016).
- Weaver, J., Mohammad, F., Buskirk, A. R., Storz, G. Identifying Small Proteins by Ribosome Profiling with Stalled Initiation Complexes. mBio. 10 (2), 02819 (2019).
- Nakahigashi, K. Effect of codon adaptation on codon-level and gene-level translation efficiency in vivo. BMC genomics. 15 (1), 1115 (2014).
- Li, G. W., Burkhardt, D., Gross, C., Weissman, J. S. Quantifying absolute protein synthesis rates reveals principles underlying allocation of cellular resources. Cell. 157 (3), 624-635 (2014).
- Michel, A. M., et al. Observation of dually decoded regions of the human genome using ribosome profiling data. Genome research. 22 (11), 2219-2229 (2012).
- Guydosh, N. R., Green, R. Dom34 rescues ribosomes in 3′ untranslated regions. Cell. 156 (5), 950-962 (2014).
- Young, D. J., Guydosh, N. R., Zhang, F., Hinnebusch, A. G., Green, R. Rli1/ABCE1 Recycles Terminating Ribosomes and Controls Translation Reinitiation in 3’UTRs In Vivo. Cell. 162 (4), 872-884 (2015).
- Ingolia, N. T. Ribosome profiling: new views of translation, from single codons to genome scale. Nature Reviews Genetics. 15 (3), 205-213 (2014).
- Dobin, A., et al. ultrafast universal RNA seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- . . Babraham Bioinformatics. Fast QC. , (2021).
- Gerashchenko, M. V., Gladyshev, V. N. Ribonuclease selection for ribosome profiling. Nucleic acids research. 45 (2), 6 (2017).
- Verbruggen, S., Menschaert, G. mQC: A post-mapping data exploration tool for ribosome profiling. Computer Methods and Programs in Biomedicine. 181, 104806 (2019).
- Cui, H., Hu, H., Zeng, J., Chen, T. DeepShape: estimating isoform-level ribosome abundance and distribution with Ribo-seq data. BMC bioinformatics. 20, 678 (2019).
- Choi, J. . RiboToolkit: an integrated platform for analysis and annotation of ribosome profiling data to decode RNA translation at codon resolution. , (2021).
- Choi, J. Dynamics of the context-specific translation arrest by chloramphenicol and linezolid. Nature chemical biology. 16 (3), 310-317 (2020).
- Tompson, J., O’Connor, M., Mills, J. A., Dahlberg, A. E. The protein synthesis inhibitors, oxazolidinones and chloramphenicol, cause extensive translational inaccuracy in vivo. Journal of Molecular Biology. 322 (2), 273-279 (2002).
- Brar, G. A., Weissman, J. S. Ribosome profiling reveals the what, when, where and how of protein synthesis. Nature reviews. Molecular cell biology. 16 (11), 651-664 (2015).
- Glaub, A., Huptas, C., Neuhaus, K., Ardern, Z. Recommendations for bacterial ribosome profiling experiments based on bioinformatic evaluation of published data. Journal of Biological Chemistry. 295, 8999-9011 (2020).
- Sorour, M. H., Hani, H. A., Shaalan, H. F., El-Sayed, M. M. H. Experimental screening of some chelating agents for calcium and magnesium removal from saline solutions. Desalination and Water Treatment. 57 (48-49), 22799-22808 (2015).
- Mohammad, F., Green, R., Buskirk, A. R. A systematically-revised ribosome profiling method for bacteria reveals pauses at single-codon resolution. Elife. 8, 42591 (2019).
- O’Connor, P. B., Li, G. W., Weissman, J. S., Atkins, J. F., Baranov, P. V. rRNA:mRNA pairing alters the length and the symmetry of mRNA-protected fragments in ribosome profiling experiments. Bioinformatics. 29 (12), 1488-1491 (2013).
- Protocol for RNA Clean & Concentrator -25. Zymo Research Available from: https://files.zymoresearch.com/protocols/_r1017_r1018_rna_clean_concentrator-25.pdf (2021)
- Protocol for use with NEBNext Small RNA Library Prep Set for Illumina (E7300, E7580, E7560, E7330). New England Biolabs Available from: https://www.international.neb.com/protocols/2018/03/27/protocol-for-use-with-nebnext-small-rna-library-prep-set-for-illumina-e7300-e7580-e7560-e7330 (2021)

