Kontinuerlig fluorescensbaseret endonukleasekoblet DNA-methyleringsassay til screening for DNA-methyltransferasehæmmere
Summary
DNA-methyltransferaser er potentielle kræftlægemiddelmål. Her præsenteres en protokol til vurdering af små molekyler til DNA-methyltransferasehæmning. Dette assay anvender en endonuklease til at koble DNA-methylering til fluorescensgenerering og gør det muligt at overvåge enzymaktivitet i realtid.
Abstract
DNA-methylering, en form for epigenetisk genregulering, er vigtig for normal cellulær funktion. I celler etablerer og opretholder proteiner kaldet DNA-methyltransferaser (DNMT’er) DNA-methyleringsmønsteret. Ændringer i det normale DNA-methyleringsmønster er forbundet med kræftudvikling og progression, hvilket gør DNMT’er potentielle kræftlægemiddelmål. Således er identifikation og karakterisering af nye små molekylehæmmere af disse enzymer af stor betydning. Dette papir præsenterer en protokol, der kan bruges til at screene for DNA-methyltransferasehæmmere. Det kontinuerlige koblede kinetikassay gør det muligt at bestemme initialhastigheder af DNA-methylering i nærvær og fravær af potentielle små molekylehæmmere. Analysen bruger den methylfølsomme endonuklease Gla I til at koble methylering af et hemimethyleret DNA-substrat til fluorescensgenerering.
Dette kontinuerlige assay gør det muligt at overvåge enzymaktivitet i realtid. Gennemførelse af analysen i små mængder i mikrotiterplader reducerer omkostningerne ved reagenser. Ved hjælp af dette assay blev der udført en lille eksempelskærm for hæmmere af DNMT1, det mest rigelige DNMT-isozym hos mennesker. Det stærkt substituerede anthraquinon naturlige produkt, laccainsyre A, er en potent, DNA-konkurrencedygtig hæmmer af DNMT1. Her undersøger vi tre potentielle små molekylehæmmere – anthraquinoner eller anthraquinonlignende molekyler med en til tre substituenter – i to koncentrationer for at beskrive analyseprotokollen. Indledende hastigheder bruges til at beregne den procentvise aktivitet, der observeres i nærværelse af hvert molekyle. En af tre undersøgte forbindelser udviser koncentrationsafhængig hæmning af DNMT1-aktivitet, hvilket indikerer, at det er en potentiel hæmmer af DNMT1.
Introduction
DNA-methylering er et vigtigt epigenetisk mærke, der regulerer genekspression og kromatinstruktur. Methylering forekommer overvejende i CpG-dinukleotider – cytosin efterfulgt af guanosin; methylgruppen tilsættes til 5-positionen af cytosin. Korrekte DNA-methyleringsmønstre og dermed korrekt genekspression er nødvendige for passende cellulær udvikling og funktion. Mange sygdomstilstande har været forbundet med ændringer i det normale methyleringsmønster 1,2,3. For eksempel er der en sammenhæng mellem kræftinitiering og progression og ændringer i DNA-methyleringsmønsteret. Typisk udviser kræftceller lavere samlede niveauer af methylcytosin, hvilket bidrager til genom ustabilitet. Samtidig er methylcytosinet, der er til stede i genomet, koncentreret i promotorregionerne af tumorundertrykkende gener, hvilket fører til genhæmning af disse vigtige proteiner. Især er epigenetiske ændringer dynamiske og reversible, i modsætning til DNA-mutationerne forbundet med tumorigenese. Dette har gjort proteinerne involveret i epigenetisk genregulering interessante lægemiddelmål 2,4.
DNA-methyltransferaser (DNMT’er) er de proteiner, der er ansvarlige for at generere og opretholde DNA-methyleringsmønstre. Tre katalytisk aktive isozymer, DNMT1, DNMT3a og DNMT3b, findes hos mennesker. Under udvikling og differentiering etablerer de novo methyltransferaserne, DNMT3a og DNMT3b, methyleringsmønstre. Begge enzymer kan binde det katalytisk inaktive DNMT3L-protein til dannelse af komplekser, der udviser øget aktivitet 1,5. Efter celledeling indeholder datterceller hemimethyleret DNA – DNA indeholdende methylcytosin i kun en streng af duplexet – fordi det nyligt syntetiserede DNA er blottet for methyleringsmærker. DNMT1’s vigtigste funktion er at methylere dette hemimethylerede DNA og dermed genetablere det fulde methyleringsmønster 1,5.
Forbindelser mellem DNMT-aktivitet og kræft er veletablerede. Overekspression af DNMT1, enten ved transkriptionelle eller posttranslationelle mekanismer, er en konsekvens af flere almindelige onkogene veje 6,7,8,9. Genetiske tilgange til lavere DNMT1-aktivitet ved hjælp af hypomorfe alleler resulterer i nedsat tumordannelse i Apc (Min) mus10. Antisense oligonukleotider, der knockdown DNMT1 hæmmer neoplasi i cellekultur og musetumormodeller11,12. Således virker hæmning af DNMT1-aktivitet som en lovende tilgang til kræftbehandling. De roller, som DNMT3 isozymer spiller, er dog ikke så ligetil. DNMT3a mutationer findes i akut myeloid leukæmi13 og myelodysplastisk syndrom14. Mindst en af de identificerede mutationer har vist sig at mindske DNA-methyleringsaktiviteten af enzymet15. DNMT3b er imidlertid overudtrykt i brystkræft16 og kolorektal cancer17. Da de forskellige DNMT-isozymer spiller forskellige roller i carcinogenese, vil identifikation af isozymespecifikke hæmmere være kritisk. Ikke alene vil disse forbindelser være nyttige til udvikling af terapi, men isozymspecifikke hæmmere ville også være et uvurderligt redskab til at dissekere hver DNMT-isozyms rolle i kræftetiologi.
Flere DNMT-hæmmere er blevet rapporteret i litteraturen. Kendte DNMT-hæmmere kan opdeles i to klasser: nukleosid og ikke-nukleosid. Nukleosidhæmmere er typisk cytidinanaloger. Disse forbindelser er inkorporeret i DNA og kovalent fælde DNMT’er. 5-azacytidin og 5-aza-2′-deoxycytidin er blevet godkendt til behandling af myelodysplastisk syndrom og akut myeloid leukæmi 4,18. Den høje toksicitet, lave biotilgængelighed og kemisk ustabilitet af disse forbindelser udgør problemer. Igangværende arbejde undersøger effektiviteten af den næste generation af nukleosidhæmmere; SGI-110, afledt af 5-aza-2′-deoxycytidin, er et eksempel 19,20. Nukleosidhæmmere er ikke isozymespecifikke og vil inaktivere ethvert DNMT-isozym, der opstår. Derfor resulterer behandling med et nukleosid-demethyleringsmiddel i udtømning af alle DNMT-isozymer 4,18. Ikke-nukleosidhæmmere behøver ikke at blive inkorporeret i DNA for at udøve deres hæmmende virkninger. I stedet binder disse molekyler direkte til DNMT’er, hvilket introducerer muligheden for isozymspecifik hæmning. Flere ikke-nukleosidhæmmere er blevet opdaget til dato, herunder SGI-102721, hydralazin 22, procainamid 23, RG108 og derivater 24 og naturlige produkter, (-)-epigallocatechin 3-gallat (EGCG)25 og laccainsyre A 26,27. De fleste af de ikke-nukleosidhæmmere, der er opdaget til dato, er ikke isozymeselektive eller viser svage præferencer for et DNMT-isozym. Derudover skal styrken af disse molekyler forbedres, især i celler 4,18. Der er således behov for at opdage eller udvikle mere potente, isozymeselektive DNMT-hæmmere.
En hindring for at opdage nye små molekylehæmmere af DNMT’er er de besværlige assays, der traditionelt bruges til at undersøge DNMT-aktivitet28. Assays er normalt diskontinuerlige med flere trin. Den enzymatiske aktivitet af DNMT’er analyseres stadig rutinemæssigt ved anvendelse af radioaktivt S-adenosylmethionin (SAM)29,30,31,32,33,34. Ikke-radioaktive assays til DNA-methylering er også blevet udviklet. For eksempel er assays, der anvender methylfølsomme begrænsningsendonukleaser og elektroforese til at adskille fordøjelsesprodukterne, blevet beskrevet35,36. Disse typer diskontinuerlige, multistep assays er ikke let modtagelige for lægemiddelopdagelse. Siden midten af 2000’erne er der udviklet flere DNA-methyleringsassays med en højere gennemstrømning28. Et scintillationsnærhedsassay blev brugt til at screene for DNMT1-hæmmere37. Et andet assay ved hjælp af en methylfølsom restriktionsendonuklease blev brugt til at screene for DNMT3a-hæmmere25,38. Mens begge assays tillod højere gennemstrømning end traditionelle DNA-methyleringsassays, kræver assays flere trin og tillader ikke observation af methyleringsaktivitet i realtid. For nylig er der beskrevet et kontinuerligt kinetikassay, der kobler dannelsen af S-adenosylhomocystein (SAH), et produkt af methyleringsreaktionen, til den spektroskopiske ændring ved 340 nm forbundet med NADPH-oxidation39. Dette assay bruger tre koblingsenzymer til at generere et spektroskopisk signal.
Vi udviklede et fluorescensbaseret endonukleasekoblet DNA-methyleringsassay, der anvender et enkelt kommercielt tilgængeligt koblingsenzym og kan generere data i realtid (figur 1). Et hårnål oligonukleotid indeholdende tre methylcytosiner anvendes som substrat. Substrat-DNA’et indeholder en fluorophore i 5′-enden og en slukker i 3′-enden. Methylering af det hemimethylerede CpG-sted genererer spaltningsstedet for endonuklease Gla I – fuldt methyleret GCGC. Gla I-spaltning af produktet oligonukleotid frigiver fluoroforen fra slukningsenheden og genererer fluorescens i realtid. Analysen kan bruges til at undersøge aktiviteten af enhver isoform af DNMT; der observeres dog højere aktivitet med DNMT1, da dette isozyme fortrinsvis methylerer hemimethyleret DNA 1,5. Endnu mere robust aktivitet observeres, hvis det autoinhibitory Replication Foci Targeting Sequence (RFTS) domæne fjernes fra DNMT1. Dette domæne, der findes i den N-terminale regulatoriske region, binder til det katalytiske sted og forhindrer DNA-binding. Fjernelse af de første ~600 aminosyrer resulterer i et afkortet enzym, der er signifikant mere aktivt end enzymet i fuld længde (~640 gange stigning i kkat/Km)40. Denne aktiverede form af enzymet, kaldet RFTS-manglende DNMT1 (aminosyrer 621–1616), giver mulighed for lettere identifikation af hæmmere på grund af dets øgede katalytiske kraft. Dette papir præsenterer en protokol til at bruge RFTS-manglende DNMT1 i assays til at screene for potentielle små molekylehæmmere. Ved anvendelse af det endonukleasekoblede kontinuerlige assay bestemmes starthastigheden i nærvær og fravær af nogle få små molekyler. Hver potentiel hæmmer undersøges ved to koncentrationer for at se efter koncentrationsafhængig DNMT1-hæmning. Den procentvise aktivitet, der blev observeret i nærværelse af de små molekyler, blev beregnet i hvert enkelt tilfælde.
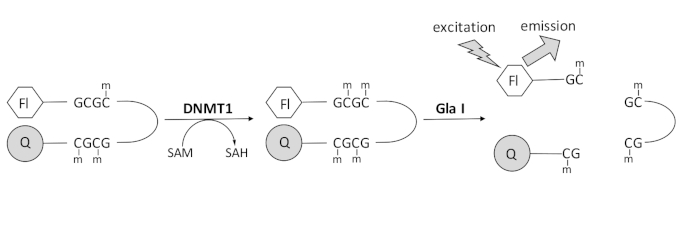
Figur 1: DNA-methyleringsassay. Et hemimethyleret hårnål-DNA med en fluorophore i 5′-enden og en quencher i 3′-enden bruges som substrat. DNMT1 katalyserer overførslen af methylgruppen fra S-adenosylmethionin til det ikke-methylerede CpG-sted og genererer S-adenosylhomocystein og fuldt methyleret DNA. DNA-produktet indeholder spaltningsstedet for endonuklease Gla I, som spalter fuldt methylerede GCGC-steder. Spaltning af produkt-DNA’et frigiver 5′-fluoroforen fra 3′-slukningsenheden og genererer fluorescens. Forkortelser: Fl = fluorophore; Q = quencher; DNMT1 = DNA-methyltransferase 1; SAM = S-adenosylmethionin; SAH = S-adenosylhomocystein. Klik her for at se en større version af denne figur.
Protocol
Representative Results
Discussion
For at identificere og karakterisere hæmmere af DNA-methyltransferaser skal enzymets aktivitet måles. Der findes flere metoder til undersøgelse af DNA-methyltransferaseaktivitet. Aktivitet overvåges almindeligvis ved hjælp af radioaktivitet; overførsel af den mærkede methylgruppe af SAM kan kvantificeres 29,30,31,32,33,34.</…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Forfatterne takker Bucknell University og Institut for Kemi for deres støtte til dette arbejde.
Materials
| 96-well Half Area Black Flat Bottom Polystyrene Not Treated Microplate | Corning | 3694 | |
| 96-Well Polystyrene Conical Bottom Plates | ThermoFisher | 249570 | |
| Bovine Serum Albumin | NEB | B9000S | |
| compound 1 | ChemBridge | 5812086 | screening compound; resuspended in DMSO to 10 mM |
| compound 2 | ChemBridge | 6722175 | screening compound; resuspended in DMSO to 10 mM |
| compound 3 | ChemBridge | 5249376 | screening compound; resuspended in DMSO to 10 mM |
| Dithiothreitol | Sigma | D0632 | |
| Gla I | SibEnzyme | E494 | methyl-sensitive endonuclease |
| Glycerol | RPI | G22025 | |
| Magnesium Chloride | Sigma | M0250 | |
| Oligonucleotide (5'-FAM-CCTATGCGmCATCAGTTTTCTGATGmCGmCATAGG-3'-Iowa Black Quencher) | IDT | custom synthesized | internally quenched hairpin DNA (substrate) |
| Potassium Glutamate | Sigma | G1501 | |
| S-adenosylmethionine | Sigma | A4377 | methyl-donating co-factor (substrate) |
| Tris Base | RPI | T60040 |
References
- Jurkowska, R. Z., Jurkowski, T. P., Jeltsch, A. Structure and function of mammalian DNA methyltransferases. Chembiochem. 12 (2), 206-222 (2011).
- Hamidi, T., Singh, A. K., Chen, T. Genetic alterations of DNA methylation machinery in human diseases. Epigenomics. 7 (2), 247-265 (2015).
- Norvil, A. B., Saha, D., Dar, M. S., Gowher, H. Effect of disease-associated germline mutations on structure function relationship of DNA methyltransferases. Genes. 10 (5), 369 (2019).
- Foulks, J. M., et al. Epigenetic drug discovery: targeting DNA methyltransferases. Journal of Biomolecular Screening. 17 (1), 2-17 (2012).
- Goll, M. G., Bestor, T. H. Eukaryotic cytosine methyltransferases. Annual Review of Biochemistry. 74, 481-514 (2005).
- Bigey, P., Ramchandani, S., Theberge, J., Araujo, F. D., Szyf, M. Transcriptional regulation of the human DNA Methyltransferase (dnmt1) gene. Gene. 242 (1-2), 407-418 (2000).
- Detich, N., Ramchandani, S., Szyf, M. A conserved 3′-untranslated element mediates growth regulation of DNA methyltransferase 1 and inhibits its transforming activity. Journal of Biological Chemistry. 276 (27), 24881-24890 (2001).
- MacLeod, A. R., Rouleau, J., Szyf, M. Regulation of DNA methylation by the Ras signaling pathway. Journal of Biological Chemistry. 270 (19), 11327-11337 (1995).
- Slack, A., Cervoni, N., Pinard, M., Szyf, M. DNA methyltransferase is a downstream effector of cellular transformation triggered by simian virus 40 large T antigen. Journal of Biological Chemistry. 274 (15), 10105-10112 (1999).
- Eads, C. A., Nickel, A. E., Laird, P. W. Complete genetic suppression of polyp formation and reduction of CpG-island hypermethylation in Apc(Min/+) Dnmt1-hypomorphic mice. Cancer Research. 62, 1296-1299 (2002).
- MacLeod, A. R., Szyf, M. Expression of antisense to DNA methyltransferase mRNA induces DNA demethylation and inhibits tumorigenesis. Journal of Biological Chemistry. 270 (14), 8037-8043 (1995).
- Ramchandani, S., MacLeod, A. R., Pinard, M., von Hofe, E., Szyf, M. Inhibition of tumorigenesis by a cytosine-DNA, methyltransferase, antisense oligodeoxynucleotide. Proceedings of the National Academy Sciences of the United States of America. 94 (2), 684-689 (1997).
- Ley, T. J., et al. DNMT3A mutations in acute myeloid leukemia. New England Journal of Medicine. 363, 2424-2433 (2010).
- Walter, M. J., et al. Recurrent DNMT3A mutations in patients with myelodysplastic syndromes. Leukemia. 25 (7), 1153-1158 (2011).
- Russler-Germain, D. A., et al. The R882H DNMT3A mutation associated with AML dominantly inhibits wild-type DNMT3A by blocking its ability to form active tetramers. Cancer Cell. 25 (4), 442-454 (2014).
- Roll, J. D., Rivenbark, A. G., Jones, W. D., Coleman, W. B. DNMT3b overexpression contributes to a hypermethylator phenotype in human breast cancer cell lines. Molecular Cancer. 7, 15 (2008).
- Nosho, K., et al. DNMT3B expression might contribute to CpG island methylator phenotype in colorectal cancer. Clinical Cancer Research. 15 (11), 3663-3671 (2009).
- Erdmann, A., Halby, L., Fahy, J., Arimondo, P. B. Targeting DNA methylation with small molecules: what’s next. Journal of Medicinal Chemistry. 58 (6), 2569-2583 (2015).
- Chuang, J. C., et al. S110, a 5-Aza-2′-deoxycytidine-containing dinucleotide, is an effective DNA methylation inhibitor in vivo and can reduce tumor growth. Molecular Cancer Therapeutics. 9 (5), 1443-1450 (2010).
- Issa, J. J., et al. Safety and tolerability of guadecitabine (SGI-110) in patients with myelodysplastic syndrome and acute myeloid leukaemia: a multicentre, randomised, dose-escalation phase 1 study. Lancet Oncology. 16 (9), 1099-1110 (2015).
- Datta, J., et al. A new class of quinoline-based DNA hypomethylating agents reactivates tumor suppressor genes by blocking DNA methyltransferase 1 activity and inducing its degradation. Cancer Research. 69 (10), 4277-4285 (2009).
- Zambrano, P., et al. A phase I study of hydralazine to demethylate and reactivate the expression of tumor suppressor genes. BMC Cancer. 5, 44 (2005).
- Lee, B. H., Yegnasubramanian, S., Lin, X., Nelson, W. G. Procainamide is a specific inhibitor of DNA methyltransferase 1. Journal of Biological Chemistry. 280 (49), 40749-40756 (2005).
- Asgatay, S., et al. Synthesis and evaluation of analogues of N-phthaloyl-l-tryptophan (RG108) as inhibitors of DNA methyltransferase 1. Journal of Medicinal Chemstry. 57 (2), 421-434 (2014).
- Ceccaldi, A., et al. C5-DNA methyltransferase inhibitors: from screening to effects on zebrafish embryo development. Chembiochem. 12 (9), 1337-1345 (2011).
- Fagan, R. L., Wu, M., Chédin, F., Brenner, C. An ultrasensitive high throughput screen for DNA methyltransferase 1-targeted molecular probes. PLoS One. 8 (11), 78752 (2013).
- Fagan, R. L., Cryderman, D. E., Kopelovich, L., Wallrath, L. L., Brenner, C. Laccaic acid A is a direct, DNA-competitive inhibitor of DNA methyltransferase 1. Journal of Biological Chemistry. 288 (33), 23858-23867 (2013).
- Eglen, R. M., Reisine, T. Screening for compounds that modulate epigenetic regulation of the transcriptome: an overview. Journal of Biomolecular Screening. 16 (10), 1137-1152 (2011).
- Holz-Schietinger, C., Matje, D. M., Reich, N. O. Mutations in DNA methyltransferase (DNMT3A) observed in acute myeloid leukemia patients disrupt processive methylation. Journal of Biological Chemistry. 287 (37), 30941-30951 (2012).
- Norvil, A. B., et al. Dnmt3b methylates DNA by a noncooperative mechanism, and its activity is unaffected by manipulations at the predicted dimer interface. Biochemistry. 57 (29), 4312-4324 (2018).
- Bashtrykov, P., Ragozin, S., Jeltsch, A. Mechanistic details of the DNA recognition by the Dnmt1 DNA methyltransferase. FEBS Letters. 586 (13), 1821-1823 (2012).
- Bashtrykov, P., et al. Targeted mutagenesis results in an activation of DNA methyltransferase 1 and confirms an autoinhibitory role of its RFTS domain. Chembiochem. 15 (5), 743-748 (2014).
- Berkyurek, A. C., et al. The DNA methyltransferase Dnmt1 directly interacts with the SET and RING finger-associated (SRA) domain of the multifunctional protein Uhrf1 to facilitate accession of the catalytic center to hemi-methylated DNA. Journal of Biological Chemistry. 289 (1), 379-386 (2014).
- Kanada, K., Takeshita, K., Suetake, I., Tajima, S., Nakagawa, A. Conserved threonine 1505 in the catalytic domain stabilizes mouse DNA methyltransferase 1. Journal of Biochemistry. 162 (4), 271-278 (2017).
- Bashtrykov, P., et al. Specificity of Dnmt1 for methylation of hemimethylated CpG sites resides in its catalytic domain. Chemistry & Biology. 19 (5), 572-578 (2012).
- Dolen, E. K., McGinnis, J. H., Tavory, R. N., Weiss, J. A., Switzer, R. L. Disease-associated mutations G589A and V590F relieve replication focus targeting sequence-mediated autoinhibition of DNA methyltransferase 1. Biochemistry. 58 (51), 5151-5159 (2019).
- Kilgore, J. A., et al. Identification of DNMT1 selective antagonists using a novel scintillation proximity assay. Journal of Biological Chemistry. 288 (27), 19673-19684 (2013).
- Ceccaldi, A., et al. Identification of novel inhibitors of DNA methylation by screening of a chemical library. ACS Chemical Biology. 8 (3), 543-548 (2013).
- Duchin, S., Vershinin, Z., Levy, D., Aharoni, A. A continuous kinetic assay for protein and DNA methyltransferase enzymatic activities. Epigenetics & Chromatin. 8, 56 (2015).
- Syeda, F., et al. The replication focus targeting sequence (RFTS) domain is a DNA-competitive inhibitor of Dnmt1. Journal of Biological Chemistry. 286 (17), 15344-15351 (2011).
- Switzer, R. L., Medrano, J., Reedel, D. A., Weiss, J. Substituted anthraquinones represent a potential scaffold for DNA methyltransferase 1-specific inhibitors. PLoS One. 14 (7), 0219830 (2019).

