Saggio di metilazione del DNA accoppiato a endonucleasi a fluorescenza continua per lo screening degli inibitori della DNA metiltransferasi
Summary
Le DNA metiltransferasi sono potenziali bersagli di farmaci antitumorali. Qui, viene presentato un protocollo per valutare piccole molecole per l’inibizione della DNA metiltransferasi. Questo test utilizza un’endonucleasi per accoppiare la metilazione del DNA alla generazione di fluorescenza e consente di monitorare l’attività enzimatica in tempo reale.
Abstract
La metilazione del DNA, una forma di regolazione genica epigenetica, è importante per la normale funzione cellulare. Nelle cellule, le proteine chiamate DNA metiltransferasi (DNMT) stabiliscono e mantengono il modello di metilazione del DNA. Le modifiche al normale modello di metilazione del DNA sono legate allo sviluppo e alla progressione del cancro, rendendo i DNMT potenziali bersagli di farmaci antitumorali. Pertanto, identificare e caratterizzare nuovi inibitori di piccole molecole di questi enzimi è di grande importanza. Questo documento presenta un protocollo che può essere utilizzato per lo screening degli inibitori della DNA metiltransferasi. Il test di cinetica accoppiata continua consente di determinare le velocità iniziali di metilazione del DNA in presenza e assenza di potenziali inibitori di piccole molecole. Il test utilizza l’endonucleasi metil-sensibile Gla I per accoppiare la metilazione di un substrato di DNA emimetilato alla generazione di fluorescenza.
Questo test continuo consente di monitorare l’attività enzimatica in tempo reale. Condurre il test in piccoli volumi in piastre di microtitolazione riduce il costo dei reagenti. Utilizzando questo test, è stato condotto un piccolo esempio di screening per gli inibitori di DNMT1, l’isoenzima DNMT più abbondante nell’uomo. Il prodotto naturale antrachinone altamente sostituito, l’acido laccaico A, è un potente inibitore competitivo del DNA di DNMT1. Qui, esaminiamo tre potenziali inibitori di piccole molecole – antrachinoni o molecole simili all’antrachinone con uno o tre sostituenti – a due concentrazioni per descrivere il protocollo di analisi. Le velocità iniziali vengono utilizzate per calcolare l’attività percentuale osservata in presenza di ciascuna molecola. Uno dei tre composti esaminati mostra un’inibizione concentrazione-dipendente dell’attività di DNMT1, indicando che si tratta di un potenziale inibitore di DNMT1.
Introduction
La metilazione del DNA è un importante marchio epigenetico che regola l’espressione genica e la struttura della cromatina. La metilazione si verifica prevalentemente nei dinucleotidi CpG – citosina seguita da guanosina; Il gruppo metilico viene aggiunto alla posizione 5 della citosina. Sono necessari corretti modelli di metilazione del DNA, e quindi una corretta espressione genica, per un appropriato sviluppo e funzione cellulare. Molti stati patologici sono stati associati a cambiamenti nel normale modello di metilazione 1,2,3. Ad esempio, esiste un legame tra l’inizio e la progressione del cancro e le alterazioni del modello di metilazione del DNA. Tipicamente, le cellule tumorali mostrano livelli complessivi più bassi di metilcitosina, che contribuisce all’instabilità del genoma. Allo stesso tempo, la metilcitosina presente nel genoma è concentrata nelle regioni promotrici dei geni oncosoppressori, che porta al silenziamento genico di queste importanti proteine. In particolare, i cambiamenti epigenetici sono dinamici e reversibili, a differenza delle mutazioni del DNA associate alla tumorigenesi. Ciò ha reso le proteine coinvolte nella regolazione genica epigenetica interessanti bersagli farmacologici 2,4.
Le DNA metiltransferasi (DNMT) sono le proteine responsabili della generazione e del mantenimento dei modelli di metilazione del DNA. Tre isoenzimi cataliticamente attivi, DNMT1, DNMT3a e DNMT3b, esistono negli esseri umani. Durante lo sviluppo e la differenziazione, le metiltransferasi de novo, DNMT3a e DNMT3b, stabiliscono modelli di metilazione. Entrambi gli enzimi possono legare la proteina DNMT3L cataliticamente inattiva per formare complessi che mostrano una maggiore attività 1,5. Dopo la divisione cellulare, le cellule figlie contengono DNA emimetilato – DNA contenente metilcitosina in un solo filamento del duplex – perché il DNA appena sintetizzato è privo di segni di metilazione. La funzione principale di DNMT1 è quella di metilare questo DNA emimetilato, ristabilendo così il modello di metilazione completo 1,5.
I legami tra l’attività di DNMT e il cancro sono ben stabiliti. La sovraespressione di DNMT1, sia attraverso meccanismi trascrizionali che post-traduzionali, è una conseguenza di diverse vie oncogeniche comuni 6,7,8,9. Gli approcci genetici per ridurre l’attività di DNMT1 utilizzando alleli ipomorfici determinano una diminuzione della formazione di tumore nei topi Apc (Min) 10. Gli oligonucleotidi antisenso che abbattono DNMT1 inibiscono la neoplasia in colture cellulari e modelli tumorali murini11,12. Pertanto, l’inibizione dell’attività DNMT1 sembra un approccio promettente alla terapia del cancro. Tuttavia, i ruoli che gli isoenzimi DNMT3 svolgono non sono così semplici. Le mutazioni di DNMT3a si trovano nella leucemia mieloide acuta13 e nella sindrome mielodisplastica14. Almeno una delle mutazioni identificate ha dimostrato di diminuire l’attività di metilazione del DNA dell’enzima15. Tuttavia, DNMT3b è sovraespresso nel cancro al seno16 e nel cancro del colon-retto17. Con i vari isoenzimi DNMT che svolgono ruoli diversi nella carcinogenesi, l’identificazione di inibitori isoenzimatici specifici sarà fondamentale. Non solo questi composti saranno utili per lo sviluppo di terapie, ma gli inibitori isoenzimatico-specifici sarebbero anche uno strumento inestimabile per analizzare il ruolo di ciascun isoenzima DNMT nell’eziologia del cancro.
In letteratura sono stati riportati diversi inibitori della DNMT. Gli inibitori DNMT noti possono essere suddivisi in due classi: nucleoside e non nucleoside. Gli inibitori nucleosidici sono tipicamente analoghi della citidina. Questi composti sono incorporati nel DNA e intrappolano covalentemente i DNMT. 5-azacitidina e 5-aza-2′-deossicitidina sono stati approvati per il trattamento della sindrome mielodisplastica e della leucemia mieloide acuta 4,18. L’elevata tossicità, la bassa biodisponibilità e l’instabilità chimica di questi composti presentano problemi. Il lavoro in corso sta esaminando l’efficacia della prossima generazione di inibitori nucleosidici; SGI-110, derivato dalla 5-aza-2′-deossicitidina, è un esempio 19,20. Gli inibitori nucleosidici non sono isoenzimo-specifici e inattiveranno qualsiasi isoenzima DNMT incontrato. Pertanto, il trattamento con un agente nucleosidico demetilante determina la deplezione di tutti gli isoenzimi DNMT 4,18. Gli inibitori non nucleosidici non hanno bisogno di essere incorporati nel DNA per esercitare i loro effetti inibitori. Invece, queste molecole si legano direttamente ai DNMT, introducendo la possibilità di inibizione isoenzima-specifica. Diversi inibitori non-nucleosidici sono stati scoperti fino ad oggi, tra cui SGI-1027 21, idralazina 22, procainamide 23, RG108 e derivati 24, e prodotti naturali, (-)-epigallocatechina 3-gallato (EGCG)25 e acido laccaico A 26,27. La maggior parte degli inibitori non nucleosidici scoperti fino ad oggi non sono isoenzimi-selettivi o mostrano preferenze deboli per un isoenzima DNMT. Inoltre, la potenza di queste molecole deve essere migliorata, specialmente nelle cellule 4,18. Pertanto, vi è la necessità di scoprire o sviluppare inibitori DNMT più potenti e isoenzimatici.
Un ostacolo alla scoperta di nuovi inibitori di piccole molecole di DNMT sono i laboriosi saggi tradizionalmente utilizzati per esaminare l’attività DNMT28. I saggi sono generalmente discontinui con più passaggi. L’attività enzimatica dei DNMT viene ancora regolarmente analizzata utilizzando S-adenosil metionina radioattiva (SAM)29,30,31,32,33,34. Sono stati sviluppati anche saggi non radioattivi per la metilazione del DNA. Ad esempio, sono stati descritti saggi che utilizzano endonucleasi di restrizione metil-sensibile ed elettroforesi per separare i prodotti della digestione35,36. Questi tipi di saggi discontinui e multistep non sono facilmente suscettibili di scoperta di farmaci. Dalla metà degli anni 2000, sono stati sviluppati diversi saggi di metilazione del DNA con un throughput più elevato28. Un saggio di prossimità a scintillazione è stato utilizzato per lo screening degli inibitori DNMT137. Un altro test che utilizza un’endonucleasi di restrizione metil-sensibile è stato utilizzato per lo screening degli inibitori DNMT3a25,38. Mentre entrambi i test hanno consentito una maggiore produttività rispetto ai tradizionali test di metilazione del DNA, i test richiedono più passaggi e non consentono l’osservazione dell’attività di metilazione in tempo reale. Più recentemente, è stato descritto un saggio cinetico continuo che accoppia la formazione di S-adenosilomocisteina (SAH), un prodotto della reazione di metilazione, al cambiamento spettroscopico a 340 nm associato all’ossidazione NADPH39. Questo test utilizza tre enzimi di accoppiamento per generare un segnale spettroscopico.
Abbiamo sviluppato un saggio di metilazione del DNA accoppiato a endonucleasi basato sulla fluorescenza che utilizza un singolo enzima di accoppiamento disponibile in commercio e può generare dati in tempo reale (Figura 1). Un oligonucleotide a forcina contenente tre metilcitosine viene utilizzato come substrato. Il DNA del substrato contiene un fluoroforo all’estremità 5′ e un quencher all’estremità 3′. La metilazione del sito CpG emimetilato genera il sito di scissione per l’endonucleasi Gla I – GCGC completamente metilato. La scissione Gla I dell’oligonucleotide prodotto rilascia il fluoroforo dal quencher e genera fluorescenza in tempo reale. Il test può essere utilizzato per esaminare l’attività di qualsiasi isoforma di DNMT; tuttavia, si osserva una maggiore attività con DNMT1 poiché questo isoenzima metila preferenzialmente il DNA emimetilato 1,5. Un’attività ancora più robusta si osserva se il dominio RFTS (Replication Foci Targeting Sequence) autoinibitorio viene rimosso da DNMT1. Questo dominio, che si trova nella regione regolatoria N-terminale, si lega al sito catalitico e impedisce il legame del DNA. La rimozione dei primi ~ 600 amminoacidi si traduce in un enzima troncato che è significativamente più attivo dell’enzima a lunghezza intera (aumento di ~ 640 volte in kcat / Km)40. Questa forma attivata dell’enzima, denominata DNMT1 privo di RFTS (aminoacidi 621-1616), consente una più facile identificazione degli inibitori grazie al suo maggiore potere catalitico. Questo articolo presenta un protocollo per utilizzare DNMT1 privo di RFTS nei saggi per lo screening di potenziali inibitori di piccole molecole. Utilizzando il saggio continuo accoppiato all’endonucleasi, la velocità iniziale viene determinata in presenza e assenza di alcune piccole molecole. Ogni potenziale inibitore viene esaminato a due concentrazioni per cercare l’inibizione DNMT1 dipendente dalla concentrazione. La percentuale di attività osservata in presenza delle piccole molecole è stata calcolata in ciascun caso.
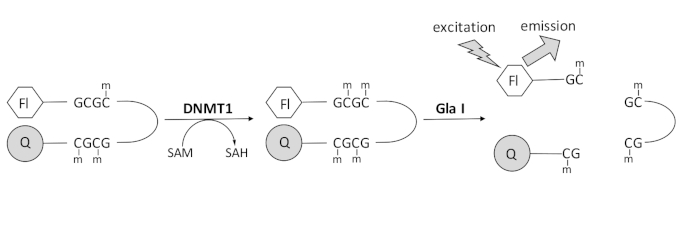
Figura 1: Saggio di metilazione del DNA. Come substrato viene utilizzato un DNA a forcina emimetilata con un fluoroforo all’estremità 5′ e un quencher all’estremità 3′. DNMT1 catalizza il trasferimento del gruppo metilico dalla S-adenosilmetionina al sito CpG non metilato, generando S-adenosilomocisteina e DNA completamente metilato. Il prodotto del DNA contiene il sito di scissione per l’endonucleasi Gla I, che fende i siti GCGC completamente metilati. La scissione del DNA del prodotto rilascia il fluoroforo 5′ dal quencher 3′, generando fluorescenza. Abbreviazioni: Fl = fluoroforo; Q = quencher; DNMT1 = DNA metiltransferasi 1; SAM = S-adenosilmetionina; SAH = S-adenosilomocisteina. Fare clic qui per visualizzare una versione ingrandita di questa figura.
Protocol
Representative Results
Discussion
Per identificare e caratterizzare gli inibitori delle DNA metiltransferasi, deve essere misurata l’attività dell’enzima. Esistono diversi metodi per esaminare l’attività della DNA metiltransferasi. L’attività è comunemente monitorata utilizzando la radioattività; il trasferimento del gruppo metilico marcato di SAM può essere quantificato 29,30,31,32,33,34.</su…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Gli autori ringraziano la Bucknell University e il Dipartimento di Chimica per il loro sostegno a questo lavoro.
Materials
| 96-well Half Area Black Flat Bottom Polystyrene Not Treated Microplate | Corning | 3694 | |
| 96-Well Polystyrene Conical Bottom Plates | ThermoFisher | 249570 | |
| Bovine Serum Albumin | NEB | B9000S | |
| compound 1 | ChemBridge | 5812086 | screening compound; resuspended in DMSO to 10 mM |
| compound 2 | ChemBridge | 6722175 | screening compound; resuspended in DMSO to 10 mM |
| compound 3 | ChemBridge | 5249376 | screening compound; resuspended in DMSO to 10 mM |
| Dithiothreitol | Sigma | D0632 | |
| Gla I | SibEnzyme | E494 | methyl-sensitive endonuclease |
| Glycerol | RPI | G22025 | |
| Magnesium Chloride | Sigma | M0250 | |
| Oligonucleotide (5'-FAM-CCTATGCGmCATCAGTTTTCTGATGmCGmCATAGG-3'-Iowa Black Quencher) | IDT | custom synthesized | internally quenched hairpin DNA (substrate) |
| Potassium Glutamate | Sigma | G1501 | |
| S-adenosylmethionine | Sigma | A4377 | methyl-donating co-factor (substrate) |
| Tris Base | RPI | T60040 |
References
- Jurkowska, R. Z., Jurkowski, T. P., Jeltsch, A. Structure and function of mammalian DNA methyltransferases. Chembiochem. 12 (2), 206-222 (2011).
- Hamidi, T., Singh, A. K., Chen, T. Genetic alterations of DNA methylation machinery in human diseases. Epigenomics. 7 (2), 247-265 (2015).
- Norvil, A. B., Saha, D., Dar, M. S., Gowher, H. Effect of disease-associated germline mutations on structure function relationship of DNA methyltransferases. Genes. 10 (5), 369 (2019).
- Foulks, J. M., et al. Epigenetic drug discovery: targeting DNA methyltransferases. Journal of Biomolecular Screening. 17 (1), 2-17 (2012).
- Goll, M. G., Bestor, T. H. Eukaryotic cytosine methyltransferases. Annual Review of Biochemistry. 74, 481-514 (2005).
- Bigey, P., Ramchandani, S., Theberge, J., Araujo, F. D., Szyf, M. Transcriptional regulation of the human DNA Methyltransferase (dnmt1) gene. Gene. 242 (1-2), 407-418 (2000).
- Detich, N., Ramchandani, S., Szyf, M. A conserved 3′-untranslated element mediates growth regulation of DNA methyltransferase 1 and inhibits its transforming activity. Journal of Biological Chemistry. 276 (27), 24881-24890 (2001).
- MacLeod, A. R., Rouleau, J., Szyf, M. Regulation of DNA methylation by the Ras signaling pathway. Journal of Biological Chemistry. 270 (19), 11327-11337 (1995).
- Slack, A., Cervoni, N., Pinard, M., Szyf, M. DNA methyltransferase is a downstream effector of cellular transformation triggered by simian virus 40 large T antigen. Journal of Biological Chemistry. 274 (15), 10105-10112 (1999).
- Eads, C. A., Nickel, A. E., Laird, P. W. Complete genetic suppression of polyp formation and reduction of CpG-island hypermethylation in Apc(Min/+) Dnmt1-hypomorphic mice. Cancer Research. 62, 1296-1299 (2002).
- MacLeod, A. R., Szyf, M. Expression of antisense to DNA methyltransferase mRNA induces DNA demethylation and inhibits tumorigenesis. Journal of Biological Chemistry. 270 (14), 8037-8043 (1995).
- Ramchandani, S., MacLeod, A. R., Pinard, M., von Hofe, E., Szyf, M. Inhibition of tumorigenesis by a cytosine-DNA, methyltransferase, antisense oligodeoxynucleotide. Proceedings of the National Academy Sciences of the United States of America. 94 (2), 684-689 (1997).
- Ley, T. J., et al. DNMT3A mutations in acute myeloid leukemia. New England Journal of Medicine. 363, 2424-2433 (2010).
- Walter, M. J., et al. Recurrent DNMT3A mutations in patients with myelodysplastic syndromes. Leukemia. 25 (7), 1153-1158 (2011).
- Russler-Germain, D. A., et al. The R882H DNMT3A mutation associated with AML dominantly inhibits wild-type DNMT3A by blocking its ability to form active tetramers. Cancer Cell. 25 (4), 442-454 (2014).
- Roll, J. D., Rivenbark, A. G., Jones, W. D., Coleman, W. B. DNMT3b overexpression contributes to a hypermethylator phenotype in human breast cancer cell lines. Molecular Cancer. 7, 15 (2008).
- Nosho, K., et al. DNMT3B expression might contribute to CpG island methylator phenotype in colorectal cancer. Clinical Cancer Research. 15 (11), 3663-3671 (2009).
- Erdmann, A., Halby, L., Fahy, J., Arimondo, P. B. Targeting DNA methylation with small molecules: what’s next. Journal of Medicinal Chemistry. 58 (6), 2569-2583 (2015).
- Chuang, J. C., et al. S110, a 5-Aza-2′-deoxycytidine-containing dinucleotide, is an effective DNA methylation inhibitor in vivo and can reduce tumor growth. Molecular Cancer Therapeutics. 9 (5), 1443-1450 (2010).
- Issa, J. J., et al. Safety and tolerability of guadecitabine (SGI-110) in patients with myelodysplastic syndrome and acute myeloid leukaemia: a multicentre, randomised, dose-escalation phase 1 study. Lancet Oncology. 16 (9), 1099-1110 (2015).
- Datta, J., et al. A new class of quinoline-based DNA hypomethylating agents reactivates tumor suppressor genes by blocking DNA methyltransferase 1 activity and inducing its degradation. Cancer Research. 69 (10), 4277-4285 (2009).
- Zambrano, P., et al. A phase I study of hydralazine to demethylate and reactivate the expression of tumor suppressor genes. BMC Cancer. 5, 44 (2005).
- Lee, B. H., Yegnasubramanian, S., Lin, X., Nelson, W. G. Procainamide is a specific inhibitor of DNA methyltransferase 1. Journal of Biological Chemistry. 280 (49), 40749-40756 (2005).
- Asgatay, S., et al. Synthesis and evaluation of analogues of N-phthaloyl-l-tryptophan (RG108) as inhibitors of DNA methyltransferase 1. Journal of Medicinal Chemstry. 57 (2), 421-434 (2014).
- Ceccaldi, A., et al. C5-DNA methyltransferase inhibitors: from screening to effects on zebrafish embryo development. Chembiochem. 12 (9), 1337-1345 (2011).
- Fagan, R. L., Wu, M., Chédin, F., Brenner, C. An ultrasensitive high throughput screen for DNA methyltransferase 1-targeted molecular probes. PLoS One. 8 (11), 78752 (2013).
- Fagan, R. L., Cryderman, D. E., Kopelovich, L., Wallrath, L. L., Brenner, C. Laccaic acid A is a direct, DNA-competitive inhibitor of DNA methyltransferase 1. Journal of Biological Chemistry. 288 (33), 23858-23867 (2013).
- Eglen, R. M., Reisine, T. Screening for compounds that modulate epigenetic regulation of the transcriptome: an overview. Journal of Biomolecular Screening. 16 (10), 1137-1152 (2011).
- Holz-Schietinger, C., Matje, D. M., Reich, N. O. Mutations in DNA methyltransferase (DNMT3A) observed in acute myeloid leukemia patients disrupt processive methylation. Journal of Biological Chemistry. 287 (37), 30941-30951 (2012).
- Norvil, A. B., et al. Dnmt3b methylates DNA by a noncooperative mechanism, and its activity is unaffected by manipulations at the predicted dimer interface. Biochemistry. 57 (29), 4312-4324 (2018).
- Bashtrykov, P., Ragozin, S., Jeltsch, A. Mechanistic details of the DNA recognition by the Dnmt1 DNA methyltransferase. FEBS Letters. 586 (13), 1821-1823 (2012).
- Bashtrykov, P., et al. Targeted mutagenesis results in an activation of DNA methyltransferase 1 and confirms an autoinhibitory role of its RFTS domain. Chembiochem. 15 (5), 743-748 (2014).
- Berkyurek, A. C., et al. The DNA methyltransferase Dnmt1 directly interacts with the SET and RING finger-associated (SRA) domain of the multifunctional protein Uhrf1 to facilitate accession of the catalytic center to hemi-methylated DNA. Journal of Biological Chemistry. 289 (1), 379-386 (2014).
- Kanada, K., Takeshita, K., Suetake, I., Tajima, S., Nakagawa, A. Conserved threonine 1505 in the catalytic domain stabilizes mouse DNA methyltransferase 1. Journal of Biochemistry. 162 (4), 271-278 (2017).
- Bashtrykov, P., et al. Specificity of Dnmt1 for methylation of hemimethylated CpG sites resides in its catalytic domain. Chemistry & Biology. 19 (5), 572-578 (2012).
- Dolen, E. K., McGinnis, J. H., Tavory, R. N., Weiss, J. A., Switzer, R. L. Disease-associated mutations G589A and V590F relieve replication focus targeting sequence-mediated autoinhibition of DNA methyltransferase 1. Biochemistry. 58 (51), 5151-5159 (2019).
- Kilgore, J. A., et al. Identification of DNMT1 selective antagonists using a novel scintillation proximity assay. Journal of Biological Chemistry. 288 (27), 19673-19684 (2013).
- Ceccaldi, A., et al. Identification of novel inhibitors of DNA methylation by screening of a chemical library. ACS Chemical Biology. 8 (3), 543-548 (2013).
- Duchin, S., Vershinin, Z., Levy, D., Aharoni, A. A continuous kinetic assay for protein and DNA methyltransferase enzymatic activities. Epigenetics & Chromatin. 8, 56 (2015).
- Syeda, F., et al. The replication focus targeting sequence (RFTS) domain is a DNA-competitive inhibitor of Dnmt1. Journal of Biological Chemistry. 286 (17), 15344-15351 (2011).
- Switzer, R. L., Medrano, J., Reedel, D. A., Weiss, J. Substituted anthraquinones represent a potential scaffold for DNA methyltransferase 1-specific inhibitors. PLoS One. 14 (7), 0219830 (2019).

