Kontinuerlig fluorescensbaserad endonukleaskopplad DNA-metyleringsanalys för att screena för DNA-metyltransferashämmare
Summary
DNA-metyltransferaser är potentiella mål för cancerläkemedel. Här presenteras ett protokoll för att bedöma små molekyler för DNA-metyltransferashämning. Denna analys använder ett endonukleas för att koppla DNA-metylering till fluorescensgenerering och gör det möjligt att övervaka enzymaktivitet i realtid.
Abstract
DNA-metylering, en form av epigenetisk genreglering, är viktig för normal cellulär funktion. I celler etablerar och upprätthåller proteiner som kallas DNA-metyltransferaser (DNMT) DNA-metyleringsmönstret. Förändringar i det normala DNA-metyleringsmönstret är kopplade till cancerutveckling och progression, vilket gör DNMT till potentiella cancerläkemedelsmål. Således är det av stor betydelse att identifiera och karakterisera nya småmolekylära hämmare av dessa enzymer. Detta dokument presenterar ett protokoll som kan användas för att screena för DNA-metyltransferashämmare. Den kontinuerliga kopplade kinetikanalysen gör det möjligt att bestämma initiala hastigheter för DNA-metylering i närvaro och frånvaro av potentiella småmolekylära hämmare. Analysen använder det metylkänsliga endonukleaset Gla I för att koppla metylering av ett hemimetylerat DNA-substrat till fluorescensgenerering.
Denna kontinuerliga analys gör det möjligt att övervaka enzymaktiviteten i realtid. Genomförande av analysen i små volymer i mikrotiterplattor minskar kostnaden för reagenser. Med hjälp av denna analys utfördes en liten exempelskärm för hämmare av DNMT1, det vanligaste DNMT-isozymet hos människor. Den mycket substituerade naturprodukten antrakinon, lakcainsyra A, är en potent, DNA-konkurrenskraftig hämmare av DNMT1. Här undersöker vi tre potentiella småmolekylära hämmare – antrakinoner eller antrakinonliknande molekyler med en till tre substituenter – i två koncentrationer för att beskriva analysprotokollet. Initialhastigheter används för att beräkna den procentuella aktiviteten som observerats i närvaro av varje molekyl. En av tre undersökta föreningar uppvisar koncentrationsberoende hämning av DNMT1-aktivitet, vilket indikerar att det är en potentiell hämmare av DNMT1.
Introduction
DNA-metylering är ett viktigt epigenetiskt märke som reglerar genuttryck och kromatinstruktur. Metylering sker huvudsakligen i CpG-dinukleotider – cytosin följt av guanosin; Metylgruppen tillsätts till 5-positionen av cytosin. Korrekta DNA-metyleringsmönster, och därmed korrekt genuttryck, behövs för lämplig cellulär utveckling och funktion. Många sjukdomstillstånd har associerats med förändringar i det normala metyleringsmönstret 1,2,3. Till exempel finns det en koppling mellan cancerinitiering och progression och förändringar i DNA-metyleringsmönstret. Vanligtvis uppvisar cancerceller lägre totala nivåer av metylcytosin, vilket bidrar till genominstabilitet. Samtidigt koncentreras metylcytosinet som är närvarande i genomet i promotorregionerna av tumörsuppressorgener, vilket leder till gendämpning av dessa viktiga proteiner. I synnerhet är epigenetiska förändringar dynamiska och reversibla, till skillnad från DNA-mutationerna associerade med tumorigenes. Detta har gjort proteinerna som är involverade i epigenetisk genreglering intressanta läkemedelsmål 2,4.
DNA-metyltransferaser (DNMT) är de proteiner som är ansvariga för att generera och upprätthålla DNA-metyleringsmönster. Tre katalytiskt aktiva isozymer, DNMT1, DNMT3a och DNMT3b, finns hos människor. Under utveckling och differentiering etablerar de novo-metyltransferaserna, DNMT3a och DNMT3b, metyleringsmönster. Båda enzymerna kan binda det katalytiskt inaktiva DNMT3L-proteinet för att bilda komplex som uppvisar ökad aktivitet 1,5. Efter celldelning innehåller dotterceller hemimetylerat DNA – DNA som innehåller metylcytosin i endast en sträng av duplexen – eftersom det nyligen syntetiserade DNA saknar metyleringsmärken. Den huvudsakliga funktionen hos DNMT1 är att metylera detta hemimetylerade DNA och därmed återupprätta det fullständiga metyleringsmönstret 1,5.
Kopplingar mellan DNMT-aktivitet och cancer är väl etablerade. Överuttryck av DNMT1, antingen genom transkriptionella eller post-translationella mekanismer, är en följd av flera vanliga onkogena vägar 6,7,8,9. Genetiska tillvägagångssätt för att sänka DNMT1-aktivitet med hypomorfa alleler resulterar i minskad tumörbildning hos Apc (Min) möss10. Antisense oligonukleotider som slår ner DNMT1 hämmar neoplasi i cellodling och mustumörmodeller11,12. Således verkar hämmande DNMT1-aktivitet som en lovande cancerterapimetod. De roller som DNMT3-isozymerna spelar är dock inte så enkla. DNMT3a-mutationer finns i akut myeloisk leukemi13 och myelodysplastiskt syndrom14. Minst en av de identifierade mutationerna har visat sig minska DNA-metyleringsaktiviteten hos enzymet15. DNMT3b är dock överuttryckt i bröstcancer16 och kolorektal cancer17. Eftersom de olika DNMT-isozymerna spelar olika roller i cancerframkallande kommer det att vara avgörande att identifiera isozymspecifika hämmare. Dessa föreningar kommer inte bara att vara användbara för utveckling av terapier, utan isozymspecifika hämmare skulle också vara ett ovärderligt verktyg för att dissekera rollen för varje DNMT-isozym i canceretiologi.
Flera DNMT-hämmare har rapporterats i litteraturen. Kända DNMT-hämmare kan delas in i två klasser: nukleosid och icke-nukleosid. Nukleosidhämmare är vanligtvis cytidinanaloger. Dessa föreningar införlivas i DNA och kovalent fälla DNMTs. 5-azacytidin och 5-aza-2′-deoxycytidin har godkänts för behandling av myelodysplastiskt syndrom och akut myeloisk leukemi 4,18. Den höga toxiciteten, låg biotillgänglighet, och kemisk instabilitet av dessa föreningar ger problem. Pågående arbete undersöker effekten av nästa generations nukleosidhämmare; SGI-110, härledd från 5-aza-2′-deoxicytidin, är ett exempel19,20. Nukleosidhämmare är inte isozymspecifika och kommer att inaktivera alla DNMT-isozym som påträffas. Därför resulterar behandling med ett nukleosiddemetyleringsmedel i uttömning av alla DNMT-isozymer 4,18. Icke-nukleosidhämmare behöver inte införlivas i DNA för att utöva sina hämmande effekter. Istället binder dessa molekyler direkt till DNMT, vilket introducerar möjligheten till isozymspecifik hämning. Flera icke-nukleosidhämmare har hittills upptäckts, inklusive SGI-1027 21, hydralazin 22, prokainamid23, RG108 och derivat 24, och naturliga produkter, (−)-epigallocatechin 3-gallat (EGCG)25 och lakcainsyra A 26,27. De flesta av de icke-nukleosidhämmare som hittills upptäckts är inte isozymeselektiva eller visar svaga preferenser för ett DNMT-isozym. Dessutom måste styrkan hos dessa molekyler förbättras, särskilt i cellerna 4,18. Således finns det ett behov av att upptäcka eller utveckla mer potenta, isozymselektiva DNMT-hämmare.
Ett hinder för att upptäcka nya småmolekylära hämmare av DNMT är de mödosamma analyser som traditionellt används för att undersöka DNMT-aktivitet28. Analyser är vanligtvis diskontinuerliga med flera steg. Den enzymatiska aktiviteten hos DNMT analyseras fortfarande rutinmässigt med radioaktivt S-adenosylmetionin (SAM)29,30,31,32,33,34. Icke-radioaktiva analyser för DNA-metylering har också utvecklats. Till exempel har analyser som använder metylkänsliga restriktionsendonukleaser och elektrofores för att separera matsmältningsprodukterna beskrivits35,36. Dessa typer av diskontinuerliga, flerstegsanalyser är inte lätt mottagliga för läkemedelsupptäckt. Sedan mitten av 2000-talet har flera DNA-metyleringsanalyser med högre genomströmning utvecklats28. En scintillationsnärhetsanalys användes för att screena för DNMT1-hämmare37. En annan analys som använder ett metylkänsligt restriktionsendonukleas användes för att screena för DNMT3a-hämmare25,38. Medan båda analyserna möjliggjorde högre genomströmning än traditionella DNA-metyleringsanalyser, kräver analyserna flera steg och tillåter inte observation av metyleringsaktivitet i realtid. På senare tid har en kontinuerlig kinetikanalys beskrivits som kopplar bildningen av S-adenosylhomocystein (SAH), en produkt av metyleringsreaktionen, till den spektroskopiska förändringen vid 340 nm associerad med NADPH-oxidation39. Denna analys använder tre kopplingsenzymer för att generera en spektroskopisk signal.
Vi utvecklade en fluorescensbaserad endonukleaskopplad DNA-metyleringsanalys som använder ett enda kommersiellt tillgängligt kopplingsenzym och kan generera data i realtid (figur 1). En hårnåloligonukleotid innehållande tre metylcytosiner används som substrat. Substrat-DNA innehåller en fluorofor på 5′-änden och en släckare på 3′-änden. Metylering av det hemimetylerade CpG-stället genererar klyvningsstället för endonukleas Gla I – helt metylerad GCGC. Gla I-klyvning av produkten oligonukleotid frigör fluoroforen från släckaren och genererar fluorescens i realtid. Analysen kan användas för att undersöka aktiviteten hos vilken isoform av DNMT som helst; emellertid observeras högre aktivitet med DNMT1 eftersom detta isozym företrädesvis metylerar hemimetylerat DNA 1,5. Ännu mer robust aktivitet observeras om den autoinhibitoriska RFTS-domänen (Replication Foci Targeting Sequence) tas bort från DNMT1. Denna domän, som finns i N-terminalregleringsregionen, binder till den katalytiska platsen och förhindrar DNA-bindning. Avlägsnande av de första ~ 600 aminosyrorna resulterar i ett stympat enzym som är signifikant mer aktivt än fullängdsenzymet (~ 640-faldig ökning av kkatt / Km) 40. Denna aktiverade form av enzymet, kallad RFTS-saknad DNMT1 (aminosyror 621–1616), möjliggör enklare identifiering av hämmare på grund av dess ökade katalytiska kraft. Detta dokument presenterar ett protokoll för att använda RFTS-saknade DNMT1 i analyser för att screena för potentiella småmolekylära hämmare. Med hjälp av den endonukleaskopplade kontinuerliga analysen bestäms initialhastigheten i närvaro och frånvaro av några små molekyler. Varje potentiell hämmare undersöks vid två koncentrationer för att leta efter koncentrationsberoende DNMT1-hämning. Den procentuella aktiviteten som observerades i närvaro av de små molekylerna beräknades i varje enskilt fall.
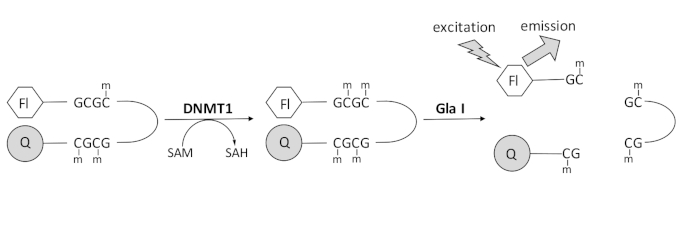
Figur 1: DNA-metyleringsanalys. Ett hemimetylerat hårnåls-DNA med en fluorofor i 5′-änden och en släckare på 3′-änden används som substrat. DNMT1 katalyserar överföringen av metylgruppen från S-adenosylmetionin till det icke-metylerade CpG-stället, vilket genererar S-adenosylhomocystein och fullständigt metylerat DNA. DNA-produkten innehåller klyvningsstället för endonukleas Gla I, som klyver helt metylerade GCGC-platser. Klyvning av produkten DNA frigör 5′-fluoroforen från 3′-släckaren och genererar fluorescens. Förkortningar: Fl = fluorofor; Q =släckare; DNMT1 = DNA-metyltransferas 1; SAM = S-adenosylmetionin; SAH = S-adenosylhomocystein. Klicka här för att se en större version av denna siffra.
Protocol
Representative Results
Discussion
För att identifiera och karakterisera hämmare av DNA-metyltransferaser måste enzymets aktivitet mätas. Det finns flera metoder för att undersöka DNA-metyltransferasaktivitet. Aktiviteten övervakas vanligtvis med hjälp av radioaktivitet; överföring av den märkta metylgruppen av SAM kan kvantifieras 29,30,31,32,33,34…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Författarna tackar Bucknell University och Institutionen för kemi för deras stöd för detta arbete.
Materials
| 96-well Half Area Black Flat Bottom Polystyrene Not Treated Microplate | Corning | 3694 | |
| 96-Well Polystyrene Conical Bottom Plates | ThermoFisher | 249570 | |
| Bovine Serum Albumin | NEB | B9000S | |
| compound 1 | ChemBridge | 5812086 | screening compound; resuspended in DMSO to 10 mM |
| compound 2 | ChemBridge | 6722175 | screening compound; resuspended in DMSO to 10 mM |
| compound 3 | ChemBridge | 5249376 | screening compound; resuspended in DMSO to 10 mM |
| Dithiothreitol | Sigma | D0632 | |
| Gla I | SibEnzyme | E494 | methyl-sensitive endonuclease |
| Glycerol | RPI | G22025 | |
| Magnesium Chloride | Sigma | M0250 | |
| Oligonucleotide (5'-FAM-CCTATGCGmCATCAGTTTTCTGATGmCGmCATAGG-3'-Iowa Black Quencher) | IDT | custom synthesized | internally quenched hairpin DNA (substrate) |
| Potassium Glutamate | Sigma | G1501 | |
| S-adenosylmethionine | Sigma | A4377 | methyl-donating co-factor (substrate) |
| Tris Base | RPI | T60040 |
References
- Jurkowska, R. Z., Jurkowski, T. P., Jeltsch, A. Structure and function of mammalian DNA methyltransferases. Chembiochem. 12 (2), 206-222 (2011).
- Hamidi, T., Singh, A. K., Chen, T. Genetic alterations of DNA methylation machinery in human diseases. Epigenomics. 7 (2), 247-265 (2015).
- Norvil, A. B., Saha, D., Dar, M. S., Gowher, H. Effect of disease-associated germline mutations on structure function relationship of DNA methyltransferases. Genes. 10 (5), 369 (2019).
- Foulks, J. M., et al. Epigenetic drug discovery: targeting DNA methyltransferases. Journal of Biomolecular Screening. 17 (1), 2-17 (2012).
- Goll, M. G., Bestor, T. H. Eukaryotic cytosine methyltransferases. Annual Review of Biochemistry. 74, 481-514 (2005).
- Bigey, P., Ramchandani, S., Theberge, J., Araujo, F. D., Szyf, M. Transcriptional regulation of the human DNA Methyltransferase (dnmt1) gene. Gene. 242 (1-2), 407-418 (2000).
- Detich, N., Ramchandani, S., Szyf, M. A conserved 3′-untranslated element mediates growth regulation of DNA methyltransferase 1 and inhibits its transforming activity. Journal of Biological Chemistry. 276 (27), 24881-24890 (2001).
- MacLeod, A. R., Rouleau, J., Szyf, M. Regulation of DNA methylation by the Ras signaling pathway. Journal of Biological Chemistry. 270 (19), 11327-11337 (1995).
- Slack, A., Cervoni, N., Pinard, M., Szyf, M. DNA methyltransferase is a downstream effector of cellular transformation triggered by simian virus 40 large T antigen. Journal of Biological Chemistry. 274 (15), 10105-10112 (1999).
- Eads, C. A., Nickel, A. E., Laird, P. W. Complete genetic suppression of polyp formation and reduction of CpG-island hypermethylation in Apc(Min/+) Dnmt1-hypomorphic mice. Cancer Research. 62, 1296-1299 (2002).
- MacLeod, A. R., Szyf, M. Expression of antisense to DNA methyltransferase mRNA induces DNA demethylation and inhibits tumorigenesis. Journal of Biological Chemistry. 270 (14), 8037-8043 (1995).
- Ramchandani, S., MacLeod, A. R., Pinard, M., von Hofe, E., Szyf, M. Inhibition of tumorigenesis by a cytosine-DNA, methyltransferase, antisense oligodeoxynucleotide. Proceedings of the National Academy Sciences of the United States of America. 94 (2), 684-689 (1997).
- Ley, T. J., et al. DNMT3A mutations in acute myeloid leukemia. New England Journal of Medicine. 363, 2424-2433 (2010).
- Walter, M. J., et al. Recurrent DNMT3A mutations in patients with myelodysplastic syndromes. Leukemia. 25 (7), 1153-1158 (2011).
- Russler-Germain, D. A., et al. The R882H DNMT3A mutation associated with AML dominantly inhibits wild-type DNMT3A by blocking its ability to form active tetramers. Cancer Cell. 25 (4), 442-454 (2014).
- Roll, J. D., Rivenbark, A. G., Jones, W. D., Coleman, W. B. DNMT3b overexpression contributes to a hypermethylator phenotype in human breast cancer cell lines. Molecular Cancer. 7, 15 (2008).
- Nosho, K., et al. DNMT3B expression might contribute to CpG island methylator phenotype in colorectal cancer. Clinical Cancer Research. 15 (11), 3663-3671 (2009).
- Erdmann, A., Halby, L., Fahy, J., Arimondo, P. B. Targeting DNA methylation with small molecules: what’s next. Journal of Medicinal Chemistry. 58 (6), 2569-2583 (2015).
- Chuang, J. C., et al. S110, a 5-Aza-2′-deoxycytidine-containing dinucleotide, is an effective DNA methylation inhibitor in vivo and can reduce tumor growth. Molecular Cancer Therapeutics. 9 (5), 1443-1450 (2010).
- Issa, J. J., et al. Safety and tolerability of guadecitabine (SGI-110) in patients with myelodysplastic syndrome and acute myeloid leukaemia: a multicentre, randomised, dose-escalation phase 1 study. Lancet Oncology. 16 (9), 1099-1110 (2015).
- Datta, J., et al. A new class of quinoline-based DNA hypomethylating agents reactivates tumor suppressor genes by blocking DNA methyltransferase 1 activity and inducing its degradation. Cancer Research. 69 (10), 4277-4285 (2009).
- Zambrano, P., et al. A phase I study of hydralazine to demethylate and reactivate the expression of tumor suppressor genes. BMC Cancer. 5, 44 (2005).
- Lee, B. H., Yegnasubramanian, S., Lin, X., Nelson, W. G. Procainamide is a specific inhibitor of DNA methyltransferase 1. Journal of Biological Chemistry. 280 (49), 40749-40756 (2005).
- Asgatay, S., et al. Synthesis and evaluation of analogues of N-phthaloyl-l-tryptophan (RG108) as inhibitors of DNA methyltransferase 1. Journal of Medicinal Chemstry. 57 (2), 421-434 (2014).
- Ceccaldi, A., et al. C5-DNA methyltransferase inhibitors: from screening to effects on zebrafish embryo development. Chembiochem. 12 (9), 1337-1345 (2011).
- Fagan, R. L., Wu, M., Chédin, F., Brenner, C. An ultrasensitive high throughput screen for DNA methyltransferase 1-targeted molecular probes. PLoS One. 8 (11), 78752 (2013).
- Fagan, R. L., Cryderman, D. E., Kopelovich, L., Wallrath, L. L., Brenner, C. Laccaic acid A is a direct, DNA-competitive inhibitor of DNA methyltransferase 1. Journal of Biological Chemistry. 288 (33), 23858-23867 (2013).
- Eglen, R. M., Reisine, T. Screening for compounds that modulate epigenetic regulation of the transcriptome: an overview. Journal of Biomolecular Screening. 16 (10), 1137-1152 (2011).
- Holz-Schietinger, C., Matje, D. M., Reich, N. O. Mutations in DNA methyltransferase (DNMT3A) observed in acute myeloid leukemia patients disrupt processive methylation. Journal of Biological Chemistry. 287 (37), 30941-30951 (2012).
- Norvil, A. B., et al. Dnmt3b methylates DNA by a noncooperative mechanism, and its activity is unaffected by manipulations at the predicted dimer interface. Biochemistry. 57 (29), 4312-4324 (2018).
- Bashtrykov, P., Ragozin, S., Jeltsch, A. Mechanistic details of the DNA recognition by the Dnmt1 DNA methyltransferase. FEBS Letters. 586 (13), 1821-1823 (2012).
- Bashtrykov, P., et al. Targeted mutagenesis results in an activation of DNA methyltransferase 1 and confirms an autoinhibitory role of its RFTS domain. Chembiochem. 15 (5), 743-748 (2014).
- Berkyurek, A. C., et al. The DNA methyltransferase Dnmt1 directly interacts with the SET and RING finger-associated (SRA) domain of the multifunctional protein Uhrf1 to facilitate accession of the catalytic center to hemi-methylated DNA. Journal of Biological Chemistry. 289 (1), 379-386 (2014).
- Kanada, K., Takeshita, K., Suetake, I., Tajima, S., Nakagawa, A. Conserved threonine 1505 in the catalytic domain stabilizes mouse DNA methyltransferase 1. Journal of Biochemistry. 162 (4), 271-278 (2017).
- Bashtrykov, P., et al. Specificity of Dnmt1 for methylation of hemimethylated CpG sites resides in its catalytic domain. Chemistry & Biology. 19 (5), 572-578 (2012).
- Dolen, E. K., McGinnis, J. H., Tavory, R. N., Weiss, J. A., Switzer, R. L. Disease-associated mutations G589A and V590F relieve replication focus targeting sequence-mediated autoinhibition of DNA methyltransferase 1. Biochemistry. 58 (51), 5151-5159 (2019).
- Kilgore, J. A., et al. Identification of DNMT1 selective antagonists using a novel scintillation proximity assay. Journal of Biological Chemistry. 288 (27), 19673-19684 (2013).
- Ceccaldi, A., et al. Identification of novel inhibitors of DNA methylation by screening of a chemical library. ACS Chemical Biology. 8 (3), 543-548 (2013).
- Duchin, S., Vershinin, Z., Levy, D., Aharoni, A. A continuous kinetic assay for protein and DNA methyltransferase enzymatic activities. Epigenetics & Chromatin. 8, 56 (2015).
- Syeda, F., et al. The replication focus targeting sequence (RFTS) domain is a DNA-competitive inhibitor of Dnmt1. Journal of Biological Chemistry. 286 (17), 15344-15351 (2011).
- Switzer, R. L., Medrano, J., Reedel, D. A., Weiss, J. Substituted anthraquinones represent a potential scaffold for DNA methyltransferase 1-specific inhibitors. PLoS One. 14 (7), 0219830 (2019).

