Sistema microfisiologico epatico umano per la valutazione della tossicità epatica indotta da farmaci in vitro
Summary
Il danno epatico indotto da farmaci (DILI) è una delle principali cause di fallimento farmacologico. È stato sviluppato un protocollo per prevedere con precisione la responsabilità DILI di un composto utilizzando un sistema microfisiologico epatico (MPS). Il modello epatico utilizza la cocoltura di cellule epatiche primarie ed endpoint traslazionali rilevanti per valutare le risposte cellulari al trattamento.
Abstract
DILI è una delle principali cause di logoramento nello sviluppo di farmaci con oltre 1000 farmaci approvati dalla FDA noti per causare potenzialmente DILI negli esseri umani. Sfortunatamente, il DILI spesso non viene rilevato fino a quando i farmaci non hanno raggiunto le fasi cliniche, mettendo a rischio la sicurezza dei pazienti e portando a perdite sostanziali per l’industria farmaceutica. Tenendo conto del fatto che i modelli 2D standard hanno limitazioni nel rilevamento di DILI, è essenziale sviluppare modelli in vitro che siano più predittivi per migliorare la traducibilità dei dati. Per comprendere in dettaglio la causalità e gli aspetti meccanicistici del DILI, è stata sviluppata una MPS epatica umana costituita da cellule parichimale e non parenchimali (NPC) epatiche primarie umane e coltivate in microtessuti 3D su un’impalcatura ingegnerizzata sotto perfusione. Gli epatociti umani primari crioconservati (PHH) e le cellule di Kupffer (HKC) sono stati cocoltivati come microtessuti nella piattaforma MPS per un massimo di due settimane e ogni composto di interesse è stato ripetutamente dosato su microtessuti epatici a sette concentrazioni di prova per un massimo di quattro giorni. Sono stati analizzati gli endpoint funzionali specifici del fegato (inclusi biomarcatori clinici come l’alanina aminotransferasi, ALT) per valutare la funzionalità epatica. L’esposizione acuta e cronica a composti di varia gravità DILI può essere valutata confrontando le risposte a microtessuti monodose e multidose. La metodologia è stata validata con un’ampia gamma di composti gravi e lievemente epatotossici. Qui mostriamo i dati relativi a pioglitazone e troglitazone, noti composti epatotossici ritirati dal mercato per aver causato insufficienza epatica. Nel complesso, è stato dimostrato che il modello MPS epatico può essere uno strumento utile per valutare la DILI e la sua associazione con i cambiamenti nella funzionalità epatica. Il modello può inoltre essere utilizzato per valutare come si comportano nuovi composti in sottogruppi di pazienti distinti e come i profili di tossicità possono essere influenzati da stati di malattia epatica (ad esempio, epatite virale, malattia del fegato grasso).
Introduction
DILI rimane la causa più comune di insufficienza epatica acuta negli Stati Uniti e in Europa ed è una delle principali cause di attrito dei composti nel processo di sviluppo del farmaco1. Quasi tutte le classi di farmaci possono causare epatotossicità, con agenti del sistema nervoso centrale e antibiotici che sono di gran lunga i trattamenti più comuni che causano DILI nei pazienti2. L’epatotossicità indotta da farmaci è causata da una complessa interazione di fattori genetici, non genetici e ambientali, che porta alla morte degli epatociti e di altri tipi di cellule del fegato, inclusi colangiociti e cellule endoteliali 1,3.
Gli agenti che causano DILI possono essere classificati in due modi: quelli che causano danni epatici dose-dipendenti prevedibili o quelli che causano DILI idiosincratico che è raro e si sviluppa indipendentemente dalla dose del farmaco, o via, o durata della somministrazione, ma è responsabile fino a un sesto di tutti gli errori epatici acuti negli Stati Uniti solo4. Sfortunatamente, DILI spesso non viene rilevato fino a quando i farmaci non hanno raggiunto le fasi cliniche del processo di sviluppo del farmaco. Il grado di danno epatico indotto da farmaci (o DILIrank) consiste di oltre un migliaio di farmaci approvati dalla FDA che sono divisi in quattro classi in base al loro potenziale di causare DILI e il loro uso nei pazienti deve essere attentamente monitorato5.
Lo studio dei meccanismi di epatotossicità dei farmaci rimane molto impegnativo e, pertanto, sono stati sviluppati molti modelli preclinici per esplorare i meccanismi del DILI. Gli attuali modelli in vitro e in vivo utilizzati per prevedere DILI nello sviluppo preclinico hanno diverse limitazioni nel fornire approfondimenti sulle interazioni complesse e sfaccettate in un corpo umano vivente. Le linee cellulari epatiche cancerose (cioè HepG2, HepaRG) coltivate in 2D sono ancora utilizzate nelle prime fasi dello sviluppo di farmaci per valutare la tossicità dei composti candidati6. Anche così, queste linee cellulari provengono da singoli donatori e mostrano livelli anormali di funzionalità epatica e non sempre mostrano un’elevata sensibilità per il rilevamento di DILI 7,8. In alternativa alle linee cellulari epatiche cancerose, i PHH rappresentano meglio la fisiologia epatica umana se coltivati in modo appropriato in vitro, sebbene esistano diverse limitazioni con la loro coltura, come un breve tempo di incubazione con farmaci, una durata relativamente breve, perdita di espressione genica epatica e cambiamenti delle funzioni metaboliche dei farmaci 9,10,11 . I PHH possono essere coltivati su proteine della matrice extracellulare in piastre di coltura cellulare 2D standard, ma come svantaggio, il rapido declino della loro funzione significa che hanno una bassa sensibilità (<50%) per la previsione DILI12.
D’altra parte, i test su modelli animali sono lenti, costosi e necessitano di una traduzione interspecie per estrapolare la previsione agli esseri umani. La maggior parte dei farmaci di nuova concezione non riesce a ottenere l’approvazione, rendendo questo processo costoso e rischioso5. Inoltre, per testare nuove modalità specifiche per l’uomo, i modelli animali sono meno adatti a causa della sequenza genica o delle differenze di risposta immunologica rispetto agli esseri umani13.
Di conseguenza, l’interesse per i modelli epatici in vitro tridimensionali (3D) più avanzati è cresciuto esponenzialmente. La coltura di PHH come strutture sferoidali generate dall’aggregazione gravitazionale in gocce sospese o su superfici di attacco ultrabasse rappresenta un metodo ad alto rendimento per valutare le passività composte14. Gli sferoidi PHH sono stati utilizzati per valutare DILI in un contesto malato (ad esempio, steatosi e colestasi)15. Un’ampia varietà di modelli è stata sviluppata per includere cocolture micro-modellate placcate di epatociti con fibroblasti stromali16, tessuti epatici biostampati 3D17, colture sferoidi 3D con o senza cellule epatiche non parenchimali15. Tuttavia, tutti questi metodi presentano ancora degli svantaggi e la coltura di PHH in un microambiente più fisiologicamente rilevante potrebbe fornire loro livelli più elevati di funzionalità per lunghi periodi di tempo per consentire lo studio dell’esposizione prolungata a potenziali epatotossici. Inoltre, per migliorare la rilevanza traslazionale di qualsiasi coltura avanzata di PHH in vitro , devono essere utilizzati endpoint funzionali clinicamente rilevanti o biomarcatori di tossicità per consentire il confronto dei dati in vivo o negli scenari clinici18.
In questo studio, abbiamo valutato se un modello di fegato in vitro MPS, noto anche come Organ-on-a-Chip (OOC), potesse essere utilizzato per comprendere gli aspetti meccanicistici dettagliati della tossicità epatica. In precedenza è stato dimostrato che l’MPS mantiene microtessuti epatici 3D altamente funzionali, sotto flusso, fino a 4 settimane19. Il sistema è stato recentemente testato dalla FDA e ha dimostrato di avere un’elevata riproducibilità durante l’esecuzione di tossicità farmacologica, metabolismo e accumulo intracellulare20. Inoltre, rispetto agli sferoidi e alle colture sandwich, il sistema aveva una funzione più stabile e una maggiore sensibilità nel rilevare la tossicità di diversi farmaci20. Ad oggi, l’MPS è stato utilizzato in un’ampia gamma di applicazioni che coprono ADME 21, modelli di malattia (HBV 22, NAFLD 23,24,25) e interazioni farmaco-farmaco 26, rendendolo potenzialmente altamente adatto alla valutazione della DILI acuta e cronica. La tecnologia qui presentata offre un’alternativa per colmare il divario tra colture cellulari più tradizionali e modelli animali e studi clinici sull’uomo, avanzando verso la simulazione delle condizioni biologiche umane per supportare la valutazione della tossicità epatica dei composti candidati nelle fasi precliniche del processo di sviluppo del farmaco.
Protocol
Representative Results
Discussion
Le MPS sono progettate per ricapitolare le unità funzionali degli organi umani in vitro e sono state sviluppate per affrontare i limiti dei modelli convenzionali di coltura cellulare 3D27. Il fegato è uno degli organi più modellati utilizzando MPS e sono stati sviluppati un’ampia varietà di sistemi. Il fegato umano è responsabile del metabolismo dei farmaci e della generazione di metaboliti tossici e la sua funzione è un elemento chiave per lo sviluppo di farmaci, compresa la valutazione della responsabilità DILI dei composti28. Qui abbiamo introdotto un nuovo metodo per valutare DILI utilizzando una MPS epatica; il protocollo consente di cercare approfondimenti meccanicistici per ciascun composto analizzato per determinare come possa causare DILI oltre ad essere un test altamente sensibile e robusto. I microtessuti epatici si formano nelle piastre MPS, che sono una cocoltura di PHH e HKC e sono altamente funzionali con alti livelli di produzione di albumina e urea, nonché un’elevata attività del CYP3A4 rispetto ai modelli epatici standard in vitro 20.
Sebbene il modello DILI qui descritto possa servire come strumento utile nelle fasi successive dei test preclinici nel processo di sviluppo del farmaco, ha anche diverse limitazioni. Come la maggior parte delle MPS attualmente disponibili sul mercato, è una piattaforma a basso rendimento e, quindi, più difficile da utilizzare per attività di screening dei farmaci su larga scala. Costituito da microtessuti formati dalla cocoltura di PHH e HKC, il modello DILI non può catturare interamente la complessità del fegato umano e un’ulteriore ottimizzazione incorporando diversi tipi di cellule (ad esempio, cellule immunitarie) sarebbe utile per aggiungere valore al modello esistente. Questa MPS monoorgano potrebbe anche essere combinata con altre piattaforme d’organo che possono condividere un mezzo comune e consentire la diafonia d’organo a livello cellulare o endocrino, e che possono aiutare a comprendere meglio le intuizioni meccanicistiche della tossicità non limitata solo al fegato stesso. Inoltre, come qualsiasi tecnologia relativamente nuova, potrebbe essere considerata costosa e quindi di accessibilità limitata.
MPS è una piattaforma utilizzata per sviluppare modelli organotipici di tessuti singoli o multi-umani. Il sistema è composto da un controller, un cavo ombelicale e un driver MPS in cui è inserita la piastra (Figura 5A). Ogni piastra MPS epatica ha 12 pozzetti aperti indipendenti per la coltura di cellule epatiche primarie in 3D su scaffold ingegnerizzati. In sintesi, il sistema è controllato QC e le piastre sono innescate al giorno -1, i PHH e gli HKC sono seminati sulle piastre al giorno zero (Figura 5B, vedi 1). Le micropompe incorporate facilitano la circolazione dei terreni di coltura cellulare attraverso gli scaffold per facilitare la formazione di microtessuti 3D (Figura 5B, vedi 2). I microtessuti formati sono QC’d al Giorno 4, dosati con diverse concentrazioni di ciascun composto ogni 48 ore per 4 giorni e analizzati per i biomarcatori endpoint al Giorno 8 (Figura 5C). La cronologia sperimentale del test DILI nella piastra MPS è illustrata nella Figura 5D.
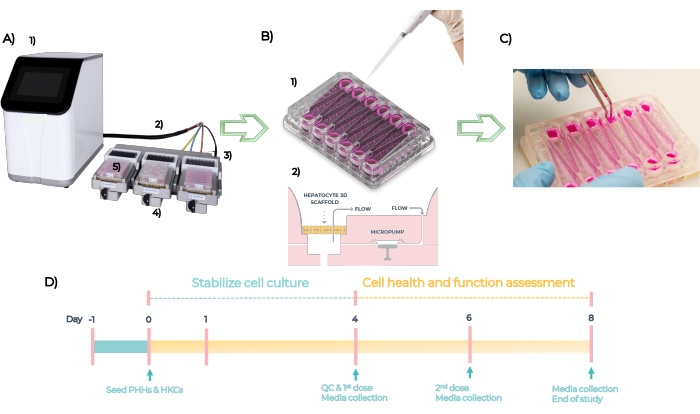
Figura 5: Il sistema microfisiologico e la cronologia sperimentale di un saggio DILI standard. (A) Il sistema microfisiologico con i suoi componenti: controller (1), cavo ombelicale (2), docking station (3), driver MPS (4) e piastra LC12 (5). (B) La semina di PHH e HKC su piastra LC12 al giorno 1 (1) e micropompe incorporate facilitano la circolazione di terreni di coltura cellulare con portate regolabili attraverso i microtessuti 3D seminati sugli scaffold (2). (C) Smontaggio dei ponteggi alla fine dello studio. (D) Cronologia sperimentale. Fare clic qui per visualizzare una versione ingrandita di questa figura.
Quando si esegue il protocollo, è importante eseguire un robusto controllo QC del sistema prima dell’avvio, controllando che il sistema funzioni correttamente pneumaticamente e che le piastre consumabili siano ispezionate visivamente e innescate in modo efficiente per garantire una funzionalità uniforme su tutti i pozzi. Avere cellule umane primarie di alta qualità è essenziale per questo protocollo, con gli epatociti noti per aderire costantemente negli esperimenti di coltura cellulare e formare interazioni 3D. Anche lo scongelamento di queste cellule è un passo critico, poiché gli epatociti primari non devono essere risospesi dall’azione del pipettaggio in quanto ciò può portare rapidamente alla morte cellulare. Avere una vitalità cellulare superiore all’85% è fondamentale per il successo della semina, poiché grandi quantità di detriti cellulari interferiscono con la formazione di microtessuti 3D. Anche il controllo QC dei microtessuti epatici formati al giorno 4 è importante e l’utente deve assicurarsi che vengano misurati livelli accettabili di LDH e urea, poiché i parametri fuori gamma potrebbero essere indicativi della formazione di tessuti di scarsa qualità e consentire una risoluzione dei problemi diretta. Infine, l’idrocortisone utilizzato nei terreni di coltura cellulare deve essere preparato fresco il giorno dell’utilizzo per prevenire qualsiasi degradazione indesiderata che potrebbe influire sulla funzionalità della coltura cellulare, in quanto è necessario per mantenere la funzionalità metabolica degli epatociti.
Pur avendo una complessità significativa, la MPS epatica non contiene tutti i tipi di cellule del fegato umano. È possibile aggiungere ulteriori tipi di cellule al modello24,29 per aumentare la rilevanza fisiologica, ma questi dovrebbero essere aggiunti solo con una chiara giustificazione per il contesto di utilizzo. Per lo studio DILI PHH sono il tipo di cellula chiave, e l’incorporazione di HKC in questo modello consente di determinare alcune risposte immunologiche. Va anche notato che i PHH isolati da fegati umani e i PHH crioconservati disponibili in commercio tendono a dimostrare alcune variazioni da lotto a lotto. Abbiamo dimostrato qui che questo protocollo produce risultati riproducibili se utilizzato con preparati di cellule di alta qualità. Tuttavia, ci si aspetterebbe qualche variazione del lotto e questo potrebbe essere ulteriormente superato utilizzando lotti raggruppati di donatori multipli. Queste limitazioni potrebbero essere superate utilizzando cellule simili agli epatociti differenziate dalle iPSC che ricapitolano molte proprietà funzionali dei PHH e che sono state utilizzate nel processo di sviluppo del farmaco30. Gli HKC mostrano anche molta variabilità e un alto livello di attivazione al momento del disgelo; pertanto, i donatori di HKC sono pre-validati internamente prima dell’uso in colture cellulari sperimentali (cocoltura con PHH convalidati) e devono avere bassi livelli di attivazione post-disgelo; questo viene valutato misurando i biomarcatori IL-6 e TNF-alfa (vedi Materiale supplementare).
I dati presentati qui confermano che il test può rilevare con precisione DILI, aiutando a identificare gli epatotossici che potrebbero non essere rilevati da 2D10,11 e persino da alcuni modelli 3D. I dati generati dalle MPS non sono ancora utilizzati come standard dall’industria farmaceutica per le richieste normative o per scopi di screening dei farmaci a causa della mancanza di standardizzazione e armonizzazione dei processi, compresa la riproducibilità tra siti20. I dati e gli approcci sperimentali qui dimostrati affrontano questo, dimostrando che il modello epatico può essere utilizzato di routine e robustamente negli schermi DILI per prevedere con precisione la responsabilità di nuovi composti.
Misurando una serie di endpoint per produrre una “firma di epatotossicità”, aiutando a identificare composti con diversi livelli di preoccupazione DILI (compresi composti non rilevabili da altri metodi in vitro) e i loro meccanismi di tossicità rivelati. Questa tecnologia può colmare il divario tra la coltura cellulare tradizionale e i modelli animali da un lato e gli studi clinici sull’uomo, avanzando verso la simulazione delle condizioni biologiche umane per la valutazione preclinica della tossicità epatica come parte del processo di sviluppo del farmaco.
Disclosures
The authors have nothing to disclose.
Acknowledgements
CN Bio Innovations Ltd. ha finanziato questo studio.
Materials
| 24 well cell culture cluster plates flat bottom | Corning | 3524 | |
| 96 well clear assay plates, flat bottom clear plastic | Greiner | 655101 | |
| 96 well plates black flat bottom | Corning | 3915 | |
| 96 Well White/Clear Bottom Plate, TC Surface | ThermoScientific | 165306 | |
| Advanced DMEM (1x) | Gibco | 12491015 | Cell culture media. |
| AssayMax Albumin ELISA Kit | AssayPro | EA3201-1 | Dilution 1:250. Time point Day 4, 6, and 8. |
| Cell Maintenance Cocktail B, (Primary Hepatocyte Maintenance Supplements) | Gibco | CM4000 | |
| CellTiter-Glo 3D Cell Viability Assay | Promega | G9682 | Dilution 1:1. Time point Day 8. |
| Chlorpromazine HCl | Sigma Aldrich | C8138 | |
| Chromacol blue lids, 9 mm Autosampler Vial Screw Thread Caps | ThermoScientific | 9-SCK(B)-ST1 | glass vial |
| Chromacol vials, 9 mm Clear Glass Screw Thread Vials | ThermoScientific | 2-SVW | |
| Class 2 Microbiological Safety Cabinets – Trimat2 1500 exhaust | Contained Air Solutions | ||
| Conical tubes 50 mL | Greiner | 227261 | |
| Cryopreserved Hepatocyte Recovery Medium (CHRM) | ThermoFisher Scientific | Gibco CM7000 | |
| Cryopreserved primary human hepatocytes | BioIVT Europe | Lot. RAS | |
| CytoTox 96 Cytotoxicty (LDH) Assay Kit | Promega | G1781 | Dilution – none. Time point Day 4, 6 and 8 |
| >Data analysis model used to generate the graph and EC:50 curves was nonlinear regression (curve fit) asymmetric sigmoidal, 5PL, where X is log(concentration | GraphPad Prism 9 | ||
| Disposable PES Filter Units 500mL | Fisher Scientific | 15913307 | |
| Disposable Pipette Basins 50ml | Fisher Scientific | 12369175 | |
| DMSO (Dimethyl sulfoxide) | Sigma-Aldrich | Sigma D2650 | |
| Dulbeco’s Phosphate Buffered Saline without Ca2+ and Mg2+ (D-PBS) | ThermoFisher Scientific | 14190-144 | |
| Easy Reader Conical Polypropylene Centrifuge Tubes 15 mL | Fisher Scientific | 11889640 | |
| Foetal bovine serum | Gibco | 10500064 | |
| Human ALT ELISA Kit | Abcam | ab 234578 | Dilution 1:5. Time point Day 6 and 8. |
| Human Cryopreserved Kupffer Cells | Lonza Europe | Lot. 190088KC | |
| hydrocortisone | Merck | H0888-1G | |
| Incubators models: New Brunswick Galaxy 170 S, New Brunswick Galaxy 170 R and CellXpert® C170. | Eppendorf | All serviced yearly; paperwork available upon request. | |
| Inverted Microscope | Leica DMIL LED | ||
| MPS know as Organ-on-a-Chip (OOC) | CN Bio Innovations Ltd. | ||
| MPS LC-12 plate | CN Bio Innovations Ltd. | ||
| Neubauer Improved C-Chip Disposable Haemocytometer (2 channel) | Cambridge Bioscience | DHC-N01-50 | |
| P450-Glo CYP3A4 Assay and Screening System | Promega | V9002 | Dilution – none. Time point Day 8 |
| PhysioMimix MPS platform | CN Bio Innovations Ltd. | ||
| Pioglitazone | MedChemExpress Tocris | HY-13956/CS-1700 | |
| Quantichrom Urea Assay Kit – Bioassay systems | Bioassay Systems | DY970-05 | Dilution 1:2 if initial reading is too high. Time point Day 4, 6 and 8. |
| Silica gel | Sigma-Aldrich | S7625 | |
| Software used to analyse and generate all the graphs was | GraphPad Prism 9 | ||
| Stripettes 10 mL | Fisher Scientific | 11839660 | |
| Stripettes 25 mL | Fisher Scientific | 11839181 | |
| Thawing plate Cocktail A, (Primary Hepatocyte Thawing and Plating Supplements) | Gibco | CM3000 | |
| Troglitazone | MedChemExpress Tocris | 97322-87-7 | |
| Trypan Blue Solution, 0.4% | Gibco | 15250061 | |
| Tubes 1.5 mL | Greiner | 616201 | |
| Weighing balance – model PA214C and AV213C | Ohaus Corp |
References
- Lisi, D. M. Drug-induced liver injury: An overview. US Pharmacist. 41 (12), 30-34 (2016).
- Kuna, L., et al. Models of drug induced liver injury (DILI)-current issues and future perspectives. Current Drug Metabolism. 19 (10), 830-838 (2018).
- Katarey, D., Verma, S. Drug-induced liver injury. Clinical Medicine. 16 (6), 104-109 (2016).
- Kullak-Ublick, G. A., et al. Drug-induced liver injury: recent advances in diagnosis and risk assessment Recent advances in clinical practice. Gut. 66, 1154-1164 (2017).
- Dirven, H., et al. Performance of pre-clinical models in predicting drug-induced liver injury in humans: a systematic review. Scientific Reports. 11 (1), 6403 (2021).
- Donato, M. T., Lahoz, A., Castell, J. V., Gomez-Lechon, M. J. Cell lines: a tool for in vitro drug metabolism studies. Current Drug Metabolism. 9 (1), 1-11 (2008).
- Wilkening, S., Stahl, F., Bader, A. Comparison of primary human hepatocytes and hepatoma cell line HepG2 with regard to their biotransformation properties. Drug Metabolism and Disposition. 31 (8), 1035-1042 (2003).
- Gerets, H. H. J., et al. Characterization of primary human hepatocytes, HepG2 cells, and HepaRG cells at the mRNA level and CYP activity in response to inducers and their predictivity for the detection of human hepatotoxins. Cell Biology and Toxicology. 28 (2), 69-87 (2012).
- Grainger, C. I., Greenwell, L. L., Lockley, D. J., Martin, G. P., Forbes, B. Culture of Calu-3 cells at the air interface provides a representative model of the airway epithelial barrier. Pharmaceutical Research. 23 (7), 1482-1490 (2006).
- Li, F., Cao, L., Parikh, S., Zuo, R. Three-dimensional spheroids with primary human liver cells and differential roles of kupffer cells in drug-induced liver injury. Journal of Pharmaceutical Sciences. 109 (6), 1912-1923 (2020).
- Proctor, W. R., et al. Utility of spherical human liver microtissues for prediction of clinical drug-induced liver injury. Archives of Toxicology. 91 (8), 2849-2863 (2017).
- Lin, C., Khetani, S. R. Advances in engineered liver models for investigating drug-induced liver injury. BioMed Research International. 2016, 1829148 (2016).
- Olson, H., et al. Concordance of the toxicity of pharmaceuticals in humans and in animals. Regulatory Toxicology and Pharmacology. 32 (1), 56-67 (2000).
- Bell, C. C., et al. Comparison of hepatic 2D sandwich cultures and 3D spheroids for long-term toxicity applications: A multicenter study. Toxicological Sciences. 162 (2), 655-666 (2018).
- Bell, C. C., et al. Characterization of primary human hepatocyte spheroids as a model system for drug-induced liver injury, liver function and disease. Scientific Reports. 6, 25187 (2016).
- Khetani, S. R., et al. Use of micropatterned co-cultures to detect compounds that cause drug-induced liver injury in humans. Toxicological Sciences. 132 (1), 107-117 (2013).
- Ma, X., et al. Deterministically patterned biomimetic human iPSC-derived hepatic model via rapid 3D bioprinting. Proceedings of the National Academy of Sciences of the united States of America. 113 (8), 2206-2211 (2016).
- Dieterle, P. Y. M., Dieterle, F. Tissue-specific, non-invasive toxicity biomarkers: translation from pre-clinical safety assessment to clinical safety monitoring. Expert Opinion on Drug Metabolism & Toxicology. 5 (9), 1023-1038 (2009).
- Rowe, C., et al. Perfused human hepatocyte microtissues identify reactive metabolite-forming and mitochondria-perturbing hepatotoxins. Toxicology in Vitro. 46, 29-38 (2018).
- Rubiano, A., et al. Characterizing the reproducibility in using a liver microphysiological system for assaying drug toxicity, metabolism, and accumulation. Clinical and Translational Science. 14 (3), 1049-1061 (2021).
- Tsamandouras, N., Kostrzewski, T., Stokes, C. L., Griffith, L. G., Hughes, D. J., Cirit, M. Quantitative assessment of population variability in hepatic drug metabolism using a perfused three-dimensional human liver microphysiological system. Journal of Pharmacology and Experimental Therapeutics. 360 (1), 95-105 (2017).
- Ortega-Prieto, A. M., et al. 3D microfluidic liver cultures as a physiological pre-clinical tool for hepatitis B virus infection. Nature Communications. 9 (1), 682 (2018).
- Kostrzewski, T., et al. Three-dimensional perfused human in vitro model of non-alcoholic fatty liver disease. World Journal of Gastroenterology. 23 (2), 204-215 (2017).
- Kostrzewski, T., et al. A microphysiological system for studying nonalcoholic steatohepatitis. Hepatology Communications. 4 (1), 77-91 (2020).
- Vacca, M., et al. Bone morphogenetic protein 8B promotes the progression of non-alcoholic steatohepatitis. Nature Metabolism. 2 (6), 514-531 (2020).
- Long, T. J., et al. Modeling therapeutic antibody-small molecule drug-drug interactions using a three-dimensional perfusable human liver co-culture platforms. Drug Metabolism and Disposition. 44, 1940-1948 (2016).
- Bai, J., Wang, C. Organoids and microphysiological systems: New tools for ophthalmic drug discovery. Frontiers in Pharmacology. 11, 407 (2020).
- Ribeiro, A. J. S., Yang, X., Patel, V., Madabushi, R., Strauss, D. G. Liver microphysiological systems for predicting and evaluating drug effects. Clinical Pharmacology & Therapeutics. 106 (1), 139-147 (2019).
- Clark, A. M., et al. A microphysiological system model of therapy for liver micrometastases hhs public access. Experimental Biology and Medicine (Maywood). 239 (9), 1170-1179 (2014).
- Qosa, H., Ribeiro, A. J. S., Hartman, N. R., Volpe, D. A. Characterization of a commercially available line of iPSC hepatocytes as models of hepatocyte function and toxicity for regulatory purposes. Journal of Pharmacological and Toxicological Methods. 110, 107083 (2021).

