Sistema microfisiológico hepático humano para evaluar la toxicidad hepática inducida por fármacos in vitro
Summary
La lesión hepática inducida por fármacos (DILI) es una causa importante de fracaso farmacológico. Se ha desarrollado un protocolo para predecir con precisión la responsabilidad DILI de un compuesto utilizando un sistema microfisiológico hepático (MPS). El modelo hepático utiliza el cocultivo de células hepáticas primarias y criterios de valoración traslacionalmente relevantes para evaluar las respuestas celulares al tratamiento.
Abstract
DILI es una causa importante de desgaste en el desarrollo de medicamentos con más de 1000 medicamentos aprobados por la FDA que se sabe que potencialmente causan DILI en humanos. Desafortunadamente, DILI a menudo no se detecta hasta que los medicamentos han alcanzado etapas clínicas, poniendo en riesgo la seguridad de los pacientes y provocando pérdidas sustanciales para la industria farmacéutica. Teniendo en cuenta que los modelos 2D estándar tienen limitaciones en la detección de DILI es esencial desarrollar modelos in vitro que sean más predictivos para mejorar la traducibilidad de los datos. Para comprender la causalidad y los aspectos mecanicistas de DILI en detalle, se ha desarrollado un MPS hepático humano que consiste en células hepáticas primarias del parénquima y no parénquima (NPC) del hígado humano y cultivadas en microtejidos 3D en un andamio diseñado bajo perfusión. Los hepatocitos humanos primarios criopreservados (PHH) y las células de Kupffer (HKC) se cocultivaron como microtejidos en la plataforma MPS durante un máximo de dos semanas, y cada compuesto de interés se dosificó repetidamente en microtejidos hepáticos a siete concentraciones de prueba durante un máximo de cuatro días. Se analizaron los criterios de valoración funcionales específicos del hígado (incluidos los biomarcadores clínicos como la alanina aminotransferasa, la ALT) para evaluar la función hepática. La exposición aguda y crónica a compuestos de diversas gravedades DILI se puede evaluar comparando las respuestas a microtejidos de dosis únicas y múltiples. La metodología ha sido validada con un amplio conjunto de compuestos graves y levemente hepatotóxicos. Aquí mostramos los datos de pioglitazona y troglitazona, conocidos compuestos hepatotóxicos retirados del mercado por causar fallas hepáticas. En general, se ha demostrado que el modelo de MPS hepático puede ser una herramienta útil para evaluar el DILI y su asociación con cambios en la función hepática. El modelo también se puede utilizar para evaluar cómo se comportan los nuevos compuestos en distintos subconjuntos de pacientes y cómo los perfiles de toxicidad pueden verse afectados por los estados de enfermedad hepática (por ejemplo, hepatitis viral, enfermedad del hígado graso).
Introduction
DILI sigue siendo la causa más común de insuficiencia hepática aguda en los EE.UU. y Europa y es una de las principales causas de desgaste de compuestos en el proceso de desarrollo de fármacos1. Casi todas las clases de medicamentos pueden causar hepatotoxicidad, siendo los agentes del sistema nervioso central y los antibióticos los tratamientos más comunes que causan DILI en pacientes2. La hepatotoxicidad inducida por fármacos es causada por una compleja interacción de factores genéticos, no genéticos y ambientales, que conduce a la muerte de los hepatocitos y otros tipos de células hepáticas, incluidos los colangiocitos y las células endoteliales 1,3.
Los agentes causantes de DILI se pueden clasificar de dos maneras: aquellos que causan daño hepático predecible dependiente de la dosis o aquellos que causan DILI idiosincrásico que es raro y se desarrolla independientemente de la dosis del fármaco, o la vía, o la duración de la administración, pero es responsable de hasta una sexta parte de todas las fallas hepáticas agudas en los EE.UU. sólo4. Desafortunadamente, DILI a menudo no se detecta hasta que los medicamentos han alcanzado las etapas clínicas del proceso de desarrollo del fármaco. El rango de lesión hepática inducida por fármacos (o DILIrank) consiste en más de mil medicamentos aprobados por la FDA que se dividen en cuatro clases de acuerdo con su potencial para causar DILI, y su uso en pacientes debe ser monitoreado de cerca5.
El estudio de los mecanismos de hepatotoxicidad del fármaco sigue siendo muy difícil y, por lo tanto, se han desarrollado muchos modelos preclínicos para explorar los mecanismos del DILI. Los modelos actuales in vitro e in vivo utilizados para predecir DILI en el desarrollo preclínico tienen varias limitaciones para proporcionar información sobre las interacciones complejas y multifacéticas en un cuerpo humano vivo. Las líneas celulares hepáticas cancerosas (es decir, HepG2, HepaRG) cultivadas en 2D todavía se utilizan en las primeras etapas del desarrollo de fármacos para evaluar la toxicidad de los compuestos candidatos6. Aun así, estas líneas celulares provienen de donantes individuales y muestran niveles anormales de función hepática, y no siempre presentan una alta sensibilidad para la detección de DILI 7,8. Como alternativa a las líneas celulares hepáticas cancerosas, los PHH representan mejor la fisiología del hígado humano si se cultivan adecuadamente in vitro, aunque existen varias limitaciones con su cultivo, como el corto tiempo de incubación con medicamentos, la vida relativamente corta, la pérdida de la expresión génica hepática y los cambios en las funciones metabólicas de los fármacos 9,10,11 . Los PHH se pueden cultivar en proteínas de la matriz extracelular en placas de cultivo celular 2D estándar, pero como inconveniente, la rápida disminución de su función significa que tienen baja sensibilidad (<50%) para la predicción DILI12.
Por otro lado, las pruebas en modelos animales son lentas, costosas y necesitan una traducción entre especies para extrapolar la predicción a los humanos. La mayoría de los medicamentos recientemente desarrollados no logran obtener aprobación, lo que hace que este proceso sea costoso y arriesgado5. Además, para probar nuevas modalidades específicas para humanos, los modelos animales son menos adecuados debido a la secuencia de genes o las diferencias de respuesta inmunológica en comparación con los humanos13.
En consecuencia, el interés en modelos hepáticos in vitro tridimensionales (3D) más avanzados ha crecido exponencialmente. El cultivo de PHH como estructuras esferoidales generadas por agregación gravitacional en gotas colgantes o en superficies de fijación ultra bajas representa un método de alto rendimiento para evaluar pasivos compuestos14. Los esferoides de PHH se han utilizado para evaluar DILI en un fondo enfermo (por ejemplo, esteatosis y colestasis)15. Se ha desarrollado una amplia variedad de modelos para incluir cocultivos micropatronados en placas de hepatocitos con fibroblastos estromales16, tejidos hepáticos bioimpresos en 3D 17, cultivos esferoides 3D con o sin células hepáticas no parenquimatosas15. Sin embargo, todos estos métodos todavía tienen inconvenientes, y el cultivo de PHH en un microambiente fisiológicamente más relevante podría proporcionarles niveles más altos de funcionalidad durante largos períodos de tiempo para permitir la investigación de la exposición prolongada a posibles hepatotóxicos. Además, para mejorar la relevancia traslacional de cualquier cultivo avanzado de PHH in vitro, se deben utilizar criterios de valoración funcionales clínicamente relevantes o biomarcadores de salida de toxicidad para permitir que los datos se comparen in vivo o en escenarios clínicos18.
En este estudio, evaluamos si un MPS, también conocido como modelo hepático in vitro de Organ-on-a-Chip (OOC), podría usarse para comprender los aspectos mecanicistas detallados de la toxicidad hepática. Se ha demostrado previamente que el MPS mantiene microtejidos hepáticos 3D altamente funcionales, bajo flujo, hasta por 4 semanas19. El sistema ha sido probado recientemente por la FDA y ha demostrado tener una alta reproducibilidad al realizar toxicidad de medicamentos, metabolismo y acumulación intracelular20. Además, en comparación con los esferoides y los cultivos sándwich, el sistema tuvo una función más estable y una mayor sensibilidad en la detección de la toxicidad de varios fármacos20. Hasta la fecha, el MPS se ha utilizado en una amplia gama de aplicaciones que cubren ADME21, modelado de enfermedades (VHB22, NAFLD 23,24,25) e interacciones farmacológicas 26, lo que lo hace potencialmente muy adecuado para evaluar el DILI agudo y crónico. La tecnología aquí presentada ofrece una alternativa para cerrar la brecha entre los cultivos celulares más tradicionales y los modelos animales y los ensayos clínicos en humanos, avanzando hacia la simulación de las condiciones biológicas humanas para apoyar la evaluación de la toxicidad hepática de los compuestos candidatos en las etapas preclínicas del proceso de desarrollo de fármacos.
Protocol
Representative Results
Discussion
Los MPS están diseñados para recapitular unidades funcionales de órganos humanos in vitro y han sido desarrollados para abordar las limitaciones de los modelos convencionales de cultivo celular 3D27. El hígado es uno de los órganos más modelados que utilizan MPS, y se ha desarrollado una amplia variedad de sistemas. El hígado humano es responsable del metabolismo de los fármacos y de la generación de metabolitos tóxicos de los fármacos, y su función es un elemento clave para modelar el desarrollo de fármacos, incluyendo la evaluación de la responsabilidad DILI de los compuestos28. Aquí hemos introducido un nuevo método para evaluar DILI utilizando un MPS hepático; el protocolo permite buscar conocimientos mecanicistas para cada compuesto ensayado para determinar cómo puede causar DILI, además de ser un ensayo altamente sensible y robusto. Los microtejidos hepáticos se forman en las placas MPS, que son un cocultivo de PHH y HKCs y son altamente funcionales con altos niveles de producción de albúmina y urea, así como alta actividad de CYP3A4 en comparación con los modelos hepáticos in vitro estándar20.
Aunque el modelo DILI descrito aquí puede servir como una herramienta útil en etapas posteriores de las pruebas preclínicas en el proceso de desarrollo de fármacos, también tiene varias limitaciones. Como la mayoría de MPS actualmente disponibles en el mercado, es una plataforma de bajo rendimiento y, por lo tanto, más difícil de usar para actividades de detección de drogas a gran escala. Al consistir en microtejidos formados por el cocultivo de PHH y HKC, el modelo DILI tampoco puede capturar completamente la complejidad del hígado humano, y una mayor optimización mediante la incorporación de diferentes tipos de células (por ejemplo, células inmunes) sería beneficiosa para agregar valor al modelo existente. Este MPS de un solo órgano también podría combinarse con otras plataformas de órganos que pueden compartir un medio común y permitir la diafonía de órganos a nivel celular o endocrino, y que pueden ayudar a comprender mejor los conocimientos mecanicistas de la toxicidad que no se limitan solo al hígado mismo. Además, como cualquier tecnología relativamente nueva, podría considerarse costosa y, por lo tanto, de accesibilidad limitada.
MPS es una plataforma utilizada para desarrollar modelos organotípicos de tejidos humanos individuales o múltiples. El sistema está compuesto por un controlador, un cable umbilical y un controlador MPS en el que se inserta la placa (Figura 5A). Cada placa MPS hepática tiene 12 pocillos abiertos independientes para cultivar células hepáticas primarias en 3D en andamios diseñados. En resumen, el sistema se verifica con control de calidad, y las placas se impriman en el día -1, los PHH y HKC se siembran en las placas en el día cero (Figura 5B, ver 1). Las microbombas integradas facilitan la circulación de los medios de cultivo celular a través de los andamios para facilitar la formación de microtejidos 3D (Figura 5B, ver 2). Los microtejidos formados se QC en el día 4, se dosifican con diferentes concentraciones de cada compuesto cada 48 h durante 4 días y se analizan para biomarcadores de punto final en el día 8 (Figura 5C). La línea de tiempo experimental del ensayo DILI en la placa MPS se representa en la Figura 5D.
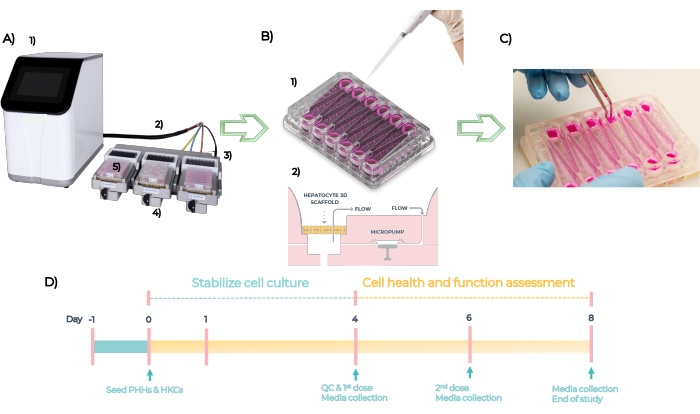
Figura 5: El sistema microfisiológico y la línea de tiempo experimental de un ensayo DILI estándar. (A) El sistema microfisiológico con sus componentes: controlador (1), cable umbilical (2), estación de acoplamiento (3), controlador MPS (4) y placa LC12 (5). (B) La siembra de PHH y HKC en la placa LC12 en el Día 1 (1) y las microbombas integradas facilitan la circulación de medios de cultivo celular con caudales sintonizables a través de los microtejidos 3D sembrados en los andamios (2). (C) Derribar los andamios al final del estudio. (D) Cronología experimental. Haga clic aquí para ver una versión más grande de esta figura.
Al realizar el protocolo, es importante que se realice una verificación de control de calidad robusta del sistema antes de comenzar, verificando que el sistema funcione neumáticamente correctamente y que las placas consumibles se inspeccionen visualmente y se imprimen de manera eficiente para garantizar una funcionalidad uniforme en todos los pozos. Tener células humanas primarias de alta calidad es esencial para este protocolo, ya que se sabe que los hepatocitos se adhieren constantemente en experimentos de cultivo celular y forman interacciones 3D. La descongelación de estas células también es un paso crítico, ya que los hepatocitos primarios no deben resuspenderse mediante la acción del pipeteo, ya que esto puede conducir rápidamente a la muerte celular. Tener una viabilidad celular superior al 85% es fundamental para una siembra exitosa, ya que grandes cantidades de desechos celulares interferirán con la formación de microtejidos 3D. El control de calidad de los microtejidos hepáticos formados en el día 4 también es importante, y el usuario debe asegurarse de que se midan los niveles aceptables de LDH y urea, ya que los parámetros fuera de rango pueden ser indicativos de la formación de tejido de mala calidad y permitir una resolución directa de problemas. Finalmente, la hidrocortisona utilizada en los medios de cultivo celular debe prepararse fresca el día de su uso para evitar cualquier degradación no deseada que pueda afectar la funcionalidad del cultivo celular, ya que es necesaria para mantener la funcionalidad metabólica de los hepatocitos.
A pesar de tener una complejidad significativa, el MPS hepático no contiene todos los tipos de células del hígado humano. Es posible agregar más tipos de células al modelo24,29 para aumentar la relevancia fisiológica, pero estas solo deben agregarse con una justificación clara para el contexto de uso. Para el estudio de DILI PHH son el tipo de célula clave, y la incorporación de HKCs en este modelo permite determinar algunas respuestas inmunológicas. También debe tenerse en cuenta que los PHH aislados de hígados humanos y los PHH criopreservados disponibles comercialmente tienden a demostrar algunas variaciones de un lote a otro. Hemos demostrado aquí que este protocolo produce resultados reproducibles cuando se utiliza con preparaciones de células de alta calidad. Sin embargo, se esperaría alguna variación de lote-lote, y esto podría superarse aún más mediante el uso de lotes agrupados de donantes múltiples. Estas limitaciones podrían ser superadas mediante el uso de células similares a los hepatocitos diferenciadas de iPSC que recapitulan muchas propiedades funcionales de los PHH y que han sido utilizadas en el proceso de desarrollo de fármacos30. Los HKC también muestran mucha variabilidad y un alto nivel de activación al descongelarse; por lo tanto, los donantes de HKC se validan previamente internamente antes de su uso en cultivo celular experimental (cocultivo con PHH validados) y deben tener bajos niveles de activación posterior al deshielo; esto se evalúa midiendo los biomarcadores IL-6 y TNF-alfa (ver Material Suplementario).
Los datos presentados aquí confirman que el ensayo puede detectar DILI con precisión, ayudando a identificar hepatotóxicos que podrían no ser detectados por 2D10,11 e incluso algunos modelos 3D. Los datos generados a partir de MPS todavía no son utilizados como estándar por la industria farmacéutica para presentaciones regulatorias o fines de detección de medicamentos debido a la falta de estandarización y armonización de procesos, incluida la reproducibilidad entre sitios20. Los datos y los enfoques experimentales demostrados aquí abordan esto, mostrando que el modelo hepático se puede usar de forma rutinaria y robusta en las pantallas DILI para predecir con precisión la responsabilidad de los nuevos compuestos.
Al medir una serie de criterios de valoración para producir una “firma de hepatotoxicidad”, ayudando a identificar compuestos con diferentes niveles de preocupación DILI (incluidos compuestos no detectables por otros métodos in vitro) y sus mecanismos de toxicidad revelados. Esta tecnología puede cerrar la brecha entre el cultivo celular tradicional y los modelos animales por un lado y los ensayos clínicos en humanos, avanzando hacia la simulación de las condiciones biológicas humanas para la evaluación preclínica de la toxicidad hepática como parte del proceso de desarrollo de fármacos.
Disclosures
The authors have nothing to disclose.
Acknowledgements
CN Bio Innovations Ltd. financió este estudio.
Materials
| 24 well cell culture cluster plates flat bottom | Corning | 3524 | |
| 96 well clear assay plates, flat bottom clear plastic | Greiner | 655101 | |
| 96 well plates black flat bottom | Corning | 3915 | |
| 96 Well White/Clear Bottom Plate, TC Surface | ThermoScientific | 165306 | |
| Advanced DMEM (1x) | Gibco | 12491015 | Cell culture media. |
| AssayMax Albumin ELISA Kit | AssayPro | EA3201-1 | Dilution 1:250. Time point Day 4, 6, and 8. |
| Cell Maintenance Cocktail B, (Primary Hepatocyte Maintenance Supplements) | Gibco | CM4000 | |
| CellTiter-Glo 3D Cell Viability Assay | Promega | G9682 | Dilution 1:1. Time point Day 8. |
| Chlorpromazine HCl | Sigma Aldrich | C8138 | |
| Chromacol blue lids, 9 mm Autosampler Vial Screw Thread Caps | ThermoScientific | 9-SCK(B)-ST1 | glass vial |
| Chromacol vials, 9 mm Clear Glass Screw Thread Vials | ThermoScientific | 2-SVW | |
| Class 2 Microbiological Safety Cabinets – Trimat2 1500 exhaust | Contained Air Solutions | ||
| Conical tubes 50 mL | Greiner | 227261 | |
| Cryopreserved Hepatocyte Recovery Medium (CHRM) | ThermoFisher Scientific | Gibco CM7000 | |
| Cryopreserved primary human hepatocytes | BioIVT Europe | Lot. RAS | |
| CytoTox 96 Cytotoxicty (LDH) Assay Kit | Promega | G1781 | Dilution – none. Time point Day 4, 6 and 8 |
| >Data analysis model used to generate the graph and EC:50 curves was nonlinear regression (curve fit) asymmetric sigmoidal, 5PL, where X is log(concentration | GraphPad Prism 9 | ||
| Disposable PES Filter Units 500mL | Fisher Scientific | 15913307 | |
| Disposable Pipette Basins 50ml | Fisher Scientific | 12369175 | |
| DMSO (Dimethyl sulfoxide) | Sigma-Aldrich | Sigma D2650 | |
| Dulbeco’s Phosphate Buffered Saline without Ca2+ and Mg2+ (D-PBS) | ThermoFisher Scientific | 14190-144 | |
| Easy Reader Conical Polypropylene Centrifuge Tubes 15 mL | Fisher Scientific | 11889640 | |
| Foetal bovine serum | Gibco | 10500064 | |
| Human ALT ELISA Kit | Abcam | ab 234578 | Dilution 1:5. Time point Day 6 and 8. |
| Human Cryopreserved Kupffer Cells | Lonza Europe | Lot. 190088KC | |
| hydrocortisone | Merck | H0888-1G | |
| Incubators models: New Brunswick Galaxy 170 S, New Brunswick Galaxy 170 R and CellXpert® C170. | Eppendorf | All serviced yearly; paperwork available upon request. | |
| Inverted Microscope | Leica DMIL LED | ||
| MPS know as Organ-on-a-Chip (OOC) | CN Bio Innovations Ltd. | ||
| MPS LC-12 plate | CN Bio Innovations Ltd. | ||
| Neubauer Improved C-Chip Disposable Haemocytometer (2 channel) | Cambridge Bioscience | DHC-N01-50 | |
| P450-Glo CYP3A4 Assay and Screening System | Promega | V9002 | Dilution – none. Time point Day 8 |
| PhysioMimix MPS platform | CN Bio Innovations Ltd. | ||
| Pioglitazone | MedChemExpress Tocris | HY-13956/CS-1700 | |
| Quantichrom Urea Assay Kit – Bioassay systems | Bioassay Systems | DY970-05 | Dilution 1:2 if initial reading is too high. Time point Day 4, 6 and 8. |
| Silica gel | Sigma-Aldrich | S7625 | |
| Software used to analyse and generate all the graphs was | GraphPad Prism 9 | ||
| Stripettes 10 mL | Fisher Scientific | 11839660 | |
| Stripettes 25 mL | Fisher Scientific | 11839181 | |
| Thawing plate Cocktail A, (Primary Hepatocyte Thawing and Plating Supplements) | Gibco | CM3000 | |
| Troglitazone | MedChemExpress Tocris | 97322-87-7 | |
| Trypan Blue Solution, 0.4% | Gibco | 15250061 | |
| Tubes 1.5 mL | Greiner | 616201 | |
| Weighing balance – model PA214C and AV213C | Ohaus Corp |
References
- Lisi, D. M. Drug-induced liver injury: An overview. US Pharmacist. 41 (12), 30-34 (2016).
- Kuna, L., et al. Models of drug induced liver injury (DILI)-current issues and future perspectives. Current Drug Metabolism. 19 (10), 830-838 (2018).
- Katarey, D., Verma, S. Drug-induced liver injury. Clinical Medicine. 16 (6), 104-109 (2016).
- Kullak-Ublick, G. A., et al. Drug-induced liver injury: recent advances in diagnosis and risk assessment Recent advances in clinical practice. Gut. 66, 1154-1164 (2017).
- Dirven, H., et al. Performance of pre-clinical models in predicting drug-induced liver injury in humans: a systematic review. Scientific Reports. 11 (1), 6403 (2021).
- Donato, M. T., Lahoz, A., Castell, J. V., Gomez-Lechon, M. J. Cell lines: a tool for in vitro drug metabolism studies. Current Drug Metabolism. 9 (1), 1-11 (2008).
- Wilkening, S., Stahl, F., Bader, A. Comparison of primary human hepatocytes and hepatoma cell line HepG2 with regard to their biotransformation properties. Drug Metabolism and Disposition. 31 (8), 1035-1042 (2003).
- Gerets, H. H. J., et al. Characterization of primary human hepatocytes, HepG2 cells, and HepaRG cells at the mRNA level and CYP activity in response to inducers and their predictivity for the detection of human hepatotoxins. Cell Biology and Toxicology. 28 (2), 69-87 (2012).
- Grainger, C. I., Greenwell, L. L., Lockley, D. J., Martin, G. P., Forbes, B. Culture of Calu-3 cells at the air interface provides a representative model of the airway epithelial barrier. Pharmaceutical Research. 23 (7), 1482-1490 (2006).
- Li, F., Cao, L., Parikh, S., Zuo, R. Three-dimensional spheroids with primary human liver cells and differential roles of kupffer cells in drug-induced liver injury. Journal of Pharmaceutical Sciences. 109 (6), 1912-1923 (2020).
- Proctor, W. R., et al. Utility of spherical human liver microtissues for prediction of clinical drug-induced liver injury. Archives of Toxicology. 91 (8), 2849-2863 (2017).
- Lin, C., Khetani, S. R. Advances in engineered liver models for investigating drug-induced liver injury. BioMed Research International. 2016, 1829148 (2016).
- Olson, H., et al. Concordance of the toxicity of pharmaceuticals in humans and in animals. Regulatory Toxicology and Pharmacology. 32 (1), 56-67 (2000).
- Bell, C. C., et al. Comparison of hepatic 2D sandwich cultures and 3D spheroids for long-term toxicity applications: A multicenter study. Toxicological Sciences. 162 (2), 655-666 (2018).
- Bell, C. C., et al. Characterization of primary human hepatocyte spheroids as a model system for drug-induced liver injury, liver function and disease. Scientific Reports. 6, 25187 (2016).
- Khetani, S. R., et al. Use of micropatterned co-cultures to detect compounds that cause drug-induced liver injury in humans. Toxicological Sciences. 132 (1), 107-117 (2013).
- Ma, X., et al. Deterministically patterned biomimetic human iPSC-derived hepatic model via rapid 3D bioprinting. Proceedings of the National Academy of Sciences of the united States of America. 113 (8), 2206-2211 (2016).
- Dieterle, P. Y. M., Dieterle, F. Tissue-specific, non-invasive toxicity biomarkers: translation from pre-clinical safety assessment to clinical safety monitoring. Expert Opinion on Drug Metabolism & Toxicology. 5 (9), 1023-1038 (2009).
- Rowe, C., et al. Perfused human hepatocyte microtissues identify reactive metabolite-forming and mitochondria-perturbing hepatotoxins. Toxicology in Vitro. 46, 29-38 (2018).
- Rubiano, A., et al. Characterizing the reproducibility in using a liver microphysiological system for assaying drug toxicity, metabolism, and accumulation. Clinical and Translational Science. 14 (3), 1049-1061 (2021).
- Tsamandouras, N., Kostrzewski, T., Stokes, C. L., Griffith, L. G., Hughes, D. J., Cirit, M. Quantitative assessment of population variability in hepatic drug metabolism using a perfused three-dimensional human liver microphysiological system. Journal of Pharmacology and Experimental Therapeutics. 360 (1), 95-105 (2017).
- Ortega-Prieto, A. M., et al. 3D microfluidic liver cultures as a physiological pre-clinical tool for hepatitis B virus infection. Nature Communications. 9 (1), 682 (2018).
- Kostrzewski, T., et al. Three-dimensional perfused human in vitro model of non-alcoholic fatty liver disease. World Journal of Gastroenterology. 23 (2), 204-215 (2017).
- Kostrzewski, T., et al. A microphysiological system for studying nonalcoholic steatohepatitis. Hepatology Communications. 4 (1), 77-91 (2020).
- Vacca, M., et al. Bone morphogenetic protein 8B promotes the progression of non-alcoholic steatohepatitis. Nature Metabolism. 2 (6), 514-531 (2020).
- Long, T. J., et al. Modeling therapeutic antibody-small molecule drug-drug interactions using a three-dimensional perfusable human liver co-culture platforms. Drug Metabolism and Disposition. 44, 1940-1948 (2016).
- Bai, J., Wang, C. Organoids and microphysiological systems: New tools for ophthalmic drug discovery. Frontiers in Pharmacology. 11, 407 (2020).
- Ribeiro, A. J. S., Yang, X., Patel, V., Madabushi, R., Strauss, D. G. Liver microphysiological systems for predicting and evaluating drug effects. Clinical Pharmacology & Therapeutics. 106 (1), 139-147 (2019).
- Clark, A. M., et al. A microphysiological system model of therapy for liver micrometastases hhs public access. Experimental Biology and Medicine (Maywood). 239 (9), 1170-1179 (2014).
- Qosa, H., Ribeiro, A. J. S., Hartman, N. R., Volpe, D. A. Characterization of a commercially available line of iPSC hepatocytes as models of hepatocyte function and toxicity for regulatory purposes. Journal of Pharmacological and Toxicological Methods. 110, 107083 (2021).

