Onderzoek naar biomoleculaire interactie tussen de moleculaire chaperonne Hsp90 en zijn clienteiwit kinase Cdc37 met behulp van field-effect biosensing-technologie
Summary
Field-effect biosensing (FEB) is een labelvrije techniek voor het detecteren van biomoleculaire interacties. Het meet de elektrische stroom door de grafeen biosensor waaraan de bindende doelen worden geïmmobiliseerd. De FEB-technologie werd gebruikt om biomoleculaire interacties tussen Hsp90 en Cdc37 te evalueren en er werd een sterke interactie tussen de twee eiwitten gedetecteerd.
Abstract
Biomoleculaire interacties spelen een veelzijdige rol in tal van cellulaire processen door functioneel relevante biologische gebeurtenissen te reguleren en te coördineren. Biomoleculen zoals eiwitten, koolhydraten, vitamines, vetzuren, nucleïnezuren en enzymen zijn fundamentele bouwstenen van levende wezens; ze verzamelen zich in complexe netwerken in biosystemen om een groot aantal levensgebeurtenissen te synchroniseren. Eiwitten maken meestal gebruik van complexe interactoomnetwerken om hun functies uit te voeren; daarom is het verplicht om dergelijke interacties te evalueren om hun belang in cellen op zowel cellulair als organismeniveau te ontrafelen. Om dit doel te bereiken, introduceren we een snel opkomende technologie, field-effect biosensing (FEB), om specifieke biomoleculaire interacties te bepalen. FEB is een benchtop, labelvrije en betrouwbare biomoleculaire detectietechniek om specifieke interacties te bepalen en maakt gebruik van hoogwaardige elektronische biosensoren. De FEB-technologie kan interacties in het nanomolaire bereik volgen vanwege de biocompatibele nanomaterialen die op het biosensoroppervlak worden gebruikt. Als proof of concept werd de eiwit-eiwitinteractie (PPI) tussen heat shock protein 90 (Hsp90) en celdelingscyclus 37 (Cdc37) opgehelderd. Hsp90 is een ATP-afhankelijke moleculaire chaperonne die een essentiële rol speelt bij het vouwen, stabiliteit, rijping en kwaliteitscontrole van veel eiwitten, waardoor meerdere vitale cellulaire functies worden gereguleerd. Cdc37 wordt beschouwd als een eiwitkinase-specifieke moleculaire chaperonne, omdat het specifiek eiwitkinasen voor Hsp90 herkent en rekruteert om hun stroomafwaartse signaaltransductieroutes te reguleren. Als zodanig wordt Cdc37 beschouwd als een co-chaperonne van Hsp90. De chaperonnekinaseroute (Hsp90/Cdc37-complex) is hypergeactiveerd bij meerdere maligniteiten die cellulaire groei bevorderen; daarom is het een potentieel doelwit voor kankertherapie. De huidige studie toont de efficiëntie van feb-technologie met behulp van het Hsp90/Cdc37-modelsysteem. FEB detecteerde een sterke PPI tussen de twee eiwitten (KD-waarden van 0,014 μM, 0,053 μM en 0,072 μM in drie onafhankelijke experimenten). Kortom, het VBO is een labelvrij en kosteneffectief PPI-detectieplatform, dat snelle en nauwkeurige metingen biedt.
Introduction
Biomoleculaire interacties:
Eiwitten zijn essentiële onderdelen van organismen en nemen deel aan tal van moleculaire routes zoals celmetabolisme, celstructuur, celsignalering, immuunresponsen, celadhesie en meer. Hoewel sommige eiwitten hun functie(s) onafhankelijk uitvoeren, interageren de meeste eiwitten met andere eiwitten met behulp van een bindende interface om de juiste biologische activiteit te coördineren1.
Biomoleculaire interacties kunnen voornamelijk worden geclassificeerd op basis van de verschillende structurele en functionele kenmerken van betrokken eiwitten2, bijvoorbeeld op basis van de eiwitoppervlakken, de complexe stabiliteit of de persistentie van interacties3. Het identificeren van essentiële eiwitten en hun rol in biomoleculaire interacties is van vitaal belang voor het begrijpen van biochemische mechanismen op moleculair niveau4. Momenteel zijn er verschillende benaderingen om deze interacties5 te detecteren: in vitro6, in silico7, in levende cellen8, ex vivo9 en in vivo10 met elk zijn eigen sterke en zwakke punten.
De in vivo assays worden uitgevoerd met behulp van het hele dier als een experimenteel hulpmiddel11, ende ex vivo assays worden uitgevoerd op weefselextracten of hele organen (bijv. Hart, hersenen, lever) in een gecontroleerde externe omgeving door minimale veranderingen in natuurlijke omstandigheden te bieden. De meest voorkomende toepassing van in vivo en ex vivo studies is het evalueren van de farmacokinetiek, farmacodynamiek en toxiciteitseffecten van potentiële farmacologische agentia vóór proeven bij mensen door hun algehele veiligheid en werkzaamheid te waarborgen12.
Biomoleculaire interacties kunnen ook worden gedetecteerd in levende cellen. Beeldvorming van levende cellen stelt ons in staat om dynamische interacties te observeren terwijl ze de reacties van een bepaalde biochemische route uitvoeren13. Bovendien kunnen detectietechnieken, zoals bioluminescentie of fluorescentieresonantie-energieoverdracht, informatie geven over waar en wanneer deze interacties plaatsvinden in de cel14. Hoewel detectie in levende cellen cruciale details biedt, vertrouwen deze detectiemethoden op optica en labels, die mogelijk niet de inheemse biologie weerspiegelen; ze zijn ook minder gecontroleerd dan in vitro methoden en vereisen gespecialiseerde expertise om15 uit te voeren.
De in silico computationele methoden worden voornamelijk gebruikt voor grootschalige screening van doelmoleculen vóór de in vitro experimenten. Computationele voorspellingsmethoden, computergebaseerde databases, moleculaire docking, kwantitatieve structuur-activiteitsrelaties en andere moleculaire dynamica-simulatiebenaderingen behoren tot de gevestigde in silico-tools 16. In vergelijking met moeizame experimentele technieken kunnen de in silico-tools gemakkelijk voorspellingen doen met een hoge gevoeligheid, maar met verminderde nauwkeurigheid in voorspellende prestaties17.
In vitro assays worden uitgevoerd met micro-organismen of biologische moleculen buiten hun standaard biologische context. Het weergeven van biomoleculaire interacties door middel van in vitro methoden is van cruciaal belang voor het begrijpen van eiwitfuncties en de biologie achter het complexe netwerk van celfuncties. De voorkeurstestmethodologie wordt gekozen op basis van de intrinsieke eigenschappen van het eiwit, kinetische waarden en de wijze en intensiteit van interacties18,19.
De interactie Hsp90/Cdc37:
De chaperonne-kinaseroute, die Hsp90 en Cdc37 verbindt, is een veelbelovend therapeutisch doelwit in de tumorbiologie20. Hsp90 speelt een centrale rol in de controle van de celcyclus, eiwitassemblage, celoverleving en signaalroutes. Eiwitten die afhankelijk zijn van Hsp90 voor hun functies worden geleverd aan Hsp90 voor complexatie via een co-chaperonne, zoals Cdc37. Het Hsp90/Cdc37-complex regelt de vouwing van de meeste eiwitkinasen en dient als hub voor een groot aantal intracellulaire signaleringsnetwerken21. Het is een veelbelovend antitumordoel vanwege de verhoogde expressie in verschillende maligniteiten, waaronder acute myeloblastische leukemie, multipel myeloom en hepatocellulair carcinoom22,23.
Veelgebruikte in vitro biomoleculaire interactie detectietechnieken
Co-immunoprecipitatie (co-IP) is een techniek die vertrouwt op antigeen-antilichaamspecificiteit om biologisch relevante interacties te identificeren24. Het belangrijkste nadeel van deze methode is het onvermogen om interacties met lage affiniteit en kinetische waarden te detecteren24. Biofysische methoden zoals isotherme titratiecalorimetrie (ITC), oppervlakteplasmonresonantie (SPR), biolaaginterferometrie (BLI) en FEB-technologie hebben de voorkeur om de kinetische waarden te bepalen.
ITC is een biofysische detectiemethode gebaseerd op de bepaling van bindingsenergie samen met een volledige thermodynamica-analyse om biomoleculaire interacties te karakteriseren25. Het belangrijkste voordeel van ITC is dat het geen etikettering of fixatie van het doeleiwit vereist. De belangrijkste problemen die ITC ondervindt, zijn de hoge concentratie doeleiwit die nodig is voor één experiment en de moeilijkheid om niet-covalente complexen te analyseren als gevolg van kleine bindende enthalpies26. Zowel SPR als BLI zijn labelvrije biofysische technieken die afhankelijk zijn van de immobilisatie van het doelmolecuul op het sensoroppervlak, gevolgd door daaropvolgende injecties van de analyt over het geïmmobiliseerde doel27,28. In SPR worden veranderingen in de brekingsindex tijdens biomoleculaire interacties gemeten27; in BLI wordt de interferentie in gereflecteerd licht in real-time geregistreerd als een verandering in golflengte als functie van tijd28. Zowel SPR als BLI delen gemeenschappelijke voordelen van het aanbieden van hoge specificiteit, gevoeligheid en detectiemogelijkheden29. In beide methoden wordt het doeleiwit geïmmobiliseerd op biosensoroppervlakken en daarom kan er enig verlies zijn van de oorspronkelijke conformatie van het doelwit, waardoor het moeilijk is om onderscheid te maken tussen specifieke versus niet-specifieke interacties30. BLI gebruikt dure wegwerpbare glasvezelbiosensoren om het doelwit te immobiliseren en is daarom een kostbare techniek31. In vergelijking met deze gevestigde biomoleculaire detectietools biedt FEB-technologie een betrouwbaar en labelvrij platform door lage nanomolelarconcentraties te gebruiken voor biomoleculaire detectie in realtime met kinetische karakterisering. De FEB-technologie overwint ook de borrelende uitdagingen in ITC en is kosteneffectiever in vergelijking met SPR of BLI.
De op veldeffecttransistor (FET) gebaseerde biosensoren is een opkomend veld voor het detecteren van biomoleculaire interacties door gevarieerde biomedische toepassingen aan te bieden. In het FET-systeem worden doelen geïmmobiliseerd naar de biosensorchips en interacties worden gedetecteerd door veranderingen in geleiding32. Het unieke kenmerk waarmee rekening moet worden gehouden bij de ontwikkeling van een efficiënte elektronische biosensor zijn de fysisch-chemische eigenschappen zoals de semi-geleidende aard en chemische stabiliteit van het coatingmateriaal dat wordt gebruikt om het sensoroppervlak te fabriceren33. Conventionele materialen zoals silicium die voor FET worden gebruikt, hebben de gevoeligheid van sensoren beperkt omdat het oxidelagen vereist die ingeklemd zitten tussen het transistorkanaal en een specifieke omgeving voor een goede werking34. Bovendien zijn siliciumtransistors gevoelig voor zoutrijke omgevingen, waardoor het moeilijk is om biologische interacties in hun natuurlijke omgeving te meten. De op grafeen gebaseerde biosensor wordt gepresenteerd als een alternatief omdat het een uitstekende chemische stabiliteit en elektrisch veld biedt. Aangezien grafeen een enkele atomaire laag koolstof is, is het zowel extreem gevoelig als een halfgeleider en chemisch compatibel met biologische oplossingen; beide kwaliteiten zijn wenselijk om compatibele elektronische biosensoren te genereren35. Het opmerkelijke ultrahoge laadpotentieel van biomoleculen dat wordt geboden door biosensoren met grafeencoating, leidt tot de ontwikkeling van op grafeen gebaseerde biosensoren FEB-technologie.
Principe van FEB-technologie: FEB is een labelvrije biomoleculaire detectietechniek die de elektrische stroom meet door de grafeenbiosensor waaraan de bindingsdoelen worden geïmmobiliseerd. Interacties tussen het geïmmobiliseerde eiwit en de analyt resulteren in veranderingen in de stroom die in realtime worden gemonitord, waardoor nauwkeurige kinetische metingenmogelijk zijn 36.
Instrumentatie: Het FEB-systeem bestaat uit een grafeenveldeffecttransistor (gFET) sensorchip en een elektronische lezer die gedurende het hele experiment een constante spanning toepast (figuur 1). De analyt wordt in oplossing aangebracht op het doeleiwit dat op het biosensoroppervlak is geïmmobiliseerd. Wanneer een interactie optreedt, wordt een verandering in de stroom gemeten en in realtime geregistreerd. Naarmate de analytconcentratie toeneemt, zal ook de fractie van gebonden analyt toenemen, waardoor hogere wisselingen in de stroom ontstaan. Met behulp van de geautomatiseerde analysesoftware die bij het instrument wordt geleverd (Tabel van materialen), wordt I-Respons gemeten en geregistreerd in termen van biosensing units (BU)37. I-Respons wordt gedefinieerd als de verandering in de stroom (I) door de biosensorchip gemeten in real-time bij de interactie van het geïmmobiliseerde doelwit met de analyt. De geautomatiseerde analysesoftware van het VBO kan zowel de I-Respons als de C-Respons op dynamische interactiegebeurtenissen analyseren, waarbij de C-Respons de veranderingen in de capaciteit (C) registreert. De variaties in zowel de I-Respons als de C-Respons komen direct overeen met de fractie van gebonden analyt en kunnen verder worden geanalyseerd om KD-waarden te genereren. De standaardvoorkeur van de geautomatiseerde analysesoftware is I-Response.
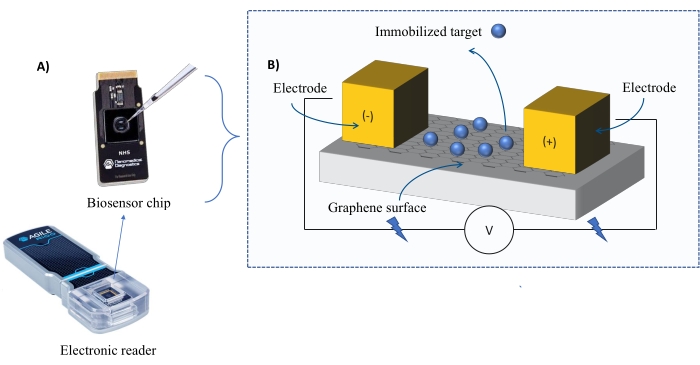
Figuur 1: Overzicht van de experimentele opstelling. (A) Op grafeen gebaseerde chip en een elektronische lezer. (B) Een overzicht van de chipcomponenten. De chip is bevestigd aan twee elektroden die stroom leveren aan het systeem. Het oppervlak van de chip is bedekt met grafeen, dat bij activering het doelwit kan binden. Klik hier om een grotere versie van deze figuur te bekijken.
Methodologie:
In eerste instantie wordt de geactiveerde biosensorchip in het FEB-apparaat ingebracht (figuur 1), gevolgd door de uitvoering van de onderstaande stappen: (1) Kalibratie: Het experiment begint met systeemkalibratie met behulp van 1x fosfaat-gebufferde zoutoplossing (PBS; pH = 7,4) om de basislijn equilibratierespons te creëren. (2) Associatie: De analyt wordt in de chip geïntroduceerd en de I-respons wordt gemonitord totdat de bindingsverzadiging is bereikt. (3) Dissociatie: De analyt wordt gedissocieerd met behulp van 1x PBS. (4) Regeneratie: Restanten van de analyt worden verwijderd met behulp van 1x PBS. (5) Wassen: In totaal worden vijf wasbeurten uitgevoerd met behulp van 1x PBS voor het grondig verwijderen van de gebonden en ongebonden analyten uit de chip.
Analyse:
Data-analyse wordt uitgevoerd met behulp van de volledig geautomatiseerde software die bij het instrument wordt geleverd. De geautomatiseerde analysesoftware genereert een Hill fit plot met eenKD-waarde . De Hill fit plot beschrijft de associatie van een analyt met het doeleiwit als functie van analytconcentraties. De concentratie waarbij een half-maximale respons wordt bereikt, is evenredig met de KD-waarde . Een lageKD-waarde vertegenwoordigt een hoge bindingsaffiniteit en vice versa.
Om de gegevens verkregen uit het VBO-experiment te valideren, worden I-responsen geëxtraheerd uit elk uitleespunt voor elke analytconcentratie met behulp van de data review/export software en kunnen ze worden geëxporteerd naar andere statistische analysesoftware (zie Tabel met materialen) zoals hieronder uitgelegd.
Protocol
Representative Results
Discussion
In deze studie werd de haalbaarheid van het gebruik van de FEB-technologie (een real-time kinetische karakteriseringsbenadering) geëvalueerd om de biomoleculaire interactie tussen Hsp90 en Cdc37 te bepalen. Het eerste verkennende experiment (eerste experiment) suggereerde dat het kiezen van de juiste analytconcentraties een cruciaal onderdeel van het experiment is en dat het experiment moet worden ontworpen door concentratiepunten boven en onder de KD-waarde op te nemen, die werden voorspeld op basis van de g…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Dit onderzoek werd ondersteund door een subsidie van de Binational Science Foundation (BSF) aan S.K.S. en N.Q.
Materials
| Automated analysis software | Agile plus software, Cardea (Nanomed) | NA CAS number: NA |
Referred to in the text as the automated analysis software supplied with the instrument. Generates automated analysis. |
| COOH-BPU (Biosensing Processing Unit) | Agile plus software, Cardea (Nanomed) | NA CAS number: NA |
biosensor chip |
| Data review software | Datalign 1.0, Cardea (Nanomed) | NA CAS number: NA |
Referred to as the supplied data review software in the text. Supplied with the instrument and allows to review and export the information data points. |
| Dialysis bag | CelluSep, Membrane filtration products | T2-10-15 CAS number: NA |
T2 tubings (6,000-8,000 MWCO), (10 mm fw, 6.4mm Ø, 0.32ml/cm, 15m) |
| EDC (1-Ethyl-3-(3-dimethylamino propyl) carbodiimide) | Cardea (Nanomed) | EDC160322-02 CAS number: 25952-53-8 |
White powder |
| ITC (Isothermal titration calorimetry) system | Microcal-PEAQ-ITC (Malvern, United Kingdom) | NA CAS number: NA |
|
| MES (2-(N-morpholino) ethane sulfonic acid) buffer | Merck | M3671-50G CAS number: 4432-31-9 |
White powder |
| NHS (N-Hydroxysulfosuccinimide) chips | Cardea (Nanomed) | NA CAS number: NA |
Graphene-based chip |
| PBS (Phosphate-buffered saline) X 10 | Bio-Lab | 001623237500 CAS number: 7758-11-4 |
Liquid transparent solution |
| Pipete | Thermo Scientific | 11855231 CAS number: NA |
Finnpipette F3 5-50 µL, yellow |
| Quench 1 (3.9 mM amino-PEG5-alcohol in 1 X PBS) | Cardea (Nanomed) | 0105-001-002-001 CAS number: NA |
Liquid, transparent solution |
| Quench 2 (1 M ethanolamine (pH=8.5)) | Cardea (Nanomed) | 0105-001-003-001 CAS number: NA |
Liquid, transparent solution |
| Recombinant protein Cdc37 | Abcam | ab256157 CAS number: NA |
|
| Recombinant protein Hsp90 beta | Abcam | ab80033 CAS number: NA |
|
| Spreadsheet | Excel, Microsoft office | NA CAS number: NA |
|
| Statistical software | GraphPad, Prism | NA CAS number: NA |
Referred to as the other statistical software. Sigma plot, phyton or other statistical programes may also be used |
| Sulfo-NHS | Cardea (Nanomed) | NHS160321-07 CAS number: 106627-54-7 |
White powder |
| Tips | Alex red | LC 1093-800-000 CAS number: NA |
Tip 1-200 µl, in bulk, 1,000 pcs |
References
- Tuncbag, N., Gursoy, A., Guney, E., Nussinov, R., Keskin, O. Architectures and functional coverage of protein-protein interfaces. Journal of Molecular Biology. 381 (3), 785-802 (2008).
- Berggård, T., Linse, S., James, P. Methods for the detection and analysis of protein–protein interactions. Proteomics. 7 (16), 2833-2842 (2007).
- Magliery, T. J., et al. Detecting protein-protein interactions with a green fluorescent protein fragment reassembly trap: Scope and mechanism. Journal of the American Chemical Society. 127 (1), 146-157 (2005).
- Xing, S., Wallmeroth, N., Berendzen, K. W., Grefen, C. Techniques for the analysis of protein-protein interactions in vivo. Plant Physiology. 171 (2), 727-758 (2016).
- Nguyen, T. N., Goodrich, J. A. Protein-protein interaction assays: Eliminating false positive interactions. Nature Methods. 3 (2), 135-139 (2006).
- Fernández-Suárez, M., Chen, T. S., Ting, A. Y. Protein-protein interaction detection in vitro and in cells by proximity biotinylation. Journal of the American Chemical Society. 130 (29), 9251-9253 (2008).
- Jiang, M., Niu, C., Cao, J., Ni, D. -. A., Chu, Z. In silico-prediction of protein–protein interactions network about MAPKs and PP2Cs reveals a novel docking site variants in Brachypodium distachyon. Scientific Reports. 8 (1), 15083 (2018).
- Yazawa, M., Sadaghiani, A. M., Hsueh, B., Dolmetsch, R. E. Induction of protein-protein interactions in live cells using light. Nature Biotechnology. 27 (10), 941-945 (2009).
- Wang, W., Goodman, M. T. Antioxidant property of dietary phenolic agents in a human LDL-oxidation ex vivo model: Interaction of protein binding activity. Nutrition Research. 19 (2), 191-202 (1999).
- Xing, S., Wallmeroth, N., Berendzen, K. W., Grefen, C. Techniques for the analysis of protein-protein interactions in vivo. Plant Physiology. 171 (2), 727-758 (2016).
- Qvit, N., Disatnik, M. -. H., Sho, E., Mochly-Rosen, D. Selective phosphorylation inhibitor of delta protein kinase C–Pyruvate dehydrogenase kinase protein–protein interactions: Application for myocardial injury in vivo. Journal of the American Chemical Society. 138 (24), 7626-7635 (2016).
- Alam, M. N., Bristi, N. J., Rafiquzzaman, M. Review on in vivo and in vitro methods evaluation of antioxidant activity. Saudi Pharmaceutical Journal. 21 (2), 143-152 (2013).
- Paulmurugan, R., Gambhir, S. S. Novel fusion protein approach for efficient high-throughput screening of small molecule–mediating protein-protein interactions in cells and living animals. Cancer Research. 65 (16), 7413-7420 (2005).
- Boute, N., Jockers, R., Issad, T. The use of resonance energy transfer in high-throughput screening: BRET versus FRET. Trends in Pharmacological Sciences. 23 (8), 351-354 (2002).
- Deriziotis, P., Graham, S. A., Estruch, S. B., Fisher, S. E. Investigating protein-protein interactions in live cells using bioluminescence resonance energy transfer. Journal of Visualized Experiments: JoVE. (87), e51438 (2014).
- Ekins, S., Mestres, J., Testa, B. In silico pharmacology for drug discovery: Methods for virtual ligand screening and profiling. British Journal of Pharmacology. 152 (1), 9-20 (2007).
- Valerio, L. G. Application of advanced in silico methods for predictive modeling and information integration. Expert Opinion on Drug Metabolism & Toxicology. 8 (4), 395-398 (2012).
- Piehler, J. New methodologies for measuring protein interactions in vivo and in vitro. Current Opinion in Structural Biology. 15 (1), 4-14 (2005).
- Ideker, T., Sharan, R. Protein networks in disease. Genome Research. 18 (4), 644-652 (2008).
- Lu, H., et al. Recent advances in the development of protein–protein interactions modulators: mechanisms and clinical trials. Signal Transduction and Targeted Therapy. 5 (1), 213 (2020).
- Jarosz, D. Hsp90: A global regulator of the genotype-to-phenotype map in cancers. Advances in Cancer Research. 129, 225-247 (2016).
- Johnson, V. A., Singh, E. K., Nazarova, L. A., Alexander, L. D., McAlpine, S. R. Macrocyclic inhibitors of Hsp90. Current Topics in Medicinal Chemistry. 10 (14), 1380-1402 (2010).
- Mahalingam, D., et al. Targeting HSP90 for cancer therapy. British Journal of Cancer. 100 (10), 1523-1529 (2009).
- Stewart, A., Fisher, R. A. Co-Immunoprecipitation: Isolation of protein signaling complexes from native tissues. Methods in Cell Biology. 112, 33-54 (2012).
- Pierce, M. M., Raman, C. S., Nall, B. T. Isothermal titration calorimetry of protein-protein interactions. Methods. 19 (2), 213-221 (1999).
- Paketurytė, V., et al. Inhibitor binding to carbonic anhydrases by isothermal titration calorimetry. Carbonic Anhydrase as Drug Target. , 79-95 (2019).
- Grote, J., Dankbar, N., Gedig, E., Koenig, S. Surface plasmon resonance/mass spectrometry interface. Analytical Chemistry. 77 (4), 1157-1162 (2005).
- Kumaraswamy, S., Tobias, R. Label-free kinetic analysis of an antibody–antigen interaction using biolayer interferometry. Methods in Molecular Biology. , 165-182 (2015).
- Wallner, J., Lhota, G., Jeschek, D., Mader, A., Vorauer-Uhl, K. Application of bio-layer interferometry for the analysis of protein/liposome interactions. Journal of Pharmaceutical and Biomedical Analysis. 72, 150-154 (2013).
- Singh, A. N., Ramadan, K., Singh, S. Experimental methods to study the kinetics of protein–protein interactions. Advances in Protein Molecular and Structural Biology Methods. , 115-124 (2022).
- Frenzel, D., Willbold, D. Kinetic titration series with biolayer interferometry. PLoS One. 9 (9), 106882 (2014).
- Vu, C. -. A., Chen, W. -. Y. Field-effect transistor biosensors for biomedical applications: Recent advances and future prospects. Sensors. 19 (19), 4214 (2019).
- Bergveld, P. A critical evaluation of direct electrical protein detection methods. Biosensors & Bioelectronics. 6 (1), 55-72 (1991).
- Lowe, B. M., Sun, K., Zeimpekis, I., Skylaris, C. K., Green, N. G. Field-effect sensors – from pH sensing to biosensing: sensitivity enhancement using streptavidin–biotin as a model system. The Analyst. 142 (22), 4173-4200 (2017).
- Goldsmith, B. R., et al. Digital biosensing by foundry-fabricated graphene sensors. Scientific Reports. 9 (1), 434 (2019).
- Afsahi, S., et al. Novel graphene-based biosensor for early detection of Zika virus infection. Biosensors and Bioelectronics. 100, 85-88 (2018).
- Afsahi, S. J., et al. Towards novel graphene-enabled diagnostic assays with improved signal-to-noise ratio. MRS Advances. 2 (60), 3733-3739 (2017).
- Roe, S. M., et al. The mechanism of Hsp90 regulation by the protein kinase-specific cochaperone p50cdc37. Cell. 116 (1), 87-98 (2004).
- Gaiser, A. M., Kretzschmar, A., Richter, K. Cdc37-Hsp90 complexes are responsive to nucleotide-induced conformational changes and binding of further cofactors. The Journal of Biological Chemistry. 285 (52), 40921-40932 (2010).
- Popescu, A. I., Găzdaru, D. M., Chilom, C. G., Bacalum, M. Biophysical interactions: Their paramount importance for life. Romanian Reports in Physics. 65 (3), 1063-1077 (2013).
- Surya, S., Abhilash, J., Geethanandan, K., Sadasivan, C., Haridas, M. A profile of protein-protein interaction: Crystal structure of a lectin-lectin complex. International Journal of Biological Macromolecules. 87, 529-536 (2016).
- Velazquez-Campoy, A., Freire, E. ITC in the post-genomic era…? Priceless. Biophysical Chemistry. 115 (23), 115-124 (2005).
- Concepcion, J., et al. Label-free detection of biomolecular interactions using bioLayer interferometry for kinetic characterization. Combinatorial Chemistry & High Throughput Screening. 12 (8), 791-800 (2009).
- Helmerhorst, E., Chandler, D. J., Nussio, M., Mamotte, C. D. Real-time and label-free bio-sensing of molecular interactions by surface plasmon resonance: A laboratory medicine perspective. The Clinical Biochemist. Reviews. 33 (4), 161-173 (2012).
- Jacob, N. T., et al. Synthetic molecules for disruption of the MYC protein-protein interface. Bioorganic & Medicinal Chemistry. 26 (14), 4234-4239 (2018).

