Esplorare l'interazione biomolecolare tra lo chaperone molecolare Hsp90 e la sua proteina chinasi cliente Cdc37 utilizzando la tecnologia di biorilevamento ad effetto di campo
Summary
Il biosensing ad effetto di campo (FEB) è una tecnica senza etichetta per rilevare le interazioni biomolecolari. Misura la corrente elettrica attraverso il biosensore di grafene a cui sono immobilizzati i bersagli leganti. La tecnologia FEB è stata utilizzata per valutare le interazioni biomolecolari tra Hsp90 e Cdc37 ed è stata rilevata una forte interazione tra le due proteine.
Abstract
Le interazioni biomolecolari svolgono ruoli versatili in numerosi processi cellulari regolando e coordinando eventi biologici funzionalmente rilevanti. Biomolecole come proteine, carboidrati, vitamine, acidi grassi, acidi nucleici ed enzimi sono elementi fondamentali degli esseri viventi; si assemblano in reti complesse nei biosistemi per sincronizzare una miriade di eventi della vita. Le proteine utilizzano tipicamente reti interatomiche complesse per svolgere le loro funzioni; quindi è obbligatorio valutare tali interazioni per svelare la loro importanza nelle cellule sia a livello cellulare che a livello di organismo. Verso questo obiettivo, introduciamo una tecnologia in rapida ascesa, il biosensing ad effetto di campo (FEB), per determinare specifiche interazioni biomolecolari. FEB è una tecnica di rilevamento biomolecolare da banco, senza etichette e affidabile per determinare interazioni specifiche e utilizza biosensori elettronici di alta qualità. La tecnologia FEB può monitorare le interazioni nell’intervallo nanomolare a causa dei nanomateriali biocompatibili utilizzati sulla superficie del biosensore. Come prova di concetto, è stata chiarita l’interazione proteina-proteina (PPI) tra la proteina da shock termico 90 (Hsp90) e il ciclo di divisione cellulare 37 (Cdc37). Hsp90 è un chaperone molecolare dipendente dall’ATP che svolge un ruolo essenziale nel ripiegamento, nella stabilità, nella maturazione e nel controllo di qualità di molte proteine, regolando così molteplici funzioni cellulari vitali. Cdc37 è considerato un chaperone molecolare specifico della proteina chinasi, in quanto riconosce e recluta specificamente le chinasi proteiche a Hsp90 per regolare le loro vie di trasduzione del segnale a valle. Come tale, Cdc37 è considerato un co-chaperone di Hsp90. La via chaperone-chinasi (complesso Hsp90/Cdc37) è iper-attivata in tumori maligni multipli che promuovono la crescita cellulare; pertanto, è un potenziale bersaglio per la terapia del cancro. Il presente studio dimostra l’efficienza della tecnologia FEB utilizzando il sistema modello Hsp90/Cdc37. FEB ha rilevato un forte PPI tra le due proteine (valori KD di 0,014 μM, 0,053 μM e 0,072 μM in tre esperimenti indipendenti). In sintesi, FEB è una piattaforma di rilevamento PPI senza etichette ed economica, che offre misurazioni rapide e accurate.
Introduction
Interazioni biomolecolari:
Le proteine sono parti essenziali degli organismi e partecipano a numerosi percorsi molecolari come il metabolismo cellulare, la struttura cellulare, la segnalazione cellulare, le risposte immunitarie, l’adesione cellulare e altro ancora. Mentre alcune proteine svolgono le loro funzioni in modo indipendente, la maggior parte delle proteine interagisce con altre proteine utilizzando un’interfaccia di legame per coordinare la corretta attività biologica1.
Le interazioni biomolecolari possono essere classificate principalmente in base alle distinte caratteristiche strutturali e funzionali delle proteine coinvolte2, ad esempio in base alle superfici proteiche, alla stabilità complessa o alla persistenza delle interazioni3. Identificare le proteine essenziali e il loro ruolo nelle interazioni biomolecolari è vitale per comprendere i meccanismi biochimici a livello molecolare4. Attualmente, ci sono vari approcci per rilevare queste interazioni5: in vitro6, in silico7, in cellule vive8, ex vivo9 e in vivo10 con ciascuno con i propri punti di forza e di debolezza.
I saggi in vivo vengono eseguiti utilizzando l’intero animale come strumento sperimentale11 e i saggiex vivo vengono eseguiti su estratti di tessuto o organi interi (ad esempio, cuore, cervello, fegato) in un ambiente esterno controllato fornendo alterazioni minime in condizioni naturali. L’applicazione più comune degli studi in vivo ed ex vivo è quella di valutare gli effetti di farmacocinetica, farmacodinamica e tossicità dei potenziali agenti farmacologici prima degli studi sull’uomo, garantendone la sicurezza e l’efficacia complessive12.
Le interazioni biomolecolari possono anche essere rilevate all’interno delle cellule viventi. L’imaging di cellule vive ci consente di osservare le interazioni dinamiche mentre eseguono le reazioni di un particolare percorso biochimico13. Inoltre, le tecniche di rilevamento, come la bioluminescenza o il trasferimento di energia di risonanza di fluorescenza, possono fornire informazioni su dove e quando queste interazioni si verificano all’interno della cellula14. Sebbene il rilevamento in cellule vive offra dettagli cruciali, queste metodologie di rilevamento si basano su ottiche ed etichette, che potrebbero non riflettere la biologia nativa; sono anche meno controllati rispetto ai metodi in vitro e richiedono competenze specializzate per eseguire15.
I metodi computazionali in silico sono utilizzati principalmente per lo screening su larga scala delle molecole bersaglio prima degli esperimenti in vitro . Metodi di previsione computazionale, database basati su computer, docking molecolare, relazioni quantitative struttura-attività e altri approcci di simulazione della dinamica molecolare sono tra gli strumenti consolidati in silico 16. Rispetto alle laboriose tecniche sperimentali, gli strumenti in silico possono facilmente fare previsioni con alta sensibilità, ma con precisione ridotta nelle prestazioni predittive17.
I saggi in vitro vengono eseguiti con microrganismi o molecole biologiche al di fuori del loro contesto biologico standard. Ritrarre le interazioni biomolecolari attraverso metodi in vitro è fondamentale per comprendere le funzioni proteiche e la biologia alla base della complessa rete di funzionamento cellulare. La metodologia di analisi preferita viene scelta in base alle proprietà intrinseche della proteina, ai valori cinetici e alla modalità e intensità delle interazioni18,19.
L’interazione Hsp90/Cdc37:
La via chaperone-chinasi, che collega Hsp90 e Cdc37, è un obiettivo terapeutico promettente nella biologia tumorale20. Hsp90 svolge un ruolo centrale nel controllo del ciclo cellulare, nell’assemblaggio delle proteine, nella sopravvivenza cellulare e nelle vie di segnalazione. Le proteine che si basano su Hsp90 per le loro funzioni vengono consegnate a Hsp90 per la complessazione attraverso un co-chaperone, come Cdc37. Il complesso Hsp90/Cdc37 controlla il ripiegamento della maggior parte delle protein chinasi e funge da hub per una moltitudine di reti di segnalazione intracellulare21. È un promettente bersaglio antitumorale grazie alla sua elevata espressione in varie neoplasie maligne, tra cui la leucemia mieloblastica acuta, il mieloma multiplo e il carcinoma epatocellulare22,23.
Tecniche di rilevamento dell’interazione biomolecolare in vitro comunemente usate
La co-immunoprecipitazione (co-IP) è una tecnica che si basa sulla specificità antigene-anticorpo per identificare interazioni biologicamente rilevanti24. Lo svantaggio principale di questo metodo è la sua incapacità di rilevare interazioni a bassa affinità e valori cinetici24. I metodi biofisici come la calorimetria a titolazione isotermica (ITC), la risonanza plasmonica di superficie (SPR), l’interferometria a biostrato (BLI) e la tecnologia FEB sono preferiti per determinare i valori cinetici.
ITC è un metodo di rilevazione biofisica basato sulla determinazione dell’energia di legame insieme a un’analisi termodinamica completa per caratterizzare le interazioni biomolecolari25. Il vantaggio principale dell’ITC è che non richiede alcuna etichettatura o fissazione della proteina bersaglio. Le principali difficoltà incontrate dall’ITC sono l’alta concentrazione di proteine bersaglio richieste per un esperimento e la difficoltà di analizzare complessi non covalenti a causa di piccole entalpie di legame26. Sia SPR che BLI sono tecniche biofisiche label-free che si basano sull’immobilizzazione della molecola bersaglio sulla superficie del sensore, seguita da successive iniezioni dell’analita sul bersaglio immobilizzato27,28. In SPR, le alterazioni dell’indice di rifrazione durante le interazioni biomolecolari sono misurate27; in BLI, l’interferenza nella luce riflessa viene registrata in tempo reale come variazione della lunghezza d’onda in funzione del tempo28. Sia SPR che BLI condividono i vantaggi comuni di offrire elevate funzionalità di specificità, sensibilità e rilevamento29. In entrambi i metodi, la proteina bersaglio è immobilizzata sulle superfici del biosensore e, quindi, potrebbe esserci una certa perdita della conformazione nativa del bersaglio, il che rende difficile discriminare tra interazioni specifiche e non specifiche30. BLI utilizza costosi biosensori monouso in fibra ottica per immobilizzare il bersaglio ed è, quindi, una tecnica costosa31. Rispetto a questi strumenti di rilevamento biomolecolari consolidati, la tecnologia FEB offre una piattaforma affidabile e priva di etichette utilizzando basse concentrazioni nanomolari per il rilevamento biomolecolare in tempo reale con caratterizzazione cinetica. La tecnologia FEB supera anche le sfide spumeggianti affrontate in ITC ed è più conveniente rispetto a SPR o BLI.
I biosensori basati su transistor ad effetto di campo (FET) sono un campo emergente per rilevare interazioni biomolecolari offrendo varie applicazioni biomediche. Nel sistema FET, i bersagli sono immobilizzati ai chip del biosensore e le interazioni vengono rilevate dai cambiamenti nella conduttanza32. La caratteristica unica da considerare nello sviluppo di un biosensore elettronico efficiente sono le proprietà fisico-chimiche come la natura semi-conduttiva e la stabilità chimica del materiale di rivestimento utilizzato per fabbricare la superficie del sensore33. I materiali convenzionali come il silicio utilizzato per il FET hanno limitato la sensibilità dei sensori perché richiede strati di ossido inseriti tra il canale del transistor e un ambiente specifico per il corretto funzionamento34. Inoltre, i transistor al silicio sono sensibili agli ambienti ad alto contenuto di sale, rendendo così difficile misurare le interazioni biologiche nel loro ambiente naturale. Il biosensore a base di grafene si presenta come un’alternativa in quanto offre un’eccellente stabilità chimica e campo elettrico. Poiché il grafene è un singolo strato atomico di carbonio, è sia estremamente sensibile come semiconduttore che chimicamente compatibile con soluzioni biologiche; entrambe queste qualità sono desiderabili per generare biosensori elettronici compatibili35. Il notevole potenziale di carico ultra elevato delle biomolecole offerto dai biosensori rivestiti di grafene ha portato allo sviluppo della tecnologia FEB dei biosensori a base di grafene.
Principio della tecnologia FEB: FEB è una tecnica di rilevamento biomolecolare senza etichetta che misura la corrente elettrica attraverso il biosensore di grafene a cui sono immobilizzati i bersagli di legame. Le interazioni tra la proteina immobilizzata e l’analita provocano alterazioni della corrente che vengono monitorate in tempo reale, consentendo misurazioni cinetiche accurate36.
Strumentazione: Il sistema FEB comprende un chip sensore a transistor a effetto di campo del grafene (gFET) e un lettore elettronico che applica una tensione costante durante l’esperimento (Figura 1). L’analita viene applicato in soluzione alla proteina bersaglio immobilizzata sulla superficie del biosensore. Quando si verifica un’interazione, un’alterazione della corrente viene misurata e registrata in tempo reale. All’aumentare della concentrazione dell’analita, aumenterà anche la frazione di analita legato, causando maggiori alternanze nella corrente. Utilizzando il software di analisi automatizzato fornito con lo strumento (Table of Materials), I-Response viene misurato e registrato in termini di unità di biorilevamento (BU)37. I-Response è definita come l’alterazione della corrente (I) attraverso il chip del biosensore misurata in tempo reale sull’interazione del bersaglio immobilizzato con l’analita. Il software di analisi automatizzata FEB può analizzare sia l’I-Response che il C-Response a eventi di interazione dinamica, dove il C-Response registra le alterazioni della capacità (C). Le variazioni sia in I-Response che in C-Response corrispondono direttamente alla frazione di analita legato e possono essere ulteriormente analizzate per generare valori KD . La preferenza predefinita del software di analisi automatizzata è I-Response.
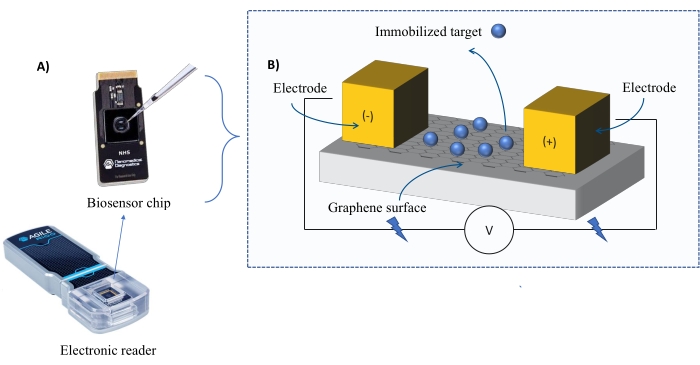
Figura 1: Panoramica della configurazione sperimentale. (A) Chip a base di grafene e lettore elettronico. (B) Una panoramica dei componenti del chip. Il chip è collegato a due elettrodi che forniscono corrente al sistema. La superficie del chip è ricoperta di grafene, che una volta attivato può legare il bersaglio. Fare clic qui per visualizzare una versione più grande di questa figura.
Metodologia:
Inizialmente, il chip del biosensore attivato viene inserito nel dispositivo FEB (Figura 1) seguito dall’esecuzione dei passaggi descritti di seguito: (1) Calibrazione: l’esperimento inizia con la calibrazione del sistema utilizzando 1x soluzione salina tamponata con fosfato (PBS; pH = 7,4) per creare la risposta di equilibrio basale. (2) Associazione: l’analita viene introdotto nel chip e l’I-Response viene monitorato fino al raggiungimento della saturazione del legame. (3) Dissociazione: l’analita viene dissociato utilizzando 1x PBS. (4) Rigenerazione: i resti dell’analita vengono rimossi utilizzando 1x PBS. (5) Lavaggio: un totale di cinque lavaggi vengono eseguiti utilizzando 1x PBS per la rimozione completa degli analiti legati e non legati dal chip.
Analisi:
L’analisi dei dati viene eseguita utilizzando il software completamente automatizzato fornito con lo strumento. Il software di analisi automatizzato genera un grafico Hill fit con un valore KD . Il diagramma di Hill fit descrive l’associazione di un analita alla proteina bersaglio in funzione delle concentrazioni di analiti. La concentrazione alla quale si ottiene una risposta semi-massimale è proporzionale al valore KD . Un valore KD basso rappresenta un’elevata affinità di legame e viceversa.
Per convalidare i dati ottenuti dall’esperimento FEB, le I-Response vengono estratte da ciascun punto di lettura per ogni concentrazione di analita utilizzando il software di revisione/esportazione dei dati e possono essere esportate in altri software di analisi statistica (vedere Tabella dei materiali) come spiegato di seguito.
Protocol
Representative Results
Discussion
In questo studio, è stata valutata la fattibilità dell’utilizzo della tecnologia FEB (un approccio di caratterizzazione cinetica in tempo reale) per determinare l’interazione biomolecolare tra Hsp90 e Cdc37. L’esperimento esplorativo iniziale (primo esperimento) ha suggerito che la scelta delle concentrazioni di analiti corrette è una parte critica dell’esperimento e che l’esperimento dovrebbe essere progettato includendo punti di concentrazione sopra e sotto il valore KD , che sono stati previsti sulla bas…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Questa ricerca è stata sostenuta da una sovvenzione della Binational Science Foundation (BSF) a S.K.S. e N.Q.
Materials
| Automated analysis software | Agile plus software, Cardea (Nanomed) | NA CAS number: NA |
Referred to in the text as the automated analysis software supplied with the instrument. Generates automated analysis. |
| COOH-BPU (Biosensing Processing Unit) | Agile plus software, Cardea (Nanomed) | NA CAS number: NA |
biosensor chip |
| Data review software | Datalign 1.0, Cardea (Nanomed) | NA CAS number: NA |
Referred to as the supplied data review software in the text. Supplied with the instrument and allows to review and export the information data points. |
| Dialysis bag | CelluSep, Membrane filtration products | T2-10-15 CAS number: NA |
T2 tubings (6,000-8,000 MWCO), (10 mm fw, 6.4mm Ø, 0.32ml/cm, 15m) |
| EDC (1-Ethyl-3-(3-dimethylamino propyl) carbodiimide) | Cardea (Nanomed) | EDC160322-02 CAS number: 25952-53-8 |
White powder |
| ITC (Isothermal titration calorimetry) system | Microcal-PEAQ-ITC (Malvern, United Kingdom) | NA CAS number: NA |
|
| MES (2-(N-morpholino) ethane sulfonic acid) buffer | Merck | M3671-50G CAS number: 4432-31-9 |
White powder |
| NHS (N-Hydroxysulfosuccinimide) chips | Cardea (Nanomed) | NA CAS number: NA |
Graphene-based chip |
| PBS (Phosphate-buffered saline) X 10 | Bio-Lab | 001623237500 CAS number: 7758-11-4 |
Liquid transparent solution |
| Pipete | Thermo Scientific | 11855231 CAS number: NA |
Finnpipette F3 5-50 µL, yellow |
| Quench 1 (3.9 mM amino-PEG5-alcohol in 1 X PBS) | Cardea (Nanomed) | 0105-001-002-001 CAS number: NA |
Liquid, transparent solution |
| Quench 2 (1 M ethanolamine (pH=8.5)) | Cardea (Nanomed) | 0105-001-003-001 CAS number: NA |
Liquid, transparent solution |
| Recombinant protein Cdc37 | Abcam | ab256157 CAS number: NA |
|
| Recombinant protein Hsp90 beta | Abcam | ab80033 CAS number: NA |
|
| Spreadsheet | Excel, Microsoft office | NA CAS number: NA |
|
| Statistical software | GraphPad, Prism | NA CAS number: NA |
Referred to as the other statistical software. Sigma plot, phyton or other statistical programes may also be used |
| Sulfo-NHS | Cardea (Nanomed) | NHS160321-07 CAS number: 106627-54-7 |
White powder |
| Tips | Alex red | LC 1093-800-000 CAS number: NA |
Tip 1-200 µl, in bulk, 1,000 pcs |
References
- Tuncbag, N., Gursoy, A., Guney, E., Nussinov, R., Keskin, O. Architectures and functional coverage of protein-protein interfaces. Journal of Molecular Biology. 381 (3), 785-802 (2008).
- Berggård, T., Linse, S., James, P. Methods for the detection and analysis of protein–protein interactions. Proteomics. 7 (16), 2833-2842 (2007).
- Magliery, T. J., et al. Detecting protein-protein interactions with a green fluorescent protein fragment reassembly trap: Scope and mechanism. Journal of the American Chemical Society. 127 (1), 146-157 (2005).
- Xing, S., Wallmeroth, N., Berendzen, K. W., Grefen, C. Techniques for the analysis of protein-protein interactions in vivo. Plant Physiology. 171 (2), 727-758 (2016).
- Nguyen, T. N., Goodrich, J. A. Protein-protein interaction assays: Eliminating false positive interactions. Nature Methods. 3 (2), 135-139 (2006).
- Fernández-Suárez, M., Chen, T. S., Ting, A. Y. Protein-protein interaction detection in vitro and in cells by proximity biotinylation. Journal of the American Chemical Society. 130 (29), 9251-9253 (2008).
- Jiang, M., Niu, C., Cao, J., Ni, D. -. A., Chu, Z. In silico-prediction of protein–protein interactions network about MAPKs and PP2Cs reveals a novel docking site variants in Brachypodium distachyon. Scientific Reports. 8 (1), 15083 (2018).
- Yazawa, M., Sadaghiani, A. M., Hsueh, B., Dolmetsch, R. E. Induction of protein-protein interactions in live cells using light. Nature Biotechnology. 27 (10), 941-945 (2009).
- Wang, W., Goodman, M. T. Antioxidant property of dietary phenolic agents in a human LDL-oxidation ex vivo model: Interaction of protein binding activity. Nutrition Research. 19 (2), 191-202 (1999).
- Xing, S., Wallmeroth, N., Berendzen, K. W., Grefen, C. Techniques for the analysis of protein-protein interactions in vivo. Plant Physiology. 171 (2), 727-758 (2016).
- Qvit, N., Disatnik, M. -. H., Sho, E., Mochly-Rosen, D. Selective phosphorylation inhibitor of delta protein kinase C–Pyruvate dehydrogenase kinase protein–protein interactions: Application for myocardial injury in vivo. Journal of the American Chemical Society. 138 (24), 7626-7635 (2016).
- Alam, M. N., Bristi, N. J., Rafiquzzaman, M. Review on in vivo and in vitro methods evaluation of antioxidant activity. Saudi Pharmaceutical Journal. 21 (2), 143-152 (2013).
- Paulmurugan, R., Gambhir, S. S. Novel fusion protein approach for efficient high-throughput screening of small molecule–mediating protein-protein interactions in cells and living animals. Cancer Research. 65 (16), 7413-7420 (2005).
- Boute, N., Jockers, R., Issad, T. The use of resonance energy transfer in high-throughput screening: BRET versus FRET. Trends in Pharmacological Sciences. 23 (8), 351-354 (2002).
- Deriziotis, P., Graham, S. A., Estruch, S. B., Fisher, S. E. Investigating protein-protein interactions in live cells using bioluminescence resonance energy transfer. Journal of Visualized Experiments: JoVE. (87), e51438 (2014).
- Ekins, S., Mestres, J., Testa, B. In silico pharmacology for drug discovery: Methods for virtual ligand screening and profiling. British Journal of Pharmacology. 152 (1), 9-20 (2007).
- Valerio, L. G. Application of advanced in silico methods for predictive modeling and information integration. Expert Opinion on Drug Metabolism & Toxicology. 8 (4), 395-398 (2012).
- Piehler, J. New methodologies for measuring protein interactions in vivo and in vitro. Current Opinion in Structural Biology. 15 (1), 4-14 (2005).
- Ideker, T., Sharan, R. Protein networks in disease. Genome Research. 18 (4), 644-652 (2008).
- Lu, H., et al. Recent advances in the development of protein–protein interactions modulators: mechanisms and clinical trials. Signal Transduction and Targeted Therapy. 5 (1), 213 (2020).
- Jarosz, D. Hsp90: A global regulator of the genotype-to-phenotype map in cancers. Advances in Cancer Research. 129, 225-247 (2016).
- Johnson, V. A., Singh, E. K., Nazarova, L. A., Alexander, L. D., McAlpine, S. R. Macrocyclic inhibitors of Hsp90. Current Topics in Medicinal Chemistry. 10 (14), 1380-1402 (2010).
- Mahalingam, D., et al. Targeting HSP90 for cancer therapy. British Journal of Cancer. 100 (10), 1523-1529 (2009).
- Stewart, A., Fisher, R. A. Co-Immunoprecipitation: Isolation of protein signaling complexes from native tissues. Methods in Cell Biology. 112, 33-54 (2012).
- Pierce, M. M., Raman, C. S., Nall, B. T. Isothermal titration calorimetry of protein-protein interactions. Methods. 19 (2), 213-221 (1999).
- Paketurytė, V., et al. Inhibitor binding to carbonic anhydrases by isothermal titration calorimetry. Carbonic Anhydrase as Drug Target. , 79-95 (2019).
- Grote, J., Dankbar, N., Gedig, E., Koenig, S. Surface plasmon resonance/mass spectrometry interface. Analytical Chemistry. 77 (4), 1157-1162 (2005).
- Kumaraswamy, S., Tobias, R. Label-free kinetic analysis of an antibody–antigen interaction using biolayer interferometry. Methods in Molecular Biology. , 165-182 (2015).
- Wallner, J., Lhota, G., Jeschek, D., Mader, A., Vorauer-Uhl, K. Application of bio-layer interferometry for the analysis of protein/liposome interactions. Journal of Pharmaceutical and Biomedical Analysis. 72, 150-154 (2013).
- Singh, A. N., Ramadan, K., Singh, S. Experimental methods to study the kinetics of protein–protein interactions. Advances in Protein Molecular and Structural Biology Methods. , 115-124 (2022).
- Frenzel, D., Willbold, D. Kinetic titration series with biolayer interferometry. PLoS One. 9 (9), 106882 (2014).
- Vu, C. -. A., Chen, W. -. Y. Field-effect transistor biosensors for biomedical applications: Recent advances and future prospects. Sensors. 19 (19), 4214 (2019).
- Bergveld, P. A critical evaluation of direct electrical protein detection methods. Biosensors & Bioelectronics. 6 (1), 55-72 (1991).
- Lowe, B. M., Sun, K., Zeimpekis, I., Skylaris, C. K., Green, N. G. Field-effect sensors – from pH sensing to biosensing: sensitivity enhancement using streptavidin–biotin as a model system. The Analyst. 142 (22), 4173-4200 (2017).
- Goldsmith, B. R., et al. Digital biosensing by foundry-fabricated graphene sensors. Scientific Reports. 9 (1), 434 (2019).
- Afsahi, S., et al. Novel graphene-based biosensor for early detection of Zika virus infection. Biosensors and Bioelectronics. 100, 85-88 (2018).
- Afsahi, S. J., et al. Towards novel graphene-enabled diagnostic assays with improved signal-to-noise ratio. MRS Advances. 2 (60), 3733-3739 (2017).
- Roe, S. M., et al. The mechanism of Hsp90 regulation by the protein kinase-specific cochaperone p50cdc37. Cell. 116 (1), 87-98 (2004).
- Gaiser, A. M., Kretzschmar, A., Richter, K. Cdc37-Hsp90 complexes are responsive to nucleotide-induced conformational changes and binding of further cofactors. The Journal of Biological Chemistry. 285 (52), 40921-40932 (2010).
- Popescu, A. I., Găzdaru, D. M., Chilom, C. G., Bacalum, M. Biophysical interactions: Their paramount importance for life. Romanian Reports in Physics. 65 (3), 1063-1077 (2013).
- Surya, S., Abhilash, J., Geethanandan, K., Sadasivan, C., Haridas, M. A profile of protein-protein interaction: Crystal structure of a lectin-lectin complex. International Journal of Biological Macromolecules. 87, 529-536 (2016).
- Velazquez-Campoy, A., Freire, E. ITC in the post-genomic era…? Priceless. Biophysical Chemistry. 115 (23), 115-124 (2005).
- Concepcion, J., et al. Label-free detection of biomolecular interactions using bioLayer interferometry for kinetic characterization. Combinatorial Chemistry & High Throughput Screening. 12 (8), 791-800 (2009).
- Helmerhorst, E., Chandler, D. J., Nussio, M., Mamotte, C. D. Real-time and label-free bio-sensing of molecular interactions by surface plasmon resonance: A laboratory medicine perspective. The Clinical Biochemist. Reviews. 33 (4), 161-173 (2012).
- Jacob, N. T., et al. Synthetic molecules for disruption of the MYC protein-protein interface. Bioorganic & Medicinal Chemistry. 26 (14), 4234-4239 (2018).

