Utforske biomolekylær interaksjon mellom molekylær chaperone Hsp90 og dets klientprotein Kinase Cdc37 ved hjelp av field-effect biosensing teknologi
Summary
Field-effect biosensing (FEB) er en etikettfri teknikk for å oppdage biomolekylære interaksjoner. Den måler den elektriske strømmen gjennom grafenbiosensoren som bindingsmålene immobiliseres til. FEB-teknologien ble brukt til å evaluere biomolekylære interaksjoner mellom Hsp90 og Cdc37, og det ble påvist et sterkt samspill mellom de to proteinene.
Abstract
Biomolekylære interaksjoner spiller allsidige roller i en rekke cellulære prosesser ved å regulere og koordinere funksjonelt relevante biologiske hendelser. Biomolekyler som proteiner, karbohydrater, vitaminer, fettsyrer, nukleinsyrer og enzymer er grunnleggende byggesteiner i levende vesener; de samles i komplekse nettverk i biosystemer for å synkronisere et utall livshendelser. Proteiner bruker vanligvis komplekse interaktivitetsnettverk for å utføre sine funksjoner; Derfor er det obligatorisk å evaluere slike interaksjoner for å avdekke deres betydning i celler på både cellulære og organismenivåer. Mot dette målet introduserer vi en raskt fremvoksende teknologi, felteffekt biosensing (FEB), for å bestemme spesifikke biomolekylære interaksjoner. FEB er en stasjonær, etikettfri og pålitelig biomolekylær deteksjonsteknikk for å bestemme spesifikke interaksjoner og bruker elektroniske biosensorer av høy kvalitet. FEB-teknologien kan overvåke interaksjoner i nanomolarområdet på grunn av de biokompatible nanomaterialene som brukes på biosensoroverflaten. Som et konseptbevis ble proteinproteininteraksjonen (PPI) mellom varmesjokkprotein 90 (Hsp90) og celledelingssyklus 37 (Cdc37) belyst. Hsp90 er en ATP-avhengig molekylær anstand som spiller en viktig rolle i folding, stabilitet, modning og kvalitetskontroll av mange proteiner, og dermed regulerer flere vitale cellulære funksjoner. Cdc37 regnes som en protein kinase-spesifikk molekylær anstand, som det spesifikt anerkjenner og rekrutterer protein kinases til Hsp90 for å regulere deres nedstrøms signaltransduksjonsveier. Som sådan regnes Cdc37 som en med-anstand av Hsp90. Chaperone-kinase-banen (Hsp90/Cdc37-komplekset) er hyperaktivert i flere maligniteter som fremmer cellulær vekst; Derfor er det et potensielt mål for kreftbehandling. Den nåværende studien viser effektiviteten til FEB-teknologi ved hjelp av Hsp90 / Cdc37-modellsystemet. FEB oppdaget en sterk PPI mellom de to proteinene (KD-verdier på 0,014 μM, 0,053 μM og 0,072 μM i tre uavhengige eksperimenter). Oppsummert er FEB en etikettfri og kostnadseffektiv PPI-deteksjonsplattform, som tilbyr raske og nøyaktige målinger.
Introduction
Biomolekylære interaksjoner:
Proteiner er essensielle deler av organismer og deltar i mange molekylære veier som cellemetabolisme, cellestruktur, cellesignalering, immunresponser, celleadhesjon og mer. Mens noen proteiner utfører sin funksjon (er) uavhengig, samhandler de fleste proteiner med andre proteiner ved hjelp av et bindende grensesnitt for å koordinere riktig biologisk aktivitet1.
Biomolekylære interaksjoner kan hovedsakelig klassifiseres basert på de distinkte strukturelle og funksjonelle egenskapene til proteiner involvert2, for eksempel basert på proteinoverflatene, den komplekse stabiliteten eller utholdenheten til interaksjoner3. Identifisering av essensielle proteiner og deres roller i biomolekylære interaksjoner er avgjørende for å forstå biokjemiske mekanismer på molekylærnivå4. For tiden er det forskjellige tilnærminger for å oppdage disse interaksjonene5: in vitro6, i silico7, i levende celler8, ex vivo9 og in vivo10 med hver har sine egne styrker og svakheter.
In vivo-analysene utføres ved hjelp av hele dyret som et eksperimentelt verktøy11, oghan eks vivo-analyser utføres på vevsekstrakter eller hele organer (f.eks. hjerte, hjerne, lever) i et kontrollert eksternt miljø ved å gi minimale endringer i naturlige forhold. Den vanligste anvendelsen av in vivo– og ex vivo-studier er å evaluere farmakokinetikk, farmakodynamikk og toksisitetseffekter av potensielle farmakologiske midler før menneskelige studier ved å sikre deres generelle sikkerhet og effekt12.
Biomolekylære interaksjoner kan også påvises i levende celler. Imaging levende celler tillater oss å observere dynamiske interaksjoner som de utfører reaksjonene av en bestemt biokjemisk vei13. Videre kan deteksjonsteknikker, for eksempel bioluminescens eller fluorescensresonansenergioverføring, gi informasjon om hvor og når disse interaksjonene skjer i celle14. Selv om deteksjon i levende celler gir viktige detaljer, er disse deteksjonsmetodene avhengige av optikk og etiketter, noe som kanskje ikke gjenspeiler den opprinnelige biologien; de er også mindre kontrollerte enn in vitro-metoder og krever spesialisert kompetanse for å utføre15.
I silico beregningsmetoder brukes primært til storskala screening av målmolekyler før in vitro-forsøkene. Beregningsmessige prediksjonsmetoder, databaserte databaser, molekylær dokking, kvantitative strukturaktivitetsrelasjoner og andre molekylære dynamikksimuleringsmetoder er blant de veletablerte i silicoverktøy 16. Sammenlignet med arbeidskrevende eksperimentelle teknikker, kan in silico-verktøyene enkelt gjøre spådommer med høy følsomhet, men med redusert nøyaktighet i prediktiv ytelse17.
In vitro-analyser utføres med mikroorganismer eller biologiske molekyler utenfor deres standard biologiske kontekst. Skildring av biomolekylære interaksjoner gjennom in vitro-metoder er avgjørende for å forstå proteinfunksjoner og biologien bak det komplekse nettverket av cellefunksjon. Den foretrukne analysemetodikken velges i henhold til proteinets iboende egenskaper, kinetiske verdier og modusen og intensiteten av interaksjoner 18,19.
Hsp90/Cdc37-interaksjonen:
Chaperone-kinase-banen, som forbinder Hsp90 og Cdc37, er et lovende terapeutisk mål i tumorbiologi20. Hsp90 spiller en sentral rolle i cellesykluskontroll, proteinmontering, celleoverlevelse og signalveier. Proteiner som er avhengige av Hsp90 for deres funksjoner, leveres til Hsp90 for kompleksitet gjennom en medprest, for eksempel Cdc37. Hsp90/Cdc37-komplekset styrer foldingen av de fleste proteinkinaser og fungerer som et knutepunkt for en rekke intracellulære signalnettverk21. Det er et lovende anti-tumor mål på grunn av sin forhøyede uttrykk i ulike maligniteter, inkludert akutt myeloblastisk leukemi, multippel myeloma, og hepatocellulært karsinom22,23.
Vanlige in vitro biomolekylære interaksjonsdeteksjonsteknikker
Co-immunoprecipitation (co-IP) er en teknikk som er avhengig av antigen-antistoff spesifisitet for å identifisere biologisk relevante interaksjoner24. Den største ulempen ved denne metoden er dens manglende evne til å oppdage interaksjoner med lav affinitet og kinetiske verdier24. Biofysiske metoder som isotermisk titreringskalorimetri (ITC), overflateplasmonresonans (SPR), biolayer interferometri (BLI) og FEB-teknologi foretrekkes for å bestemme kinetiske verdier.
ITC er en biofysisk deteksjonsmetode basert på bestemmelse av bindende energi sammen med en komplett termodynamikkanalyse for å karakterisere biomolekylære interaksjoner25. Den viktigste fordelen med ITC er at det ikke krever merking eller fiksering av målproteinet. De viktigste vanskelighetene som ITC møter er den høye konsentrasjonen av målprotein som kreves for ett eksperiment og vanskeligheten med å analysere ikke-kovalente komplekser på grunn av små bindende entalpier26. Både SPR og BLI er etikettfrie biofysiske teknikker som er avhengige av immobilisering av målmolekylet på sensoroverflaten, etterfulgt av påfølgende injeksjoner av analytten over det immobiliserte målet27,28. I SPR måles endringer i brytningsindeksen under biomolekylære interaksjoner27; I BLI registreres interferensen i reflektert lys i sanntid som en endring i bølgelengden som en funksjon av tid28. Både SPR og BLI deler felles fordeler ved å tilby høy spesifisitet, følsomhet og deteksjonsevne29. I begge metoder er målproteinet immobilisert på biosensoroverflater, og derfor kan det være noe tap av målets opprinnelige konformasjon, noe som gjør det vanskelig å diskriminere mellom spesifikke kontra ikke-spesifikke interaksjoner30. BLI bruker dyre engangs fiberoptiske biosensorer for å immobilisere målet, og er derfor en kostbar teknikk31. Sammenlignet med disse veletablerte biomolekylære deteksjonsverktøyene, tilbyr FEB-teknologi en pålitelig og etikettfri plattform ved å bruke lave nanomolarkonsentrasjoner for biomolekylær deteksjon i sanntid med kinetisk karakterisering. FEB-teknologien overvinner også de boblende utfordringene i ITC og er mer kostnadseffektiv sammenlignet med SPR eller BLI.
Den felteffekttransistor (FET) basert biosensorer er et fremvoksende felt for å oppdage biomolekylære interaksjoner ved å tilby varierte biomedisinske applikasjoner. I FET-systemet blir mål immobilisert til biosensorbrikkene og interaksjoner oppdages ved endringer i ledning32. Den unike egenskapen som skal vurderes i utviklingen av en effektiv elektronisk biosensor er de fysisk-kjemiske egenskapene som den halvledende naturen og kjemisk stabilitet av beleggmaterialet som brukes til å fremstille sensoroverflaten33. Konvensjonelle materialer som silisium som brukes til FET har begrenset følsomheten til sensorer fordi det krever oksidlag smurt mellom transistorkanalen og et spesifikt miljø for riktig funksjon34. Videre er silisiumtransistorer følsomme for høye saltmiljøer, noe som gjør det vanskelig å måle biologiske interaksjoner i sitt naturlige miljø. Den grafenbaserte biosensoren presenteres som et alternativ, da den tilbyr utmerket kjemisk stabilitet og elektrisk felt. Siden grafen er et enkelt atomlag av karbon, er det både ekstremt følsomt som en halvleder og kjemisk kompatibel med biologiske løsninger; begge disse egenskapene er ønskelige å generere kompatible elektroniske biosensorer35. Det bemerkelsesverdige ultrahøye lastepotensialet til biomolekyler som tilbys av grafenbelagte biosensorer fører til utvikling av grafenbaserte biosensorer FEB-teknologi.
Prinsipp for FEB-teknologi: FEB er en etikettfri biomolekylær deteksjonsteknikk som måler den elektriske strømmen gjennom grafenbiosensoren som bindingsmålene immobiliseres til. Interaksjoner mellom det immobiliserte proteinet og analytten resulterer i endringer i strøm som overvåkes i sanntid, noe som muliggjør nøyaktige kinetiske målinger36.
Instrumentering: FEB-systemet består av en grafenfelteffekttransistorsensor (gFET) og en elektronisk leser som bruker konstant spenning gjennom hele eksperimentet (figur 1). Analytten påføres i løsning på målproteinet immobilisert på biosensoroverflaten. Når en interaksjon oppstår, måles og registreres en endring i strømmen i sanntid. Etter hvert som analyttkonsentrasjonen øker, vil brøkdelen av bundet analytt også øke, noe som forårsaker høyere vekslinger i strømmen. Ved hjelp av den automatiserte analyseprogramvaren som følger med instrumentet (Materialfortegnelse), måles og registreres I-Response når det gjelder biosensing-enheter (BU)37. I-Response er definert som endringen i den nåværende (I) gjennom biosensorbrikken målt i sanntid etter samspillet mellom det immobiliserte målet med analytten. Feb automatisert analyse programvare kan analysere både I-Response og C-Response til dynamiske interaksjon hendelser, der C-Response registrerer endringene i kapasitans (C). Variasjonene i både I-Response og C-Response samsvarer direkte med brøkdelen av bundet analytt og kan analyseres ytterligere for å generere KD-verdier . Standardinnstillingen for den automatiserte analyseprogramvaren er I-Response.
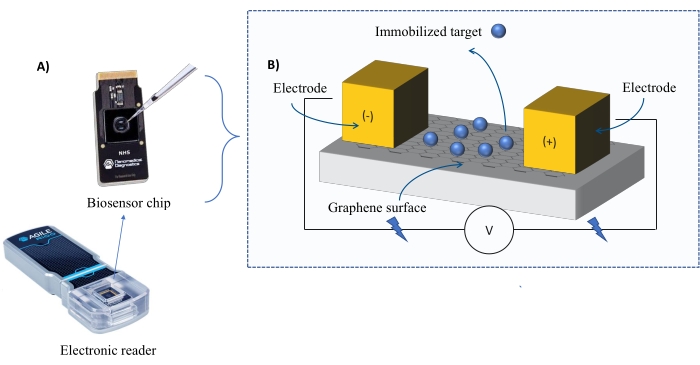
Figur 1: Oversikt over det eksperimentelle oppsettet. (A) Grafenbasert brikke og en elektronisk leser. (B) En oversikt over brikkekomponentene. Brikken er festet til to elektroder som leverer strøm til systemet. Overflaten på brikken er dekket med grafen, som når den aktiveres kan binde målet. Klikk her for å se en større versjon av denne figuren.
Metodikk:
I utgangspunktet settes den aktiverte biosensorbrikken inn i FEB-enheten (figur 1) etterfulgt av utførelsen av trinnene som er beskrevet nedenfor: (1) Kalibrering: Eksperimentet starter med systemkalibrering ved hjelp av 1x fosfatbufret saltvann (PBS; pH = 7,4) for å skape baseline likevektsrespons. (2) Forening: Analytten innføres i brikken, og I-Response overvåkes til bindingsmetningen er nådd. (3) Dissosiasjon: Analytten er dissosiert ved hjelp av 1x PBS. (4) Regenerering: Rester av analytten fjernes ved hjelp av 1x PBS. (5) Vasking: Totalt fem vasker utføres ved hjelp av 1x PBS for grundig fjerning av de bundne og ubundne analyttene fra brikken.
Analyse:
Dataanalyse utføres ved hjelp av den helautomatiske programvaren som følger med instrumentet. Den automatiserte analyseprogramvaren genererer en Hill fit-tomt med en KD-verdi . Hill fit plott beskriver foreningen av en analytt til målproteinet som en funksjon av analyttkonsentrasjoner. Konsentrasjonen der en halvmaksimal respons oppnås er proporsjonal med KD-verdien . En lav KD-verdi representerer høy bindingsaffinitet og omvendt.
For å validere dataene som er hentet fra FEB-eksperimentet, blir I-Responses hentet fra hvert avlesningspunkt for hver analyttkonsentrasjon ved hjelp av datagjennomgangs-/eksportprogramvaren og kan eksporteres til annen statistisk analyseprogramvare (se Tabell over materialer) som forklart nedenfor.
Protocol
Representative Results
Discussion
I denne studien ble muligheten for å bruke FEB-teknologien (en kinetisk karakteriseringstilnærming i sanntid) evaluert for å bestemme den biomolekylære interaksjonen mellom Hsp90 og Cdc37. Det første utforskende eksperimentet (første eksperiment) antydet at valg av riktige analyttkonsentrasjoner er en kritisk del av eksperimentet, og at eksperimentet bør utformes ved å inkludere konsentrasjonspunkter over og under KD-verdien , som ble spådd basert på dataene som er tilgjengelige i litteraturen.
<…Disclosures
The authors have nothing to disclose.
Acknowledgements
Denne forskningen ble støttet av et stipend fra Binational Science Foundation (BSF) til S.K.S. og N.Q.
Materials
| Automated analysis software | Agile plus software, Cardea (Nanomed) | NA CAS number: NA |
Referred to in the text as the automated analysis software supplied with the instrument. Generates automated analysis. |
| COOH-BPU (Biosensing Processing Unit) | Agile plus software, Cardea (Nanomed) | NA CAS number: NA |
biosensor chip |
| Data review software | Datalign 1.0, Cardea (Nanomed) | NA CAS number: NA |
Referred to as the supplied data review software in the text. Supplied with the instrument and allows to review and export the information data points. |
| Dialysis bag | CelluSep, Membrane filtration products | T2-10-15 CAS number: NA |
T2 tubings (6,000-8,000 MWCO), (10 mm fw, 6.4mm Ø, 0.32ml/cm, 15m) |
| EDC (1-Ethyl-3-(3-dimethylamino propyl) carbodiimide) | Cardea (Nanomed) | EDC160322-02 CAS number: 25952-53-8 |
White powder |
| ITC (Isothermal titration calorimetry) system | Microcal-PEAQ-ITC (Malvern, United Kingdom) | NA CAS number: NA |
|
| MES (2-(N-morpholino) ethane sulfonic acid) buffer | Merck | M3671-50G CAS number: 4432-31-9 |
White powder |
| NHS (N-Hydroxysulfosuccinimide) chips | Cardea (Nanomed) | NA CAS number: NA |
Graphene-based chip |
| PBS (Phosphate-buffered saline) X 10 | Bio-Lab | 001623237500 CAS number: 7758-11-4 |
Liquid transparent solution |
| Pipete | Thermo Scientific | 11855231 CAS number: NA |
Finnpipette F3 5-50 µL, yellow |
| Quench 1 (3.9 mM amino-PEG5-alcohol in 1 X PBS) | Cardea (Nanomed) | 0105-001-002-001 CAS number: NA |
Liquid, transparent solution |
| Quench 2 (1 M ethanolamine (pH=8.5)) | Cardea (Nanomed) | 0105-001-003-001 CAS number: NA |
Liquid, transparent solution |
| Recombinant protein Cdc37 | Abcam | ab256157 CAS number: NA |
|
| Recombinant protein Hsp90 beta | Abcam | ab80033 CAS number: NA |
|
| Spreadsheet | Excel, Microsoft office | NA CAS number: NA |
|
| Statistical software | GraphPad, Prism | NA CAS number: NA |
Referred to as the other statistical software. Sigma plot, phyton or other statistical programes may also be used |
| Sulfo-NHS | Cardea (Nanomed) | NHS160321-07 CAS number: 106627-54-7 |
White powder |
| Tips | Alex red | LC 1093-800-000 CAS number: NA |
Tip 1-200 µl, in bulk, 1,000 pcs |
References
- Tuncbag, N., Gursoy, A., Guney, E., Nussinov, R., Keskin, O. Architectures and functional coverage of protein-protein interfaces. Journal of Molecular Biology. 381 (3), 785-802 (2008).
- Berggård, T., Linse, S., James, P. Methods for the detection and analysis of protein–protein interactions. Proteomics. 7 (16), 2833-2842 (2007).
- Magliery, T. J., et al. Detecting protein-protein interactions with a green fluorescent protein fragment reassembly trap: Scope and mechanism. Journal of the American Chemical Society. 127 (1), 146-157 (2005).
- Xing, S., Wallmeroth, N., Berendzen, K. W., Grefen, C. Techniques for the analysis of protein-protein interactions in vivo. Plant Physiology. 171 (2), 727-758 (2016).
- Nguyen, T. N., Goodrich, J. A. Protein-protein interaction assays: Eliminating false positive interactions. Nature Methods. 3 (2), 135-139 (2006).
- Fernández-Suárez, M., Chen, T. S., Ting, A. Y. Protein-protein interaction detection in vitro and in cells by proximity biotinylation. Journal of the American Chemical Society. 130 (29), 9251-9253 (2008).
- Jiang, M., Niu, C., Cao, J., Ni, D. -. A., Chu, Z. In silico-prediction of protein–protein interactions network about MAPKs and PP2Cs reveals a novel docking site variants in Brachypodium distachyon. Scientific Reports. 8 (1), 15083 (2018).
- Yazawa, M., Sadaghiani, A. M., Hsueh, B., Dolmetsch, R. E. Induction of protein-protein interactions in live cells using light. Nature Biotechnology. 27 (10), 941-945 (2009).
- Wang, W., Goodman, M. T. Antioxidant property of dietary phenolic agents in a human LDL-oxidation ex vivo model: Interaction of protein binding activity. Nutrition Research. 19 (2), 191-202 (1999).
- Xing, S., Wallmeroth, N., Berendzen, K. W., Grefen, C. Techniques for the analysis of protein-protein interactions in vivo. Plant Physiology. 171 (2), 727-758 (2016).
- Qvit, N., Disatnik, M. -. H., Sho, E., Mochly-Rosen, D. Selective phosphorylation inhibitor of delta protein kinase C–Pyruvate dehydrogenase kinase protein–protein interactions: Application for myocardial injury in vivo. Journal of the American Chemical Society. 138 (24), 7626-7635 (2016).
- Alam, M. N., Bristi, N. J., Rafiquzzaman, M. Review on in vivo and in vitro methods evaluation of antioxidant activity. Saudi Pharmaceutical Journal. 21 (2), 143-152 (2013).
- Paulmurugan, R., Gambhir, S. S. Novel fusion protein approach for efficient high-throughput screening of small molecule–mediating protein-protein interactions in cells and living animals. Cancer Research. 65 (16), 7413-7420 (2005).
- Boute, N., Jockers, R., Issad, T. The use of resonance energy transfer in high-throughput screening: BRET versus FRET. Trends in Pharmacological Sciences. 23 (8), 351-354 (2002).
- Deriziotis, P., Graham, S. A., Estruch, S. B., Fisher, S. E. Investigating protein-protein interactions in live cells using bioluminescence resonance energy transfer. Journal of Visualized Experiments: JoVE. (87), e51438 (2014).
- Ekins, S., Mestres, J., Testa, B. In silico pharmacology for drug discovery: Methods for virtual ligand screening and profiling. British Journal of Pharmacology. 152 (1), 9-20 (2007).
- Valerio, L. G. Application of advanced in silico methods for predictive modeling and information integration. Expert Opinion on Drug Metabolism & Toxicology. 8 (4), 395-398 (2012).
- Piehler, J. New methodologies for measuring protein interactions in vivo and in vitro. Current Opinion in Structural Biology. 15 (1), 4-14 (2005).
- Ideker, T., Sharan, R. Protein networks in disease. Genome Research. 18 (4), 644-652 (2008).
- Lu, H., et al. Recent advances in the development of protein–protein interactions modulators: mechanisms and clinical trials. Signal Transduction and Targeted Therapy. 5 (1), 213 (2020).
- Jarosz, D. Hsp90: A global regulator of the genotype-to-phenotype map in cancers. Advances in Cancer Research. 129, 225-247 (2016).
- Johnson, V. A., Singh, E. K., Nazarova, L. A., Alexander, L. D., McAlpine, S. R. Macrocyclic inhibitors of Hsp90. Current Topics in Medicinal Chemistry. 10 (14), 1380-1402 (2010).
- Mahalingam, D., et al. Targeting HSP90 for cancer therapy. British Journal of Cancer. 100 (10), 1523-1529 (2009).
- Stewart, A., Fisher, R. A. Co-Immunoprecipitation: Isolation of protein signaling complexes from native tissues. Methods in Cell Biology. 112, 33-54 (2012).
- Pierce, M. M., Raman, C. S., Nall, B. T. Isothermal titration calorimetry of protein-protein interactions. Methods. 19 (2), 213-221 (1999).
- Paketurytė, V., et al. Inhibitor binding to carbonic anhydrases by isothermal titration calorimetry. Carbonic Anhydrase as Drug Target. , 79-95 (2019).
- Grote, J., Dankbar, N., Gedig, E., Koenig, S. Surface plasmon resonance/mass spectrometry interface. Analytical Chemistry. 77 (4), 1157-1162 (2005).
- Kumaraswamy, S., Tobias, R. Label-free kinetic analysis of an antibody–antigen interaction using biolayer interferometry. Methods in Molecular Biology. , 165-182 (2015).
- Wallner, J., Lhota, G., Jeschek, D., Mader, A., Vorauer-Uhl, K. Application of bio-layer interferometry for the analysis of protein/liposome interactions. Journal of Pharmaceutical and Biomedical Analysis. 72, 150-154 (2013).
- Singh, A. N., Ramadan, K., Singh, S. Experimental methods to study the kinetics of protein–protein interactions. Advances in Protein Molecular and Structural Biology Methods. , 115-124 (2022).
- Frenzel, D., Willbold, D. Kinetic titration series with biolayer interferometry. PLoS One. 9 (9), 106882 (2014).
- Vu, C. -. A., Chen, W. -. Y. Field-effect transistor biosensors for biomedical applications: Recent advances and future prospects. Sensors. 19 (19), 4214 (2019).
- Bergveld, P. A critical evaluation of direct electrical protein detection methods. Biosensors & Bioelectronics. 6 (1), 55-72 (1991).
- Lowe, B. M., Sun, K., Zeimpekis, I., Skylaris, C. K., Green, N. G. Field-effect sensors – from pH sensing to biosensing: sensitivity enhancement using streptavidin–biotin as a model system. The Analyst. 142 (22), 4173-4200 (2017).
- Goldsmith, B. R., et al. Digital biosensing by foundry-fabricated graphene sensors. Scientific Reports. 9 (1), 434 (2019).
- Afsahi, S., et al. Novel graphene-based biosensor for early detection of Zika virus infection. Biosensors and Bioelectronics. 100, 85-88 (2018).
- Afsahi, S. J., et al. Towards novel graphene-enabled diagnostic assays with improved signal-to-noise ratio. MRS Advances. 2 (60), 3733-3739 (2017).
- Roe, S. M., et al. The mechanism of Hsp90 regulation by the protein kinase-specific cochaperone p50cdc37. Cell. 116 (1), 87-98 (2004).
- Gaiser, A. M., Kretzschmar, A., Richter, K. Cdc37-Hsp90 complexes are responsive to nucleotide-induced conformational changes and binding of further cofactors. The Journal of Biological Chemistry. 285 (52), 40921-40932 (2010).
- Popescu, A. I., Găzdaru, D. M., Chilom, C. G., Bacalum, M. Biophysical interactions: Their paramount importance for life. Romanian Reports in Physics. 65 (3), 1063-1077 (2013).
- Surya, S., Abhilash, J., Geethanandan, K., Sadasivan, C., Haridas, M. A profile of protein-protein interaction: Crystal structure of a lectin-lectin complex. International Journal of Biological Macromolecules. 87, 529-536 (2016).
- Velazquez-Campoy, A., Freire, E. ITC in the post-genomic era…? Priceless. Biophysical Chemistry. 115 (23), 115-124 (2005).
- Concepcion, J., et al. Label-free detection of biomolecular interactions using bioLayer interferometry for kinetic characterization. Combinatorial Chemistry & High Throughput Screening. 12 (8), 791-800 (2009).
- Helmerhorst, E., Chandler, D. J., Nussio, M., Mamotte, C. D. Real-time and label-free bio-sensing of molecular interactions by surface plasmon resonance: A laboratory medicine perspective. The Clinical Biochemist. Reviews. 33 (4), 161-173 (2012).
- Jacob, N. T., et al. Synthetic molecules for disruption of the MYC protein-protein interface. Bioorganic & Medicinal Chemistry. 26 (14), 4234-4239 (2018).

