Moleküler Chaperone Hsp90 ve İstemci Protein Kinaz Cdc37 Arasındaki Biyomoleküler Etkileşimi Alan Etkili Biyosensing Teknolojisini Kullanarak Keşfetmek
Summary
Alan etkili biyosensing (FEB), biyomoleküler etkileşimleri tespit etmek için etiketsiz bir tekniktir. Elektrik akımını, bağlanma hedeflerinin hareketsiz bırakıldığı grafen biyosensörü aracılığıyla ölçer. Hsp90 ve Cdc37 arasındaki biyomoleküler etkileşimleri değerlendirmek için FEB teknolojisi kullanıldı ve iki protein arasında güçlü bir etkileşim tespit edildi.
Abstract
Biyomoleküler etkileşimler, fonksiyonel olarak ilgili biyolojik olayları düzenleyerek ve koordine ederek çok sayıda hücresel süreçte çok yönlü roller oynar. Proteinler, karbonhidratlar, vitaminler, yağ asitleri, nükleik asitler ve enzimler gibi biyomoleküller canlıların temel yapı taşlarıdır; sayısız yaşam olayını senkronize etmek için biyosistemlerdeki karmaşık ağlarda bir araya gelirler. Proteinler tipik olarak işlevlerini yerine getirmek için karmaşık interaktom ağları kullanırlar; bu nedenle, hem hücresel hem de organizma düzeylerindeki hücrelerdeki önemlerini çözmek için bu tür etkileşimleri değerlendirmek zorunludur. Bu amaç doğrultusunda, spesifik biyomoleküler etkileşimleri belirlemek için hızla gelişen bir teknoloji olan alan etkisi biyosensing’i (FEB) tanıtıyoruz. FEB, belirli etkileşimleri belirlemek için tezgah üstü, etiketsiz ve güvenilir bir biyomoleküler tespit tekniğidir ve yüksek kaliteli elektronik tabanlı biyosensörler kullanır. FEB teknolojisi, biyosensör yüzeyinde kullanılan biyouyumlu nanomalzemeler nedeniyle nanomolar aralıktaki etkileşimleri izleyebilir. Kavramın bir kanıtı olarak, ısı şoku proteini 90 (Hsp90) ve hücre bölünme döngüsü 37 (Cdc37) arasındaki protein-protein etkileşimi (PPI) açıklığa kavuşturulmuştur. Hsp90, birçok proteinin katlanması, stabilitesi, olgunlaşması ve kalite kontrolünde önemli bir rol oynayan, böylece çoklu hayati hücresel fonksiyonları düzenleyen ATP’ye bağımlı bir moleküler şaperondur. Cdc37, protein kinazına özgü bir moleküler şaperon olarak kabul edilir, çünkü protein kinazlarını özellikle tanır ve aşağı akış sinyal iletim yollarını düzenlemek için Hsp90’a alır. Bu nedenle, Cdc37, Hsp90’ın bir ko-şaperonu olarak kabul edilir. Şaperon-kinaz yolu (Hsp90 / Cdc37 kompleksi) hücresel büyümeyi teşvik eden çoklu malignitelerde hiper-aktive edilir; bu nedenle, kanser tedavisi için potansiyel bir hedeftir. Bu çalışma, Hsp90/Cdc37 model sistemini kullanarak FEB teknolojisinin verimliliğini göstermektedir. FEB, iki protein arasında güçlü bir PPI tespit etti (üç bağımsız deneyde 0.014 μM, 0.053 μM ve 0.072 μM KD değerleri). Özetle, FEB, hızlı ve doğru ölçümler sunan, etiketsiz ve uygun maliyetli bir PPI algılama platformudur.
Introduction
Biyomoleküler etkileşimler:
Proteinler organizmaların temel parçalarıdır ve hücre metabolizması, hücre yapısı, hücre sinyalizasyonu, bağışıklık tepkileri, hücre yapışması ve daha fazlası gibi çok sayıda moleküler yola katılırlar. Bazı proteinler işlevlerini bağımsız olarak yerine getirirken, çoğu protein, uygun biyolojik aktiviteyi koordine etmek için bir bağlayıcı arayüz kullanarak diğer proteinlerle etkileşime girer1.
Biyomoleküler etkileşimler esas olarak, örneğin protein yüzeylerine, karmaşık stabiliteye veya etkileşimlerin kalıcılığına bağlı olarak2 ile ilgili proteinlerin farklı yapısal ve fonksiyonel özelliklerine göre sınıflandırılabilir3. Esansiyel proteinleri ve biyomoleküler etkileşimlerdeki rollerini tanımlamak, moleküler düzeyde biyokimyasal mekanizmaları anlamak için hayati önemesahiptir 4. Şu anda, bu etkileşimleri tespit etmek için çeşitli yaklaşımlar vardır5: in vitro6, in siliko7, canlı hücrelerde8, ex vivo 9 ve in vivo10, her biri kendi güçlü ve zayıf yönlerine sahiptir.
İn vivo tahliller, tüm hayvanı deneysel bir araç olarak kullanarak gerçekleştirilir11 veex vivo tahliller, doğal koşullarda minimum değişiklik sağlayarak kontrollü bir dış ortamda doku ekstraktları veya tüm organlar (örneğin, kalp, beyin, karaciğer) üzerinde gerçekleştirilir. İn vivo ve ex vivo çalışmaların en yaygın uygulaması, potansiyel farmakolojik ajanların farmakokinetik, farmakodinamik ve toksisite etkilerini, genel güvenlik ve etkinliklerini sağlayarak insan denemelerinden önce değerlendirmektir12.
Biyomoleküler etkileşimler canlı hücreler içinde de tespit edilebilir. Canlı hücrelerin görüntülenmesi, belirli bir biyokimyasal yolun reaksiyonlarını yürütürken dinamik etkileşimleri gözlemlememizi sağlar13. Ayrıca, biyolüminesans veya floresan rezonans enerji transferi gibi tespit teknikleri, bu etkileşimlerin hücre14 içinde nerede ve ne zaman gerçekleştiği hakkında bilgi sağlayabilir. Canlı hücrelerde algılama çok önemli ayrıntılar sunsa da, bu tespit metodolojileri yerel biyolojiyi yansıtmayabilecek optik ve etiketlere dayanır; Ayrıca in vitro yöntemlerden daha az kontrollüdürler ve15’i gerçekleştirmek için özel uzmanlık gerektirirler.
In silico hesaplama yöntemleri öncelikle in vitro deneylerden önce hedef moleküllerin büyük ölçekli taranması için kullanılır. Hesaplamalı tahmin yöntemleri, bilgisayar tabanlı veritabanları, moleküler kenetlenme, nicel yapı-aktivite ilişkileri ve diğer moleküler dinamik simülasyon yaklaşımları köklü in silico araçları arasındadır16. Zahmetli deneysel tekniklerle karşılaştırıldığında, in silico aletleri yüksek hassasiyetle kolayca tahminlerde bulunabilir, ancak öngörücü performansta daha düşük doğrulukla17.
In vitro testler, standart biyolojik bağlamlarının dışındaki mikroorganizmalar veya biyolojik moleküllerle gerçekleştirilir. Biyomoleküler etkileşimleri in vitro yöntemlerle tasvir etmek, protein fonksiyonlarını ve karmaşık hücre işleyiş ağının arkasındaki biyolojiyi anlamak için kritik öneme sahiptir. Tercih edilen tahlil metodolojisi, proteinin içsel özelliklerine, kinetik değerlerine ve etkileşimlerin modu ve yoğunluğuna göre seçilir18,19.
Hsp90/Cdc37 etkileşimi:
Hsp90 ve Cdc37’yi birbirine bağlayan şaperon-kinaz yolu, tümör biyolojisi20’de umut verici bir terapötik hedeftir. Hsp90, hücre döngüsü kontrolünde, protein montajında, hücre sağkalımında ve sinyal yollarında merkezi bir rol oynar. İşlevleri için Hsp90’a güvenen proteinler, Cdc37 gibi bir ko-şaperon aracılığıyla kompleksleşme için Hsp90’a verilir. Hsp90 / Cdc37 kompleksi, çoğu protein kinazının katlanmasını kontrol eder ve çok sayıda hücre içi sinyal ağı için bir merkez görevi görür21. Akut miyeloblastik lösemi, multipl miyelom ve hepatosellüler karsinom22,23 gibi çeşitli malignitelerde yüksek ekspresyonu nedeniyle umut verici bir anti-tümör hedefidir.
Yaygın olarak kullanılan in vitro biyomoleküler etkileşim tespit teknikleri
Ko-immünopresipitasyon (co-IP), biyolojik olarak ilgili etkileşimleri tanımlamak için antijen-antikor özgüllüğüne dayanan bir tekniktir24. Bu yöntemin birincil dezavantajı, düşük afiniteli etkileşimleri ve kinetik değerleri tespit edememesidir24. Kinetik değerlerin belirlenmesinde izotermal titrasyon kalorimetrisi (ITC), yüzey plazmon rezonansı (SPR), biyolayer interferometrisi (BLI) ve FEB teknolojisi gibi biyofiziksel yöntemler tercih edilmektedir.
ITC, biyomoleküler etkileşimleri karakterize etmek için eksiksiz bir termodinamik analiz ile birlikte bağlanma enerjisinin belirlenmesine dayanan biyofiziksel bir tespit yöntemidir25. ITC’nin birincil avantajı, hedef proteinin herhangi bir etiketlenmesini veya sabitlenmesini gerektirmemesidir. ITC’nin karşılaştığı başlıca zorluklar, bir deney için gereken yüksek hedef protein konsantrasyonu ve küçük bağlayıcı entalpiler nedeniyle kovalent olmayan komplekslerin analizindeki zorluktur26. Hem SPR hem de BLI, sensör yüzeyindeki hedef molekülün immobilizasyonuna dayanan etiketsiz biyofiziksel tekniklerdir, ardından hareketsiz hedef27,28 üzerine analitin daha sonra enjekte edilmesidir. SPR’de, biyomoleküler etkileşimler sırasında kırılma indisindeki değişiklikler ölçülür27; BLI’da, yansıyan ışıktaki girişim,zamanın 28’inin bir fonksiyonu olarak dalga boyundaki bir değişiklik olarak gerçek zamanlı olarak kaydedilir. Hem SPR hem de BLI, yüksek özgüllük, hassasiyet ve algılama yetenekleri sunmanın ortak avantajlarını paylaşır29. Her iki yöntemde de, hedef protein biyosensör yüzeyleri üzerinde hareketsiz hale getirilir ve bu nedenle, hedefin doğal konformasyonunda bir miktar kayıp olabilir, bu da spesifik ve spesifik olmayan etkileşimler arasında ayrım yapmayı zorlaştırır30. BLI, hedefi hareketsiz hale getirmek için pahalı tek kullanımlık fiber optik biyosensörler kullanır ve bu nedenle pahalı bir tekniktir31. Bu köklü biyomoleküler tespit araçlarıyla karşılaştırıldığında, FEB teknolojisi, kinetik karakterizasyon ile gerçek zamanlı olarak biyomoleküler algılama için düşük nanomolar konsantrasyonlar kullanarak güvenilir ve etiketsiz bir platform sunar. FEB teknolojisi ayrıca ITC’de karşılaşılan köpüren zorlukların üstesinden gelir ve SPR veya BLI’ya kıyasla daha uygun maliyetlidir.
Alan etkili transistör (FET) tabanlı biyosensörler, çeşitli biyomedikal uygulamalar sunarak biyomoleküler etkileşimleri tespit etmek için ortaya çıkan bir alandır. FET sisteminde, hedefler biyosensör çiplerine hareketsiz hale getirilir ve etkileşimler iletkenlikteki değişikliklerle tespit edilir32. Verimli bir elektronik biyosensörün geliştirilmesinde göz önünde bulundurulması gereken benzersiz özellik, sensör yüzeyini imal etmek için kullanılan kaplama malzemesinin yarı iletken doğası ve kimyasal kararlılığı gibi fizikokimyasal özelliklerdir33. FET için kullanılan silikon gibi geleneksel malzemeler, sensörlerin hassasiyetini sınırlamıştır, çünkü transistör kanalı ile düzgün çalışması için belirli bir ortam arasında sıkışmış oksit tabakaları gerektirir34. Dahası, silikon transistörler yüksek tuz ortamlarına duyarlıdır, bu nedenle doğal ortamlarındaki biyolojik etkileşimleri ölçmeyi zorlaştırır. Grafen bazlı biyosensör, mükemmel kimyasal stabilite ve elektrik alanı sunduğu için alternatif olarak sunulmaktadır. Grafen tek bir atomik karbon tabakası olduğundan, hem yarı iletken olarak son derece hassastır hem de biyolojik çözeltilerle kimyasal olarak uyumludur; Bu niteliklerin her ikisi de uyumlu elektronik biyosensörler üretmek için arzu edilir35. Grafen kaplı biyosensörler tarafından sunulan biyomoleküllerin olağanüstü ultra yüksek yükleme potansiyeli, grafen bazlı biyosensörlerin FEB teknolojisinin geliştirilmesine yol açmaktadır.
FEB teknolojisinin prensibi: FEB, bağlanma hedeflerinin hareketsiz bırakıldığı grafen biyosensör aracılığıyla elektrik akımını ölçen etiketsiz bir biyomoleküler tespit tekniğidir. İmmobilize protein ve analit arasındaki etkileşimler, gerçek zamanlı olarak izlenen akımdaki değişikliklere neden olur ve doğru kinetik ölçümler sağlar36.
Enstrümantasyon: FEB sistemi, bir grafen alan etkili transistör (gFET) sensör çipi ve deney boyunca sabit bir voltaj uygulayan bir elektronik okuyucudan oluşur (Şekil 1). Analit, biyosensör yüzeyinde hareketsiz hale getirilmiş hedef proteine çözelti halinde uygulanır. Bir etkileşim meydana geldiğinde, akımdaki bir değişiklik gerçek zamanlı olarak ölçülür ve kaydedilir. Analit konsantrasyonu arttıkça, bağlı analitin fraksiyonu da artacak ve akımda daha yüksek değişimlere neden olacaktır. Cihazla birlikte verilen otomatik analiz yazılımı (Malzeme Tablosu) kullanılarak I-Response, biyolojik algılama birimleri (BU)37 cinsinden ölçülür ve kaydedilir. I-Response, hareketsiz hedefin analit ile etkileşimi üzerine gerçek zamanlı olarak ölçülen biyosensör çipi aracılığıyla akımdaki (I) değişiklik olarak tanımlanır. FEB otomatik analiz yazılımı, C-Yanıtının kapasitanstaki (C) değişiklikleri kaydettiği dinamik etkileşim olaylarına hem I-Yanıtını hem de C-Yanıtını analiz edebilir. Hem I-Response hem de C-Response’deki varyasyonlar doğrudan bağlı analitin fraksiyonuna karşılık gelir ve KD değerleri üretmek için daha fazla analiz edilebilir. Otomatik analiz yazılımının varsayılan tercihi I-Response’dir.
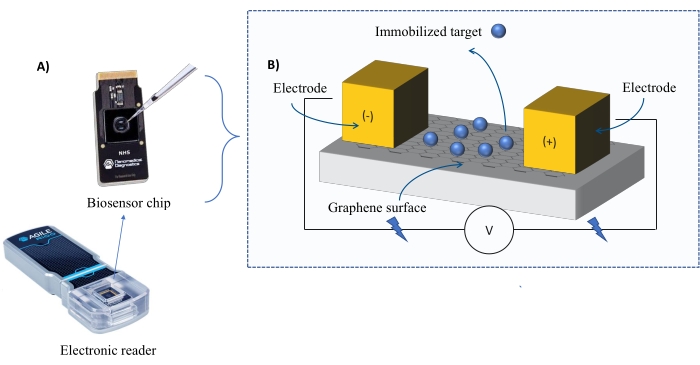
Şekil 1: Deney düzeneğine genel bakış . (A) Grafen esaslı çip ve elektronik okuyucu. (B) Çip bileşenlerine genel bakış. Çip, sisteme akım sağlayan iki elektrota bağlanır. Çipin yüzeyi, etkinleştirildiğinde hedefi bağlayabilen grafen ile kaplıdır. Bu şeklin daha büyük bir versiyonunu görüntülemek için lütfen buraya tıklayın.
Metodoloji:
Başlangıçta, aktif biyosensör çipi FEB cihazına yerleştirilir (Şekil 1) ve ardından aşağıda özetlenen adımların uygulanması: (1) Kalibrasyon: Deney, temel denge tepkisini oluşturmak için 1x fosfat tamponlu salin (PBS; pH = 7.4) kullanılarak sistem kalibrasyonu ile başlar. (2) İlişkilendirme: Analit, çipe verilir ve bağlanma doygunluğuna ulaşılana kadar I-Response izlenir. (3) Ayrışma: Analit, 1x PBS kullanılarak ayrıştırılır. (4) Rejenerasyon: Analitin kalıntıları 1x PBS kullanılarak çıkarılır. (5) Yıkama: Bağlı ve bağlanmamış analitlerin çipten tamamen çıkarılması için 1x PBS kullanılarak toplam beş yıkama yapılır.
Analiz:
Veri analizi, cihazla birlikte verilen tam otomatik yazılım kullanılarak gerçekleştirilir. Otomatik analiz yazılımı, KD değerine sahip bir Hill fit grafiği oluşturur. Hill fit grafiği, bir analitin hedef proteinle ilişkisini analit konsantrasyonlarının bir fonksiyonu olarak tanımlar. Yarım maksimum yanıtın elde edildiği konsantrasyon, KD değeri ile orantılıdır. Düşük bir KD değeri, yüksek bağlanma afinitesini temsil eder ve bunun tersi de geçerlidir.
FEB deneyinden elde edilen verileri doğrulamak için, I-Yanıtları, veri gözden geçirme/dışa aktarma yazılımı kullanılarak her bir analit konsantrasyonu için her okuma noktasından çıkarılır ve aşağıda açıklandığı gibi diğer istatistiksel analiz yazılımlarına (bkz.
Protocol
Representative Results
Discussion
Bu çalışmada, Hsp90 ve Cdc37 arasındaki biyomoleküler etkileşimi belirlemek için FEB teknolojisinin (gerçek zamanlı kinetik karakterizasyon yaklaşımı) kullanılmasının fizibilitesi değerlendirilmiştir. İlk keşif deneyi (ilk deney), uygun analit konsantrasyonlarının seçilmesinin deneyin kritik bir parçası olduğunu ve deneyin, literatürde bulunan verilere dayanarak tahmin edilen KD değerinin üstünde ve altında konsantrasyon noktaları dahil edilerek tasarlanması gerektiğini öne s?…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Bu araştırma, İki Uluslu Bilim Vakfı’ndan (BSF) S.K.S. ve N.Q.’ya verilen bir hibe ile desteklenmiştir.
Materials
| Automated analysis software | Agile plus software, Cardea (Nanomed) | NA CAS number: NA |
Referred to in the text as the automated analysis software supplied with the instrument. Generates automated analysis. |
| COOH-BPU (Biosensing Processing Unit) | Agile plus software, Cardea (Nanomed) | NA CAS number: NA |
biosensor chip |
| Data review software | Datalign 1.0, Cardea (Nanomed) | NA CAS number: NA |
Referred to as the supplied data review software in the text. Supplied with the instrument and allows to review and export the information data points. |
| Dialysis bag | CelluSep, Membrane filtration products | T2-10-15 CAS number: NA |
T2 tubings (6,000-8,000 MWCO), (10 mm fw, 6.4mm Ø, 0.32ml/cm, 15m) |
| EDC (1-Ethyl-3-(3-dimethylamino propyl) carbodiimide) | Cardea (Nanomed) | EDC160322-02 CAS number: 25952-53-8 |
White powder |
| ITC (Isothermal titration calorimetry) system | Microcal-PEAQ-ITC (Malvern, United Kingdom) | NA CAS number: NA |
|
| MES (2-(N-morpholino) ethane sulfonic acid) buffer | Merck | M3671-50G CAS number: 4432-31-9 |
White powder |
| NHS (N-Hydroxysulfosuccinimide) chips | Cardea (Nanomed) | NA CAS number: NA |
Graphene-based chip |
| PBS (Phosphate-buffered saline) X 10 | Bio-Lab | 001623237500 CAS number: 7758-11-4 |
Liquid transparent solution |
| Pipete | Thermo Scientific | 11855231 CAS number: NA |
Finnpipette F3 5-50 µL, yellow |
| Quench 1 (3.9 mM amino-PEG5-alcohol in 1 X PBS) | Cardea (Nanomed) | 0105-001-002-001 CAS number: NA |
Liquid, transparent solution |
| Quench 2 (1 M ethanolamine (pH=8.5)) | Cardea (Nanomed) | 0105-001-003-001 CAS number: NA |
Liquid, transparent solution |
| Recombinant protein Cdc37 | Abcam | ab256157 CAS number: NA |
|
| Recombinant protein Hsp90 beta | Abcam | ab80033 CAS number: NA |
|
| Spreadsheet | Excel, Microsoft office | NA CAS number: NA |
|
| Statistical software | GraphPad, Prism | NA CAS number: NA |
Referred to as the other statistical software. Sigma plot, phyton or other statistical programes may also be used |
| Sulfo-NHS | Cardea (Nanomed) | NHS160321-07 CAS number: 106627-54-7 |
White powder |
| Tips | Alex red | LC 1093-800-000 CAS number: NA |
Tip 1-200 µl, in bulk, 1,000 pcs |
References
- Tuncbag, N., Gursoy, A., Guney, E., Nussinov, R., Keskin, O. Architectures and functional coverage of protein-protein interfaces. Journal of Molecular Biology. 381 (3), 785-802 (2008).
- Berggård, T., Linse, S., James, P. Methods for the detection and analysis of protein–protein interactions. Proteomics. 7 (16), 2833-2842 (2007).
- Magliery, T. J., et al. Detecting protein-protein interactions with a green fluorescent protein fragment reassembly trap: Scope and mechanism. Journal of the American Chemical Society. 127 (1), 146-157 (2005).
- Xing, S., Wallmeroth, N., Berendzen, K. W., Grefen, C. Techniques for the analysis of protein-protein interactions in vivo. Plant Physiology. 171 (2), 727-758 (2016).
- Nguyen, T. N., Goodrich, J. A. Protein-protein interaction assays: Eliminating false positive interactions. Nature Methods. 3 (2), 135-139 (2006).
- Fernández-Suárez, M., Chen, T. S., Ting, A. Y. Protein-protein interaction detection in vitro and in cells by proximity biotinylation. Journal of the American Chemical Society. 130 (29), 9251-9253 (2008).
- Jiang, M., Niu, C., Cao, J., Ni, D. -. A., Chu, Z. In silico-prediction of protein–protein interactions network about MAPKs and PP2Cs reveals a novel docking site variants in Brachypodium distachyon. Scientific Reports. 8 (1), 15083 (2018).
- Yazawa, M., Sadaghiani, A. M., Hsueh, B., Dolmetsch, R. E. Induction of protein-protein interactions in live cells using light. Nature Biotechnology. 27 (10), 941-945 (2009).
- Wang, W., Goodman, M. T. Antioxidant property of dietary phenolic agents in a human LDL-oxidation ex vivo model: Interaction of protein binding activity. Nutrition Research. 19 (2), 191-202 (1999).
- Xing, S., Wallmeroth, N., Berendzen, K. W., Grefen, C. Techniques for the analysis of protein-protein interactions in vivo. Plant Physiology. 171 (2), 727-758 (2016).
- Qvit, N., Disatnik, M. -. H., Sho, E., Mochly-Rosen, D. Selective phosphorylation inhibitor of delta protein kinase C–Pyruvate dehydrogenase kinase protein–protein interactions: Application for myocardial injury in vivo. Journal of the American Chemical Society. 138 (24), 7626-7635 (2016).
- Alam, M. N., Bristi, N. J., Rafiquzzaman, M. Review on in vivo and in vitro methods evaluation of antioxidant activity. Saudi Pharmaceutical Journal. 21 (2), 143-152 (2013).
- Paulmurugan, R., Gambhir, S. S. Novel fusion protein approach for efficient high-throughput screening of small molecule–mediating protein-protein interactions in cells and living animals. Cancer Research. 65 (16), 7413-7420 (2005).
- Boute, N., Jockers, R., Issad, T. The use of resonance energy transfer in high-throughput screening: BRET versus FRET. Trends in Pharmacological Sciences. 23 (8), 351-354 (2002).
- Deriziotis, P., Graham, S. A., Estruch, S. B., Fisher, S. E. Investigating protein-protein interactions in live cells using bioluminescence resonance energy transfer. Journal of Visualized Experiments: JoVE. (87), e51438 (2014).
- Ekins, S., Mestres, J., Testa, B. In silico pharmacology for drug discovery: Methods for virtual ligand screening and profiling. British Journal of Pharmacology. 152 (1), 9-20 (2007).
- Valerio, L. G. Application of advanced in silico methods for predictive modeling and information integration. Expert Opinion on Drug Metabolism & Toxicology. 8 (4), 395-398 (2012).
- Piehler, J. New methodologies for measuring protein interactions in vivo and in vitro. Current Opinion in Structural Biology. 15 (1), 4-14 (2005).
- Ideker, T., Sharan, R. Protein networks in disease. Genome Research. 18 (4), 644-652 (2008).
- Lu, H., et al. Recent advances in the development of protein–protein interactions modulators: mechanisms and clinical trials. Signal Transduction and Targeted Therapy. 5 (1), 213 (2020).
- Jarosz, D. Hsp90: A global regulator of the genotype-to-phenotype map in cancers. Advances in Cancer Research. 129, 225-247 (2016).
- Johnson, V. A., Singh, E. K., Nazarova, L. A., Alexander, L. D., McAlpine, S. R. Macrocyclic inhibitors of Hsp90. Current Topics in Medicinal Chemistry. 10 (14), 1380-1402 (2010).
- Mahalingam, D., et al. Targeting HSP90 for cancer therapy. British Journal of Cancer. 100 (10), 1523-1529 (2009).
- Stewart, A., Fisher, R. A. Co-Immunoprecipitation: Isolation of protein signaling complexes from native tissues. Methods in Cell Biology. 112, 33-54 (2012).
- Pierce, M. M., Raman, C. S., Nall, B. T. Isothermal titration calorimetry of protein-protein interactions. Methods. 19 (2), 213-221 (1999).
- Paketurytė, V., et al. Inhibitor binding to carbonic anhydrases by isothermal titration calorimetry. Carbonic Anhydrase as Drug Target. , 79-95 (2019).
- Grote, J., Dankbar, N., Gedig, E., Koenig, S. Surface plasmon resonance/mass spectrometry interface. Analytical Chemistry. 77 (4), 1157-1162 (2005).
- Kumaraswamy, S., Tobias, R. Label-free kinetic analysis of an antibody–antigen interaction using biolayer interferometry. Methods in Molecular Biology. , 165-182 (2015).
- Wallner, J., Lhota, G., Jeschek, D., Mader, A., Vorauer-Uhl, K. Application of bio-layer interferometry for the analysis of protein/liposome interactions. Journal of Pharmaceutical and Biomedical Analysis. 72, 150-154 (2013).
- Singh, A. N., Ramadan, K., Singh, S. Experimental methods to study the kinetics of protein–protein interactions. Advances in Protein Molecular and Structural Biology Methods. , 115-124 (2022).
- Frenzel, D., Willbold, D. Kinetic titration series with biolayer interferometry. PLoS One. 9 (9), 106882 (2014).
- Vu, C. -. A., Chen, W. -. Y. Field-effect transistor biosensors for biomedical applications: Recent advances and future prospects. Sensors. 19 (19), 4214 (2019).
- Bergveld, P. A critical evaluation of direct electrical protein detection methods. Biosensors & Bioelectronics. 6 (1), 55-72 (1991).
- Lowe, B. M., Sun, K., Zeimpekis, I., Skylaris, C. K., Green, N. G. Field-effect sensors – from pH sensing to biosensing: sensitivity enhancement using streptavidin–biotin as a model system. The Analyst. 142 (22), 4173-4200 (2017).
- Goldsmith, B. R., et al. Digital biosensing by foundry-fabricated graphene sensors. Scientific Reports. 9 (1), 434 (2019).
- Afsahi, S., et al. Novel graphene-based biosensor for early detection of Zika virus infection. Biosensors and Bioelectronics. 100, 85-88 (2018).
- Afsahi, S. J., et al. Towards novel graphene-enabled diagnostic assays with improved signal-to-noise ratio. MRS Advances. 2 (60), 3733-3739 (2017).
- Roe, S. M., et al. The mechanism of Hsp90 regulation by the protein kinase-specific cochaperone p50cdc37. Cell. 116 (1), 87-98 (2004).
- Gaiser, A. M., Kretzschmar, A., Richter, K. Cdc37-Hsp90 complexes are responsive to nucleotide-induced conformational changes and binding of further cofactors. The Journal of Biological Chemistry. 285 (52), 40921-40932 (2010).
- Popescu, A. I., Găzdaru, D. M., Chilom, C. G., Bacalum, M. Biophysical interactions: Their paramount importance for life. Romanian Reports in Physics. 65 (3), 1063-1077 (2013).
- Surya, S., Abhilash, J., Geethanandan, K., Sadasivan, C., Haridas, M. A profile of protein-protein interaction: Crystal structure of a lectin-lectin complex. International Journal of Biological Macromolecules. 87, 529-536 (2016).
- Velazquez-Campoy, A., Freire, E. ITC in the post-genomic era…? Priceless. Biophysical Chemistry. 115 (23), 115-124 (2005).
- Concepcion, J., et al. Label-free detection of biomolecular interactions using bioLayer interferometry for kinetic characterization. Combinatorial Chemistry & High Throughput Screening. 12 (8), 791-800 (2009).
- Helmerhorst, E., Chandler, D. J., Nussio, M., Mamotte, C. D. Real-time and label-free bio-sensing of molecular interactions by surface plasmon resonance: A laboratory medicine perspective. The Clinical Biochemist. Reviews. 33 (4), 161-173 (2012).
- Jacob, N. T., et al. Synthetic molecules for disruption of the MYC protein-protein interface. Bioorganic & Medicinal Chemistry. 26 (14), 4234-4239 (2018).

