Een op massaspectrometrie gebaseerde benadering om fosfoproteïnefosfatasen en hun interactoren te identificeren
Summary
Hier presenteren we een protocol voor de verrijking van endogene fosfoproteïnefosfatasen en hun interagerende eiwitten uit cellen en weefsels en hun identificatie en kwantificering door op massaspectrometrie gebaseerde proteomica.
Abstract
De meeste cellulaire processen worden gereguleerd door dynamische eiwitfosforylering. Meer dan driekwart van de eiwitten is gefosforyleerd en fosfoproteïnefosfatasen (PPP’s) coördineren meer dan 90% van alle cellulaire serine / threonine-defosforylering. Deregulering van eiwitfosforylering is betrokken bij de pathofysiologie van verschillende ziekten, waaronder kanker en neurodegeneratie. Ondanks hun wijdverspreide activiteit zijn de moleculaire mechanismen die PPP’s beheersen en die gecontroleerd door PPP’s slecht gekarakteriseerd. Hier wordt een proteomische benadering beschreven die fosfataseremmerparels en massaspectrometrie (PIB-MS) wordt genoemd om PPP’s, hun posttranslationele modificaties en hun interactoren in slechts 12 uur te identificeren en te kwantificeren met behulp van een cellijn of weefsel. PIB-MS maakt gebruik van een niet-selectieve PPP-remmer, microcystine-LR (MCLR), geïmmobiliseerd op sepharose-kralen om endogene PPP’s en hun bijbehorende eiwitten te vangen en te verrijken (het PPPome genoemd). Deze methode vereist geen exogene expressie van gelabelde versies van PPP’s of het gebruik van specifieke antilichamen. PIB-MS biedt een innovatieve manier om de evolutionair geconserveerde PPP’s te bestuderen en ons huidige begrip van defosforyleringssignalering uit te breiden.
Introduction
Eiwitfosforylering regelt de meeste cellulaire processen, inclusief maar niet beperkt tot de reactie op DNA-schade, groeifactorsignalering en de passage door mitose 1,2,3. In zoogdiercellen worden de meeste eiwitten op een bepaald moment gefosforyleerd op een of meer serine-, threonine- of tyrosineresiduen, waarbij fosfostropenen en fosfoothreonines ongeveer 98% van alle fosforyleringsplaatsen uitmaken 2,3. Hoewel kinasen uitgebreid zijn bestudeerd in cellulaire signalering, is de rol van PPP’s in de regulatie van dynamische cellulaire processen nog steeds in opkomst.
Fosforyleringsdynamiek wordt gecontroleerd door het dynamische samenspel tussen kinasen en fosfatasen. In zoogdiercellen zijn er meer dan 400 eiwitkinasen die serine / threoninefosforylering katalyseren. Meer dan 90% van deze sites wordt gedefosforyleerd door fosfoproteïnefosfatasen (PPP’s), een kleine familie van enzymen die bestaat uit PP1, PP2A, PP2B, PP4-7, PPT en PPZ 2,3. PP1 en PP2A zijn verantwoordelijk voor de meerderheid van fosfosforrine en fosfotheonine defosforylering in een cel 2,3,4. Het opmerkelijke verschil in aantal tussen kinasen en fosfatasen en het gebrek aan specificiteit van PPP katalytische subeenheden in vitro leidden tot de overtuiging dat kinasen de belangrijkste determinant zijn van fosforylering 2,3. Meerdere studies hebben echter aangetoond dat fosfatasen substraatspecificiteit vaststellen door de vorming van multimere holo-enzymen 5,6,7,8,9. PP1 is bijvoorbeeld een heterodimeer dat bestaat uit een katalytische subeenheid en op een gegeven moment een van de meer dan 150 regulerende subeenheden 6,7,8. Omgekeerd is PP2A een heterotrimeer dat wordt gevormd uit een steiger (A), een regulerende (B) en een katalytische (C) subeenheid 2,3,9. Er zijn vier verschillende families van PP2A regulerende subeenheden (B55, B56, PR72 en striatine), elk met meerdere genen, splicevarianten en lokalisatiepatronen 2,3,9. Het multimere karakter van PPP’s vult de leemte in het aantal kinasen en PPP-katalytische subeenheden. Het creëert echter analytische uitdagingen voor het bestuderen van PPP-signalering. Om PPP-signalering uitgebreid te analyseren, is het van cruciaal belang om de verschillende holo-enzymen in een cel of weefsel te onderzoeken. Er is grote vooruitgang geboekt bij het bestuderen van het menselijke kinoom door het gebruik van kinaseremmerparels, multiplexremmerparels of kinobeads genoemd, een chemische proteomische strategie waarbij kinaseremmers worden geïmmobiliseerd op kralen en massaspectrometrie wordt gebruikt om verrijkte kinasen en hun interactoren te identificeren 10,11,12,13.
We hebben een vergelijkbare aanpak vastgesteld om PPP-biologie te bestuderen. Deze techniek omvat affiniteitsvangst van PPP-katalytische subeenheden met behulp van kralen met een geïmmobiliseerde, niet-selectieve PPP-remmer genaamd microcystine-LR (MCLR) genaamd fosfataseremmerparels (PIBs)14,15. In tegenstelling tot andere methoden die de endogene tagging of expressie van exogene PPP-subeenheden vereisen die de eiwitactiviteit of lokalisatie kunnen veranderen, maakt PIB-MS de verrijking mogelijk van endogene PPP-katalytische subeenheden, hun bijbehorende regulerende en steigerende subeenheden en interagerende eiwitten (het PPPome genoemd) uit cellen en weefsels op een bepaald tijdstip of onder specifieke behandelingsomstandigheden. MCLR remt PP1, PP2A, PP4-6, PPT en PPZ bij nanomolaire concentraties, waardoor PIB’s zeer effectief zijn in het verrijken voor de PPPome16. Deze methode kan worden geschaald voor gebruik op elk uitgangsmateriaal, van cellen tot klinische monsters. Hier beschrijven we in detail het gebruik van PIB’s en massaspectrometrie (PIB-MS) om het endogene PPPome en de modificatietoestanden efficiënt vast te leggen, te identificeren en te kwantificeren.
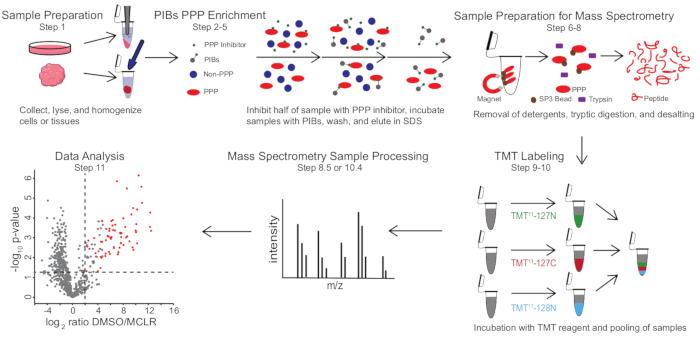
Figuur 1: Visuele samenvatting van het PIB-MS protocol. In een PIB-MS-experiment kunnen monsters worden verkregen in verschillende vormen, van cellen tot tumoren. Het monster wordt verzameld, gelyseerd en gehomogeniseerd voorafgaand aan PPP-verrijking. Ter verrijking voor PPP’s wordt het lysaat geïncubeerd met PIB’s met of zonder PPP-remmer, zoals MCLR. De PIB’s worden vervolgens gewassen en PPP’s worden geëlueerd in denatureringsomstandigheden. De monsters worden voorbereid voor massaspectrometrie-analyse door de verwijdering van detergentia door sp3-eiwitverrijking, tryptische vertering en ontzouting. Monsters kunnen dan optioneel TMT-gelabeld worden voorafgaand aan massaspectrometrie analyse. Klik hier om een grotere versie van deze figuur te bekijken.
PIB-MS omvat lysis en klaring van cellen of weefsels, incubatie van het lysaat met PIBs, elutie en analyse van het eluaat via western blotting of op massaspectrometrie gebaseerde benaderingen (figuur 1). De toevoeging van vrije MCLR kan worden gebruikt als een controle om specifieke PIB-bindmiddelen te onderscheiden van niet-specifieke interactoren. Voor de meeste toepassingen kan een labelvrije aanpak worden gebruikt om eiwitten in eluaten direct te identificeren. In gevallen waarin een grotere precisie in kwantificering of de identificatie van soorten met een lage abundantie nodig is, kan verdere verwerking met tandem mass-tag (TMT) -etikettering worden gebruikt om de dekking te vergroten en de input te verminderen.
Protocol
Representative Results
Discussion
PIB-MS is een chemische proteomics-benadering die wordt gebruikt om het PPPome kwantitatief te profileren uit verschillende monsterbronnen in één analyse. Er is veel werk verricht met behulp van kinaseremmerkralen om het kinoom te bestuderen en hoe het verandert in kanker en andere ziektetoestanden 10,11,12,13. Toch blijft de studie van de PPPome achter. We verwachten dat deze aanpak in staat…
Disclosures
The authors have nothing to disclose.
Acknowledgements
A.N.K. erkent de steun van NIH R33 CA225458 en R35 GM119455. Wij danken de laboratoria van Kettenbach en Gerber voor hun nuttige discussie.
Materials
| Acetonitrile (ACN) | Honeywell | AH015-4 | CAUTION: ACN is flammable and toxic; wear gloves, and work in a chemical fume hood. |
| Anhydrous Acetonitrile | Sigma-Aldrich | 271004-100ML | CAUTION: ACN is flammable and toxic; wear gloves, and work in a chemical fume hood. |
| Benchtop centrifuge | Eppendorf | model no. 5424 | |
| Beta-glycerophosphoric acid, disodium salt pentahydrate | Acros Organics | 410991000 | |
| Centrifuge | Eppendorf | model no. 5810 R 15 amp version | |
| Distilled water | |||
| DMSO | Fisher Scientific | BP231-100 | |
| Dounce tissue grinder | Fisherbrand Pellet Pestles | 12-141-363 | |
| Empore solid phase extraction disk, C18 | CDS Analytical | 76333-132 | |
| Eppendorf tubes, 1.5 mL | Eppendorf | 22363204 | CRITICAL: Other tubes may leach polymer into sample, contaminating the analysis. |
| Eppendorf tubes, 2 mL | Eppendorf | 22363352 | CRITICAL: Other tubes may leach polymer into sample, contaminating the analysis. |
| Extraction plate manifold | Waters | WAT097944 | |
| Falcon tubes, 50 mL | VWR | 21008 | |
| Generic blunt end needle and plunger | |||
| Generic magnetic separation rack | |||
| HEPES | Sigma-Aldrich | H3375 | |
| Hydrogen chloride (HCl) | VWR Chemicals BDH | BDH3028 | CAUTION: HCl is corrosive; wear gloves and work in a chemical fume hood. |
| Hydroxylamine solution 50% (wt/vol) | Sigma-Aldrich | 467804 | |
| Incubator, 65 °C | VWR | model no. 1380FM | |
| Koptec Pure Ethanol, 200 Proof | Decon Labs | V1001 | |
| Methanol for HPLC (MeOH) | Sigma-Aldrich | 34860-4L-R | CAUTION: MeOH is flammable and toxic; wear gloves, and work in a chemical fume hood. |
| Microcystin LR (MCLR) | Cayman Chemical | 10007188 | CAUTION: MCLR is toxic; wear gloves when handling and avoid skin contact. |
| PBS, 1× without calcium and magnesium, pH 7.4 ± 0.1 | Corning | 21-040-CV | |
| pH test strips, such as MilliporeSigma MColorpHast pH test strips and indicator papers | Fisher Scientific | M1095310001 | |
| PIBs | For protocol for the generation of PIBs, see Moorhead et al., 2007. | ||
| Pierce BCA Protein Assay Kit | Thermo Scientific | 23225 | |
| Pipette tips, 10 μL | Eppendorf | 22491504 | CRITICAL: Other tips may leach polymer into samples, contaminating the analysis. |
| Pipette tips, 1000 μL | Eppendorf | 22491555 | CRITICAL: Other tips may leach polymer into samples, contaminating the analysis. |
| Pipette tips, 200 μL | Eppendorf | 22491539 | CRITICAL: Other tips may leach polymer into samples, contaminating the analysis. |
| plastic syringe, 10 mL | BD | 309604 | |
| Protease inhibitor cocktail III | Research Products International | P50700-1 | |
| Q Exactive Plus Hybrid Quadrupole-Orbitrap Mass Spectrometer, Oribtrap Fusion, Orbitrap Fusion Lumos, or Orbitrap Eclipse Tribrid Mass Spectrometer | Thermo Scientific | ||
| Refrigerated benchtop centrifuge | Eppendorf | model no. 5424 R | |
| Rotator (Labquake Shaker Rotisserie) | Thermo Scientific | 13-687-12Q | 8 rpm rotation |
| Sample collection plate, 96- well, 1 mL | Waters | WAT058957 | |
| SDS | Fisher Scientific | BP1311-1 | |
| Sequencing grade modified trypsin | Promega | V511C | |
| Sodium azide | EMD Chemicals | SX0299-1 | CAUTION: Sodium azide is explosive and toxic; wear gloves, work in a chemical fume hood and avoid contact with metals. |
| Sodium chloride (NaCl) | Fisher Chemical | S27110 | |
| Sonicator (Branson digital sonifier) | model no. SFX 250 | ||
| SPE C18 desalting plate | Waters | 186001828BA | |
| SpeedBeads magnetic carboxylate modified particles (SP3 beads) | Cytiva | 6.51521E+13 | |
| Thermomixer | Eppendorf | model no. 5350 | |
| TMT10plex Isobaric Label Reagent Set plus TMT11-131C Label Reagent, 3 × 0.8 mg per tag | ThermoFisher | A37725 | |
| Trifluoroacetic acid (TFA) | Honeywell | T6508-25ML | CAUTION: TFA is corrosive and will irritate skin on contact. Wear gloves and eye protection, and work in a chemical fume hood. |
| Tris Base | Research Products International | T60040 | |
| Triton X-100 | Sigma-Aldrich | T9284 | |
| Vacuum centrifuge and vapor trap | Thermo Scientific | model nos. SpeedVac SPD120 and RVT5105 | |
| Vortexer (Vortex-Genie 2) | Scientific Industries | ||
| Water LC-MS | Honeywell | LC365-4 |
References
- Nilsson, J. Protein phosphatases in the regulation of mitosis. Journal of Cell Biology. 218 (2), 395-409 (2019).
- Brautigan, D. L. Protein Ser/Thr phosphatases–the ugly ducklings of cell signalling. The FEBS Journal. 280 (2), 324-345 (2013).
- Brautigan, D. L., Shenolikar, S. Protein serine/threonine phosphatases: keys to unlocking regulators and substrates. Annual Review of Biochemistry. 87, 921-964 (2018).
- Janssens, V., Goris, J. Protein phosphatase 2A: A highly regulated family of serine/threonine phosphatases implicated in cell growth and signalling. Biochemical Journal. 353, 417-439 (2001).
- Virshup, D. M., Shenolikar, S. From promiscuity to precision: protein phosphatases get a makeover. Molecular Cell. 33 (5), 537-545 (2009).
- Bollen, M., Peti, W., Ragusa, M. J., Beullens, M. The extended PP1 toolkit: designed to create specificity. Trends in Biochemical Sciences. 35 (8), 450-458 (2010).
- Qian, J., Winkler, C., Bollen, M. 4D-networking by mitotic phosphatases. Current Opinion in Cell Biology. 25 (6), 697-703 (2013).
- Heroes, E., et al. The PP1 binding code: a molecular-lego strategy that governs specificity. The FEBS Journal. 280 (2), 584-595 (2013).
- Eichhorn, P. J., Creyghton, M. P., Bernards, R. Protein phosphatase 2A regulatory subunits and cancer. Biochimica et Biophysica Acta. 1795 (1), 1-15 (2009).
- Bantscheff, M., et al. Quantitative chemical proteomics reveals mechanisms of action of clinical ABL kinase inhibitors. Nature Biotechnology. 25 (9), 1035-1044 (2007).
- Klaeger, S., et al. Chemical proteomics reveals ferrochelatase as a common off-target of kinase inhibitors. ACS Chemical Biology. 11 (5), 1245-1254 (2016).
- Duncan, J. S., et al. Dynamic reprogramming of the kinome in response to targeted MEK inhibition in triple-negative breast cancer. Cell. 149 (2), 307-321 (2012).
- Cooper, M. J., et al. Application of multiplexed kinase inhibitor beads to study kinome adaptations in drug-resistant leukemia. PLoS ONE. 8 (6), 66755 (2013).
- Lyons, S. P., et al. A quantitative chemical proteomic strategy for profiling phosphoprotein phosphatases from yeast to humans. Molecular and Cellular Proteomics. 17 (12), 2448-2461 (2018).
- Nasa, I., et al. Quantitative kinase and phosphatase profiling reveal that CDK1 phosphorylates PP2Ac to promote mitotic entry. Science Signaling. 13 (648), (2020).
- Swingle, M., Ni, L., Honkanen, R. E. Small-molecule inhibitors of ser/thr protein phosphatases: specificity, use and common forms of abuse. Methods in Molecular Biology. 365, 23-38 (2007).
- Moorhead, G. B. G., Haystead, T. A. J., MacKintosh, C. Synthesis and use of the protein phosphatase affinity matrices microcystin-sepharose and microcystin-biotin-sepharose. Methods in Molecular Biology. 365, 39-45 (2007).
- Brauer, B. L., Wiredu, K., Mitchell, S., Moorhead, G. B., Gerber, S. A., Kettenbach, A. N. Affinity-based profiling of endogenous phosphoprotein phosphatases by mass spectrometry. Nature Protocols. 16 (10), 4919-4943 (2021).
- Hughes, C. S., Moggridge, S., Müller, T., Sorensen, P. H., Morin, G. B., Krijgsveld, J. Single-pot, solid-phase-enhanced sample preparation for proteomics experiments. Nature Protocols. 14 (1), 68-85 (2019).
- Rappsilber, J., Mann, M., Ishihama, Y. Protocol for micro-purification, enrichment, pre-fractionation and storage of peptides for proteomics using StageTips. Nature Protocols. 2 (8), 1896-1906 (2007).
- Zecha, J., et al. TMT labeling for the masses: A robust and cost-efficient, in-solution labeling approach. Molecular and Cellular Proteomics. 18 (7), 1468-1478 (2019).
- Elias, J. E., Gygi, S. P. Target-decoy search strategy for increased confidence in large-scale protein identifications by mass spectrometry. Nature Methods. 4 (3), 207-214 (2007).
- Eng, J. K., Jahan, T. A., Hoopmann, M. R. Comet: an open-source MS/MS sequence database search tool. Proteomics. 13 (1), 22-24 (2013).
- Tyanova, S., et al. The Perseus computational platform for comprehensive analysis of (prote)omics data. Nature Methods. 13 (9), 731-740 (2016).
- Yu, S. H., Ferretti, D., Schessner, J. P., Rudolph, J. D., Borner, G. H. H., Cox, J. Expanding the Perseus software for omics data analysis With custom plugins. Current Protocols in Bioinformatics. 71 (1), 1-29 (2020).

