Criblage à haut débit d’isolats microbiens ayant un impact sur la santé de Caenorhabditis elegans
Summary
Les microbes intestinaux peuvent avoir un impact positif ou négatif sur la santé de leur hôte via des mécanismes spécifiques ou conservés. Caenorhabditis elegans est une plate-forme pratique pour dépister de tels microbes. Le présent protocole décrit le dépistage à haut débit de 48 isolats bactériens pour déterminer leur impact sur la résistance au stress des nématodes, utilisés comme indicateur de la santé des vers.
Abstract
Avec sa petite taille, sa courte durée de vie et sa génétique facile, Caenorhabditis elegans offre une plate-forme pratique pour étudier l’impact des isolats microbiens sur la physiologie de l’hôte. Il devient également fluorescent en bleu lors de la mort, fournissant un moyen pratique de localiser la mort. Cette propriété a été exploitée pour développer des tests de survie de C. elegans sans marquage à haut débit (LFASS). Celles-ci impliquent un enregistrement de fluorescence en accéléré des populations de vers placées dans des plaques multipuits, à partir desquelles le temps médian de mort de la population peut être déduit. La présente étude adopte l’approche LFASS pour dépister simultanément plusieurs isolats microbiens afin de déterminer les effets sur la sensibilité de C. elegans à la chaleur sévère et aux stress oxydatifs. Un tel pipeline de dépistage microbien, qui peut notamment être utilisé pour présélectionner les probiotiques, en utilisant une résistance sévère au stress comme indicateur de la santé de l’hôte, est rapporté ici. Le protocole décrit comment cultiver à la fois des collections d’isolats de microbiote intestinal de C. elegans et des populations de vers synchrones dans des réseaux multipuits avant de les combiner pour les essais. L’exemple fourni couvre l’analyse de 47 isolats bactériens et d’une souche témoin sur deux souches de vers, dans deux essais de stress en parallèle. Cependant, le pipeline d’approche est facilement évolutif et applicable à l’examen de nombreuses autres modalités. Ainsi, il fournit une configuration polyvalente pour étudier rapidement un paysage multiparamétrique de conditions biologiques et biochimiques qui ont un impact sur la santé de C. elegans.
Introduction
Le corps humain abrite environ 10 à 100 billions de cellules microbiennes vivantes (bactéries, champignons archées), qui se trouvent principalement dans les environnements intestinaux, cutanés et muqueux1. Dans un état sain, ceux-ci offrent des avantages à leur hôte, y compris la production de vitamines, la maturation du système immunitaire, la stimulation des réponses immunitaires innées et adaptatives aux agents pathogènes, la régulation du métabolisme des graisses, la modulation des réponses au stress, etc., avec un impact sur la croissance et le développement, l’apparition de la maladie et le vieillissement 2,3,4,5 . Le microbiote intestinal évolue également considérablement tout au long de la vie. L’évolution la plus radicale se produit pendant la petite enfance et la petite enfance6, mais des changements significatifs se produisent également avec l’âge, notamment une diminution de l’abondance de Bifidobacterium et une augmentation des espèces de Clostridium, Lactobacillus, Enterobacteriaceae et Enterococcus 7. Le mode de vie peut altérer davantage la composition microbienne intestinale conduisant à une dysbiose (perte de bactéries bénéfiques, prolifération de bactéries opportunistes), entraînant diverses pathologies telles que les maladies inflammatoires de l’intestin, le diabète et l’obésité5, mais contribuant également aux maladies d’Alzheimer et de Parkinson 8,9,10,11.
Cette prise de conscience a contribué de manière critique à affiner le concept de l’axe intestin-cerveau (ACS), où les interactions entre la physiologie intestinale (y compris maintenant les microbes qu’elle contient) et le système nerveux sont considérées comme le principal régulateur du métabolisme animal et des fonctions physiologiques12. Cependant, le rôle précis du microbiote dans la signalisation intestin-cerveau et les mécanismes d’action associés sont loin d’être entièrement compris13. Le microbiote intestinal étant un déterminant clé du vieillissement en bonne santé, la façon dont les bactéries modulent le processus de vieillissement est devenue un sujet de recherche intense et de controverse 6,14,15.
Avec la démonstration que le ver rond Caenorhabditis elegans héberge un véritable microbiote intestinal dominé – comme chez d’autres espèces – par Bacteroidetes, Firmicutes, et Actinobacteria 16,17,18,19,20, son ascension rapide en tant que plate-forme expérimentale pour étudier les interactions commensales hôte-intestin21,22,23,24 ,25,26 a considérablement élargi notre arsenal d’enquête26,27,28,29. En particulier, les approches expérimentales à haut débit disponibles pour C. elegans pour étudier les interactions gène-alimentation, gène-médicament, gène-pathogène, etc., peuvent être adaptées pour explorer rapidement l’impact des isolats et des cocktails bactériens sur la santé et le vieillissement de C. elegans.
Le présent protocole décrit un pipeline expérimental permettant de dépister immédiatement des réseaux d’isolats ou de mélanges bactériens placés dans des plaques multipuits pour déterminer les effets sur la résistance au stress de C. elegans en tant qu’indicateur de la santé, ce qui peut être utilisé pour identifier les probiotiques. Il explique comment développer de grandes populations de vers et manipuler des réseaux bactériens dans des formats de plaques à 96 et 384 puits avant de traiter les vers pour une analyse automatisée de la résistance au stress à l’aide d’un lecteur de plaque de fluorescence (Figure 1). L’approche est basée sur des tests de survie automatisés sans marquage (LFASS)30 qui exploitent le phénomène de fluorescence de mort31, par lequel les vers mourants produisent une explosion de fluorescence bleue qui peut être utilisée pour déterminer le moment de la mort. La fluorescence bleue est émise par les esters glucosyles de l’acide anthranilique stockés dans les granules intestinaux de C. elegans (un type d’organite apparenté au lysosome), qui éclatent lorsqu’une cascade nécrotique est déclenchée dans l’intestin du ver à la mort31.
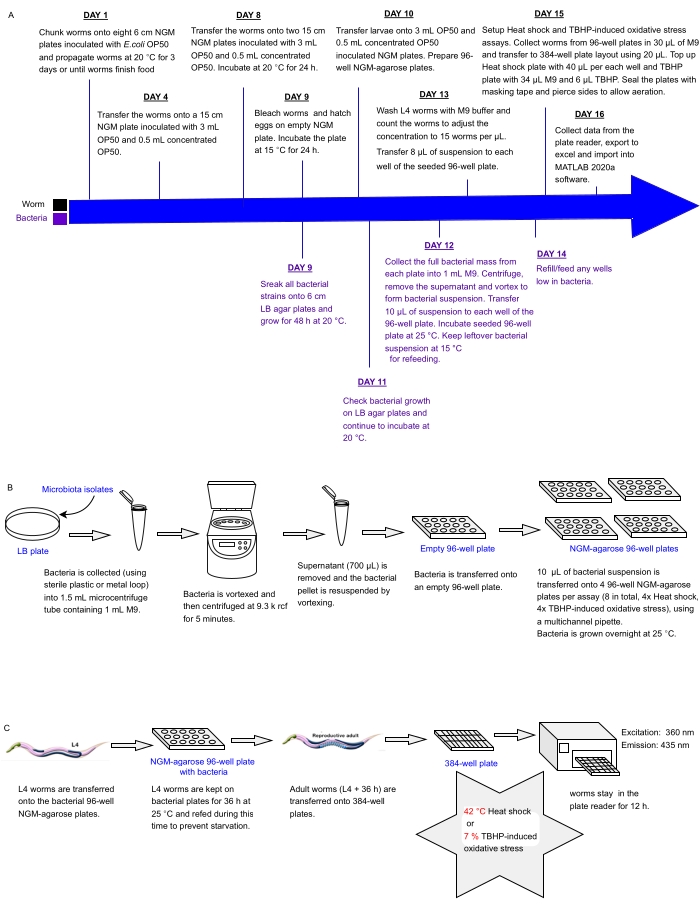
Figure 1 : Flux de travail expérimental pour le criblage à haut débit d’isolats bactériens ayant un impact sur la résistance de C. elegans au stress. (A) Calendrier pour l’entretien et la mise en place des tests vers et bactériens. (B) Installation et manipulation de réseaux de plaques bactériennes à 96 puits. (C) Installation de plaques à vis sans fin à 384 puits. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Protocol
Representative Results
Discussion
C. elegans offre de nombreux avantages pour le criblage rapide de plusieurs paramètres expérimentaux à la fois, en raison de sa petite taille, de sa transparence, de son développement rapide, de sa courte durée de vie, de son faible coût et de sa facilité de manipulation. Son génome, son plan corporel, son système nerveux, son intestin et son microbiome considérablement plus simples, mais suffisamment complexes et similaires à ceux des humains, en font un modèle préclinique puissant, où des informa…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Nous remercions la CCG Minnesota (Madison, États-Unis, NIH – P40 OD010440) pour avoir fourni des souches de vers et OP50 et le Pr. Hinrich Schulenburg (CAU, Kiel, Allemagne) pour avoir fourni tous les isolats microbiens environnementaux représentés ici. Ce travail a été financé par une subvention UKRI-BBSRC à AB (BB/S017127/1). JM est financé par une bourse de doctorat FHM de l’Université de Lancaster.
Materials
| 10 cm diameter plates (Non-vented) | Fisher Scientific | 10720052 | Venting is not necessary for bacterial cultures |
| 15 cm diameter plates (Vented) | Fisher Scientific | 168381 | |
| 384-well black, transparent flat bottom plates | Corning | 3712 or 3762 | Not essential to be sterile for fast stress assays |
| 6 cm diameter plates (Vented) | Fisher Scientific | 150288 | Venting is necessary for worm cultures to avoid hypoxia |
| 96-well transparent plates (Biolite) | Thermo | 130188 | |
| Agar (<4% ash) | Sigma-Aldrich | 102218041 | Good quality agar is important for the structural integrity of the culture media, to avoid worm burrowing |
| Agarose | Fisher Scientific | BP1356 | |
| Avanti Centrifuge J-26 XP | Beckman coulter | ||
| Bleach | Honeywell | 425044 | |
| Calcium chloride | Sigma-Aldrich | C5080 | |
| Centrifuge 5415 R | Eppendorf | ||
| Centrifuge 5810 R | Eppendorf | ||
| Cholesterol | Sigma-Aldrich | C8667 | |
| LB agar | Difco | 240110 | |
| LB broth | Invitrogen | 12795084 | |
| LoBind tips | VWR | 732-1488 | Lo-bind reduce worm loss during transfers |
| LoBind tubes | Eppendorf | 22431081 | |
| Magnesium sulfate | Fisher Scientific | M/1100/53 | |
| Plate reader- infinite M nano+ | Tecan | Monochromator setup enables fluorescence tuning but adequate filter-based setups may be used | |
| Plate reader- Spark | Tecan | ||
| Potassium phosphate monobasic | Honeywell | P0662 | |
| Sodium chloride | Sigma-Aldrich | S/3160/63 | |
| Stereomicroscope setup with transillumination base | Leica | MZ6, or M80 | Magnification from 0.6-0.8x up to 40-60x is necessary, as is a good quality transillumination base with a deformable, titable or slidable mirror to adjust contrast |
| t-BHP (tert-Butyl hydroperoxide) | Sigma-Aldrich | 458139 | |
| Transparent adhesive seals Nunc | Fisher Scientific | 101706871 | It is important that it is transparent and that it can tolerate the temperatures involved in the assays. |
| Tryptophan | Sigma-Aldrich | 1278-7099 | |
| Yeast extract | Fisher Scientific | BP1422 |
References
- Krishna, S., et al. Integrating microbiome network: establishing linkages between plants, microbes and human health. The Open Microbiology Journal. 13, 330-342 (2019).
- Amon, P., Sanderson, I. What is the microbiome. Archives of Disease in Childhood – Education & Practice Edition. 102 (5), 257-260 (2017).
- Belkaid, Y., Harrison, O. J. Homeostatic immunity and the microbiota. Immunity. 46 (4), 562-576 (2017).
- Cabreiro, F., Gems, D. Worms need microbes too: microbiota, health and aging in Caenorhabditis elegans. EMBO Molecular Medicine. 5 (9), 1300-1310 (2013).
- Vaga, S., et al. Compositional and functional differences of the mucosal microbiota along the intestine of healthy individuals. Scientific Reports. 10 (1), 14977 (2020).
- Nagpal, R., et al. Gut microbiome and aging: Physiological and mechanistic insights. Nutrition and Healthy Aging. 4 (4), 267-285 (2018).
- Mitsuoka, T. Establishment of intestinal bacteriology. Biosci Microbiota Food Health. 33 (3), 99-116 (2014).
- Bonfili, L., et al. Microbiota modulation as preventative and therapeutic approach in Alzheimer’s disease. The FEBS Journal. 288 (9), 2836-2855 (2021).
- Vendrik, K. E. W., et al. Fecal microbiota transplantation in neurological disorders. Frontiers in Cellular and Infection Microbiology. 10, 98 (2020).
- Wang, Q., et al. The role of gut dysbiosis in Parkinson’s disease: mechanistic insights and therapeutic options. Brain. 144 (9), 2571-2593 (2021).
- Zhu, X., et al. The relationship between the gut microbiome and neurodegenerative diseases. Neuroscience Bulletin. 37 (10), 1510-1522 (2021).
- Miller, I. The gut-brain axis: historical reflections. Microbial Ecology in Health and Disease. 29 (1), 1542921 (2018).
- Foster, J. A., Rinaman, L., Cryan, J. F. Stress & the gut-brain axis: Regulation by the microbiome. Neurobiology of Stress. 7, 124-136 (2017).
- Coman, V., Vodnar, D. C. Gut microbiota and old age: Modulating factors and interventions for healthy longevity. Experimental Gerontology. 141, 111095 (2020).
- Conway, J., Duggal, N. A. Ageing of the gut microbiome: Potential influences on immune senescence and inflammageing. Ageing Research Reviews. 68, 101323 (2021).
- Berg, M., et al. Assembly of the Caenorhabditis elegans gut microbiota from diverse soil microbial environments. The ISME Journal. 10 (8), 1998-2009 (2016).
- Dirksen, P., et al. CeMbio – The Caenorhabditis elegans Microbiome Resource. G3: Genes, Genomes, Genetics. 10 (9), 3025-3039 (2020).
- Dirksen, P., et al. The native microbiome of the nematode Caenorhabditis elegans: gateway to a new host-microbiome model. BMC Biology. 14, 38 (2016).
- Samuel, B. S., Rowedder, H., Braendle, C., Felix, M. A., Ruvkun, G. Caenorhabditis elegans responses to bacteria from its natural habitats. Proceedings of the National Academy of Sciences of the United States of America. 113 (27), 3941-3949 (2016).
- Zimmermann, J., et al. The functional repertoire contained within the native microbiota of the model nematode Caenorhabditis elegans. The ISME Journal. 14 (1), 26-38 (2020).
- Dinic, M., et al. Host-commensal interaction promotes health and lifespan in Caenorhabditis elegans through the activation of HLH-30/TFEB-mediated autophagy. Aging. 13 (6), 8040-8054 (2021).
- Goya, M. E., et al. Probiotic Bacillus subtilis protects against alpha-Synuclein aggregation in C. elegans. Cell Reports. 30 (2), 367-380 (2020).
- Hacariz, O., Viau, C., Karimian, F., Xia, J. The symbiotic relationship between Caenorhabditis elegans and members of its microbiome contributes to worm fitness and lifespan extension. BMC Genomics. 22 (1), 364 (2021).
- Shin, M. G., et al. Bacteria-derived metabolite, methylglyoxal, modulates the longevity of C. elegans through TORC2/SGK-1/DAF-16 signaling. Proceedings of the National Academy of Sciences of the United States of America. 117 (29), 17142-17150 (2020).
- Zhang, F., et al. Natural genetic variation drives microbiome selection in the Caenorhabditis elegans gut. Current Biology. 31 (12), 2603-2618 (2021).
- Zhang, F., et al. High-throughput assessment of changes in the Caenorhabditis elegans gut microbiome. Methods in Molecular Biology. 2144, 131-144 (2020).
- Chan, J. P., et al. Using bacterial transcriptomics to investigate targets of host-bacterial interactions in Caenorhabditis elegans. Scientific Reports. 9 (1), 5545 (2019).
- Hartsough, L. A., et al. Optogenetic control of gut bacterial metabolism to promote longevity. Elife. 9, 56849 (2020).
- Pryor, R., et al. Host-microbe-drug-nutrient screen identifies bacterial effectors of Metformin therapy. Cell. 178 (6), 1299-1312 (2019).
- Benedetto, A., et al. New label-free automated survival assays reveal unexpected stress resistance patterns during C. elegans aging. Aging Cell. 18 (5), 12998 (2019).
- Coburn, C., et al. Anthranilate fluorescence marks a calcium-propagated necrotic wave that promotes organismal death in C. elegans. PLOS Biology. 11 (7), 1001613 (2013).
- Porta-de-la-Riva, M., Fontrodona, L., Villanueva, A., Ceron, J. Basic Caenorhabditis elegans methods: synchronization and observation. Journal of Visualized Experiments. (64), e4019 (2012).
- Stiernagle, T. Maintenance of C. elegans. WormBook. , 1-11 (2006).
- Naomi, R., et al. Probiotics for Alzheimer’s disease: a systematic review. Nutrients. 14 (1), 20 (2021).
- Zheng, S. Y., et al. Potential roles of gut microbiota and microbial metabolites in Parkinson’s disease. Ageing Research Reviews. 69, 101347 (2021).
- Gill, M. S., Olsen, A., Sampayo, J. N., Lithgow, G. J. An automated high-throughput assay for survival of the nematode Caenorhabditis elegans. Free Radical Biology and Medicine. 35 (6), 558-565 (2003).
- Park, H. -. E. H., Jung, Y., Lee, S. -. J. V. Survival assays using Caenorhabditis elegans. Molecules and Cells. 40 (2), 90-99 (2017).
- Partridge, F. A., et al. An automated high-throughput system for phenotypic screening of chemical libraries on C. elegans and parasitic nematodes. International Journal for Parasitology: Drugs and Drug Resistance. 8 (1), 8-21 (2018).
- Rahman, M., et al. NemaLife chip: a micropillar-based microfluidic culture device optimized for aging studies in crawling C. elegans. Scientific Reports. 10 (1), 16190 (2020).
- Stroustrup, N., et al. The Caenorhabditis elegans lifespan machine. Nature Methods. 10 (7), 665-670 (2013).
- Xian, B., et al. WormFarm: a quantitative control and measurement device toward automated Caenorhabditis elegans aging analysis. Aging Cell. 12 (3), 398-409 (2013).
- Brown, A. E., Schafer, W. R. Unrestrained worms bridled by the light. Nature Methods. 8 (2), 129-130 (2011).
- Churgin, M. A., et al. Longitudinal imaging of Caenorhabditis elegans in a microfabricated device reveals variation in behavioral decline during aging. Elife. 6, 26652 (2017).
- Jushaj, A., et al. Optimized criteria for locomotion-based healthspan evaluation in C. elegans using the WorMotel system. PLoS One. 15 (3), 0229583 (2020).
- Nambyiah, P., Brown, A. E. X. Quantitative behavioural phenotyping to investigate anaesthesia induced neurobehavioural impairment. Scientific Reports. 11 (1), 19398 (2021).
- Squiban, B., Belougne, J., Ewbank, J., Zugasti, O. Quantitative and automated high-throughput genome-wide RNAi screens in C. elegans. Journal of Visualized Experiments. (60), e3448 (2012).
- Zugasti, O., et al. Activation of a G protein-coupled receptor by its endogenous ligand triggers the innate immune response of Caenorhabditis elegans. Nature Immunology. 15 (9), 833-838 (2014).
- Zugasti, O., et al. A quantitative genome-wide RNAi screen in C. elegans for antifungal innate immunity genes. BMC Biology. 14, 35 (2016).

