Screening ad alta produttività di isolati microbici con impatto sulla salute di Caenorhabditis elegans
Summary
I microbi intestinali possono avere un impatto positivo o negativo sulla salute del loro ospite attraverso meccanismi specifici o conservati. Caenorhabditis elegans è una comoda piattaforma per lo screening di tali microbi. Il presente protocollo descrive lo screening ad alto rendimento di 48 isolati batterici per l’impatto sulla resistenza allo stress dei nematodi, utilizzati come proxy per la salute dei vermi.
Abstract
Con le sue piccole dimensioni, la breve durata della vita e la facile genetica, Caenorhabditis elegans offre una comoda piattaforma per studiare l’impatto degli isolati microbici sulla fisiologia dell’ospite. Fluorescente anche in blu quando muore, fornendo un mezzo conveniente per individuare la morte. Questa proprietà è stata sfruttata per sviluppare saggi di sopravvivenza di C. elegans (LFASS) label-free ad alto rendimento. Questi comportano la registrazione della fluorescenza time-lapse delle popolazioni di vermi impostate in piastre multipozzetto, da cui può essere derivato il tempo mediano di morte della popolazione. Il presente studio adotta l’approccio LFASS per lo screening di più isolati microbici contemporaneamente per gli effetti sulla suscettibilità di C. elegans al calore severo e agli stress ossidativi. Tale pipeline di screening microbico, che può essere utilizzata in particolare per preselezionare i probiotici, utilizzando una grave resistenza allo stress come proxy per la salute dell’ospite è riportata qui. Il protocollo descrive come far crescere sia le collezioni di isolati di microbiota intestinale di C. elegans che le popolazioni di vermi sincroni in array multipozzetti prima di combinarli per i test. L’esempio fornito copre il test di 47 isolati batterici e un ceppo di controllo su due ceppi di vermi, in due saggi di stress in parallelo. Tuttavia, la pipeline di approccio è facilmente scalabile e applicabile allo screening di molte altre modalità. Pertanto, fornisce una configurazione versatile per esaminare rapidamente un panorama multiparametrico di condizioni biologiche e biochimiche che influiscono sulla salute di C. elegans.
Introduction
Il corpo umano ospita circa 10-100 trilioni di cellule microbiche vive (batteri, funghi archaea), che si trovano principalmente negli ambienti dell’intestino, della pelle e delle mucose1. In uno stato sano, questi forniscono benefici al loro ospite, tra cui la produzione di vitamine, la maturazione del sistema immunitario, la stimolazione delle risposte immunitarie innate e adattative agli agenti patogeni, la regolazione del metabolismo dei grassi, la modulazione delle risposte allo stress e altro ancora, con un impatto sulla crescita e lo sviluppo, l’insorgenza della malattia e l’invecchiamento 2,3,4,5 . Anche il microbiota intestinale si evolve considerevolmente nel corso della vita. L’evoluzione più drastica si verifica durante l’infanzia e la prima infanzia6, ma cambiamenti significativi si verificano anche con l’età, tra cui una diminuzione dell’abbondanza di Bifidobacterium e un aumento di Clostridium, Lactobacillus, Enterobacteriaceae e Enterococcus specie7. Lo stile di vita può alterare ulteriormente la composizione microbica intestinale portando alla disbiosi (perdita di batteri benefici, crescita eccessiva di batteri opportunisti), causando varie patologie come malattie infiammatorie intestinali, diabete e obesità5, ma anche contribuendo alle malattie di Alzheimer e Parkinson 8,9,10,11.
Questa consapevolezza ha contribuito in modo critico ad affinare il concetto di asse intestino-cervello (GBA), dove le interazioni tra la fisiologia intestinale (ora inclusi i microbi al suo interno) e il sistema nervoso sono considerate il principale regolatore del metabolismo animale e delle funzioni fisiologiche12. Tuttavia, il ruolo preciso del microbiota nella segnalazione intestino-cervello e i meccanismi d’azione associati sono lontani dall’essere pienamente compresi13. Con il microbiota intestinale che è un determinante chiave dell’invecchiamento sano, il modo in cui i batteri modulano il processo di invecchiamento è diventato oggetto di intense ricerche e polemiche 6,14,15.
Con la dimostrazione che il nematode Caenorhabditis elegans ospita un microbiota intestinale in buona fede dominato – come in altre specie – da Bacteroidetes, Firmicutes e Actinobacteria 16,17,18,19,20, la sua rapida ascesa come piattaforma sperimentale per studiare le interazioni commensali ospite-intestino21,22,23,24 ,25,26 ha notevolmente ampliato il nostro arsenale investigativo26,27,28,29. In particolare, gli approcci sperimentali ad alto rendimento disponibili per C. elegans per studiare le interazioni gene-dieta, gene-farmaco, gene-patogeno, ecc., possono essere adattati per esplorare rapidamente come isolati e cocktail batterici influenzano la salute e l’invecchiamento di C. elegans.
Il presente protocollo descrive una pipeline sperimentale per lo screening simultaneo di array di isolati o miscele batteriche impostati in piastre multipozzetto per gli effetti sulla resistenza allo stress di C. elegans come proxy per la salute, che può essere utilizzato per identificare i probiotici. Descrive in dettaglio come far crescere grandi popolazioni di vermi e gestire array batterici in formati di piastre a 96 e 384 pozzetti prima di elaborare i vermi per l’analisi automatizzata della resistenza allo stress utilizzando un lettore di piastre a fluorescenza (Figura 1). L’approccio si basa su saggi di sopravvivenza automatizzati label-free (LFASS)30 che sfruttano il fenomeno della fluorescenza di morte31, per cui i vermi morenti producono un’esplosione di fluorescenza blu che può essere utilizzata per individuare il momento della morte. La fluorescenza blu è emessa dagli esteri glucosilici dell’acido antranilico immagazzinati nei granuli intestinali di C. elegans (un tipo di organello correlato al lisosoma), che scoppiano quando una cascata necrotica viene innescata nell’intestino del verme alla morte31.
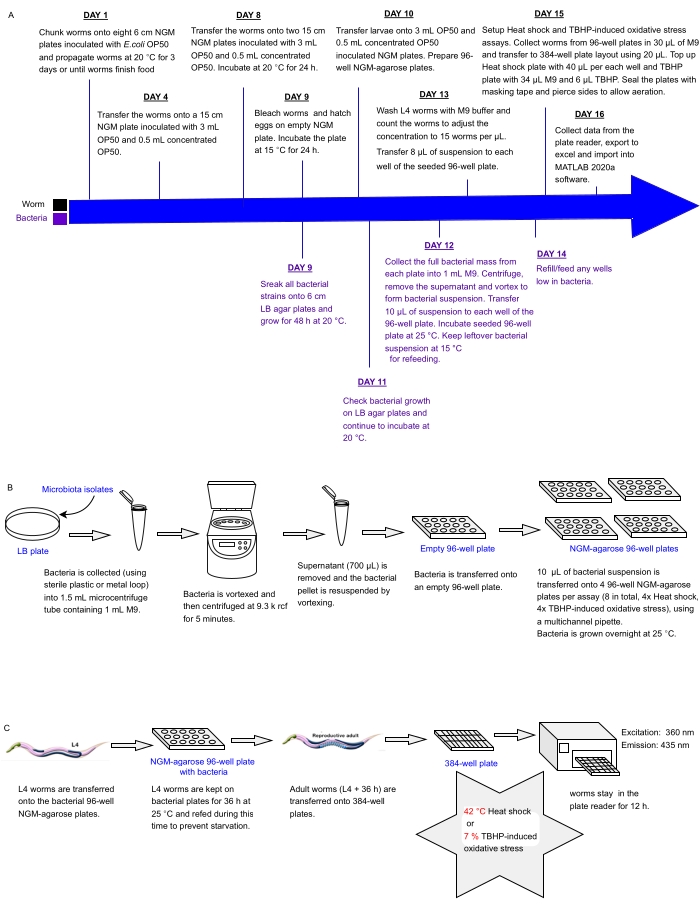
Figura 1: Flusso di lavoro sperimentale per lo screening ad alta produttività di isolati batterici con impatto sulla resistenza allo stress di C. elegans . (A) Cronologia per la manutenzione di vermi e batteri e la configurazione del test. (B) Configurazione e gestione dell’array di piastre batteriche a 96 pozzetti. (C) Configurazione della piastra a vite senza fine a 384 pozzetti. Fare clic qui per visualizzare una versione ingrandita di questa figura.
Protocol
Representative Results
Discussion
C. elegans offre molti vantaggi per lo screening rapido di più parametri sperimentali contemporaneamente, grazie alle sue dimensioni ridotte, trasparenza, sviluppo rapido, breve durata, economicità e facilità di manipolazione. Il suo genoma considerevolmente più semplice, il piano corporeo, il sistema nervoso, l’intestino e il microbioma, ma complessi e abbastanza simili agli esseri umani, lo rendono un potente modello preclinico, in cui è possibile ottenere informazioni meccanicistiche durante i test di ef…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Ringraziamo il CGC Minnesota (Madison, USA, NIH – P40 OD010440) per aver fornito ceppi di vermi e OP50 e Pr. Hinrich Schulenburg (CAU, Kiel, Germania) per aver fornito tutti gli isolati microbici ambientali qui raffigurati. Questo lavoro è stato finanziato da una sovvenzione UKRI-BBSRC ad AB (BB/S017127/1). JM è finanziato da una borsa di studio di dottorato FHM della Lancaster University.
Materials
| 10 cm diameter plates (Non-vented) | Fisher Scientific | 10720052 | Venting is not necessary for bacterial cultures |
| 15 cm diameter plates (Vented) | Fisher Scientific | 168381 | |
| 384-well black, transparent flat bottom plates | Corning | 3712 or 3762 | Not essential to be sterile for fast stress assays |
| 6 cm diameter plates (Vented) | Fisher Scientific | 150288 | Venting is necessary for worm cultures to avoid hypoxia |
| 96-well transparent plates (Biolite) | Thermo | 130188 | |
| Agar (<4% ash) | Sigma-Aldrich | 102218041 | Good quality agar is important for the structural integrity of the culture media, to avoid worm burrowing |
| Agarose | Fisher Scientific | BP1356 | |
| Avanti Centrifuge J-26 XP | Beckman coulter | ||
| Bleach | Honeywell | 425044 | |
| Calcium chloride | Sigma-Aldrich | C5080 | |
| Centrifuge 5415 R | Eppendorf | ||
| Centrifuge 5810 R | Eppendorf | ||
| Cholesterol | Sigma-Aldrich | C8667 | |
| LB agar | Difco | 240110 | |
| LB broth | Invitrogen | 12795084 | |
| LoBind tips | VWR | 732-1488 | Lo-bind reduce worm loss during transfers |
| LoBind tubes | Eppendorf | 22431081 | |
| Magnesium sulfate | Fisher Scientific | M/1100/53 | |
| Plate reader- infinite M nano+ | Tecan | Monochromator setup enables fluorescence tuning but adequate filter-based setups may be used | |
| Plate reader- Spark | Tecan | ||
| Potassium phosphate monobasic | Honeywell | P0662 | |
| Sodium chloride | Sigma-Aldrich | S/3160/63 | |
| Stereomicroscope setup with transillumination base | Leica | MZ6, or M80 | Magnification from 0.6-0.8x up to 40-60x is necessary, as is a good quality transillumination base with a deformable, titable or slidable mirror to adjust contrast |
| t-BHP (tert-Butyl hydroperoxide) | Sigma-Aldrich | 458139 | |
| Transparent adhesive seals Nunc | Fisher Scientific | 101706871 | It is important that it is transparent and that it can tolerate the temperatures involved in the assays. |
| Tryptophan | Sigma-Aldrich | 1278-7099 | |
| Yeast extract | Fisher Scientific | BP1422 |
References
- Krishna, S., et al. Integrating microbiome network: establishing linkages between plants, microbes and human health. The Open Microbiology Journal. 13, 330-342 (2019).
- Amon, P., Sanderson, I. What is the microbiome. Archives of Disease in Childhood – Education & Practice Edition. 102 (5), 257-260 (2017).
- Belkaid, Y., Harrison, O. J. Homeostatic immunity and the microbiota. Immunity. 46 (4), 562-576 (2017).
- Cabreiro, F., Gems, D. Worms need microbes too: microbiota, health and aging in Caenorhabditis elegans. EMBO Molecular Medicine. 5 (9), 1300-1310 (2013).
- Vaga, S., et al. Compositional and functional differences of the mucosal microbiota along the intestine of healthy individuals. Scientific Reports. 10 (1), 14977 (2020).
- Nagpal, R., et al. Gut microbiome and aging: Physiological and mechanistic insights. Nutrition and Healthy Aging. 4 (4), 267-285 (2018).
- Mitsuoka, T. Establishment of intestinal bacteriology. Biosci Microbiota Food Health. 33 (3), 99-116 (2014).
- Bonfili, L., et al. Microbiota modulation as preventative and therapeutic approach in Alzheimer’s disease. The FEBS Journal. 288 (9), 2836-2855 (2021).
- Vendrik, K. E. W., et al. Fecal microbiota transplantation in neurological disorders. Frontiers in Cellular and Infection Microbiology. 10, 98 (2020).
- Wang, Q., et al. The role of gut dysbiosis in Parkinson’s disease: mechanistic insights and therapeutic options. Brain. 144 (9), 2571-2593 (2021).
- Zhu, X., et al. The relationship between the gut microbiome and neurodegenerative diseases. Neuroscience Bulletin. 37 (10), 1510-1522 (2021).
- Miller, I. The gut-brain axis: historical reflections. Microbial Ecology in Health and Disease. 29 (1), 1542921 (2018).
- Foster, J. A., Rinaman, L., Cryan, J. F. Stress & the gut-brain axis: Regulation by the microbiome. Neurobiology of Stress. 7, 124-136 (2017).
- Coman, V., Vodnar, D. C. Gut microbiota and old age: Modulating factors and interventions for healthy longevity. Experimental Gerontology. 141, 111095 (2020).
- Conway, J., Duggal, N. A. Ageing of the gut microbiome: Potential influences on immune senescence and inflammageing. Ageing Research Reviews. 68, 101323 (2021).
- Berg, M., et al. Assembly of the Caenorhabditis elegans gut microbiota from diverse soil microbial environments. The ISME Journal. 10 (8), 1998-2009 (2016).
- Dirksen, P., et al. CeMbio – The Caenorhabditis elegans Microbiome Resource. G3: Genes, Genomes, Genetics. 10 (9), 3025-3039 (2020).
- Dirksen, P., et al. The native microbiome of the nematode Caenorhabditis elegans: gateway to a new host-microbiome model. BMC Biology. 14, 38 (2016).
- Samuel, B. S., Rowedder, H., Braendle, C., Felix, M. A., Ruvkun, G. Caenorhabditis elegans responses to bacteria from its natural habitats. Proceedings of the National Academy of Sciences of the United States of America. 113 (27), 3941-3949 (2016).
- Zimmermann, J., et al. The functional repertoire contained within the native microbiota of the model nematode Caenorhabditis elegans. The ISME Journal. 14 (1), 26-38 (2020).
- Dinic, M., et al. Host-commensal interaction promotes health and lifespan in Caenorhabditis elegans through the activation of HLH-30/TFEB-mediated autophagy. Aging. 13 (6), 8040-8054 (2021).
- Goya, M. E., et al. Probiotic Bacillus subtilis protects against alpha-Synuclein aggregation in C. elegans. Cell Reports. 30 (2), 367-380 (2020).
- Hacariz, O., Viau, C., Karimian, F., Xia, J. The symbiotic relationship between Caenorhabditis elegans and members of its microbiome contributes to worm fitness and lifespan extension. BMC Genomics. 22 (1), 364 (2021).
- Shin, M. G., et al. Bacteria-derived metabolite, methylglyoxal, modulates the longevity of C. elegans through TORC2/SGK-1/DAF-16 signaling. Proceedings of the National Academy of Sciences of the United States of America. 117 (29), 17142-17150 (2020).
- Zhang, F., et al. Natural genetic variation drives microbiome selection in the Caenorhabditis elegans gut. Current Biology. 31 (12), 2603-2618 (2021).
- Zhang, F., et al. High-throughput assessment of changes in the Caenorhabditis elegans gut microbiome. Methods in Molecular Biology. 2144, 131-144 (2020).
- Chan, J. P., et al. Using bacterial transcriptomics to investigate targets of host-bacterial interactions in Caenorhabditis elegans. Scientific Reports. 9 (1), 5545 (2019).
- Hartsough, L. A., et al. Optogenetic control of gut bacterial metabolism to promote longevity. Elife. 9, 56849 (2020).
- Pryor, R., et al. Host-microbe-drug-nutrient screen identifies bacterial effectors of Metformin therapy. Cell. 178 (6), 1299-1312 (2019).
- Benedetto, A., et al. New label-free automated survival assays reveal unexpected stress resistance patterns during C. elegans aging. Aging Cell. 18 (5), 12998 (2019).
- Coburn, C., et al. Anthranilate fluorescence marks a calcium-propagated necrotic wave that promotes organismal death in C. elegans. PLOS Biology. 11 (7), 1001613 (2013).
- Porta-de-la-Riva, M., Fontrodona, L., Villanueva, A., Ceron, J. Basic Caenorhabditis elegans methods: synchronization and observation. Journal of Visualized Experiments. (64), e4019 (2012).
- Stiernagle, T. Maintenance of C. elegans. WormBook. , 1-11 (2006).
- Naomi, R., et al. Probiotics for Alzheimer’s disease: a systematic review. Nutrients. 14 (1), 20 (2021).
- Zheng, S. Y., et al. Potential roles of gut microbiota and microbial metabolites in Parkinson’s disease. Ageing Research Reviews. 69, 101347 (2021).
- Gill, M. S., Olsen, A., Sampayo, J. N., Lithgow, G. J. An automated high-throughput assay for survival of the nematode Caenorhabditis elegans. Free Radical Biology and Medicine. 35 (6), 558-565 (2003).
- Park, H. -. E. H., Jung, Y., Lee, S. -. J. V. Survival assays using Caenorhabditis elegans. Molecules and Cells. 40 (2), 90-99 (2017).
- Partridge, F. A., et al. An automated high-throughput system for phenotypic screening of chemical libraries on C. elegans and parasitic nematodes. International Journal for Parasitology: Drugs and Drug Resistance. 8 (1), 8-21 (2018).
- Rahman, M., et al. NemaLife chip: a micropillar-based microfluidic culture device optimized for aging studies in crawling C. elegans. Scientific Reports. 10 (1), 16190 (2020).
- Stroustrup, N., et al. The Caenorhabditis elegans lifespan machine. Nature Methods. 10 (7), 665-670 (2013).
- Xian, B., et al. WormFarm: a quantitative control and measurement device toward automated Caenorhabditis elegans aging analysis. Aging Cell. 12 (3), 398-409 (2013).
- Brown, A. E., Schafer, W. R. Unrestrained worms bridled by the light. Nature Methods. 8 (2), 129-130 (2011).
- Churgin, M. A., et al. Longitudinal imaging of Caenorhabditis elegans in a microfabricated device reveals variation in behavioral decline during aging. Elife. 6, 26652 (2017).
- Jushaj, A., et al. Optimized criteria for locomotion-based healthspan evaluation in C. elegans using the WorMotel system. PLoS One. 15 (3), 0229583 (2020).
- Nambyiah, P., Brown, A. E. X. Quantitative behavioural phenotyping to investigate anaesthesia induced neurobehavioural impairment. Scientific Reports. 11 (1), 19398 (2021).
- Squiban, B., Belougne, J., Ewbank, J., Zugasti, O. Quantitative and automated high-throughput genome-wide RNAi screens in C. elegans. Journal of Visualized Experiments. (60), e3448 (2012).
- Zugasti, O., et al. Activation of a G protein-coupled receptor by its endogenous ligand triggers the innate immune response of Caenorhabditis elegans. Nature Immunology. 15 (9), 833-838 (2014).
- Zugasti, O., et al. A quantitative genome-wide RNAi screen in C. elegans for antifungal innate immunity genes. BMC Biology. 14, 35 (2016).

