检查骨骼肌中肌内脂肪形成及其细胞起源的指南
Summary
用肌内脂肪代替健康的肌肉组织是人类疾病和病症的突出特征。该协议概述了如何可视化,成像和量化肌内脂肪,从而可以严格研究肌内脂肪形成的机制。
Abstract
纤维脂肪源性祖细胞(FAPs)是间充质基质细胞,在骨骼肌稳态和再生过程中起关键作用。FAPs构建和维护充当分子肌纤维支架的细胞外基质。此外,FAPs对于肌纤维再生是必不可少的,因为它们分泌肌肉干细胞(MuSC)感知的多种有益因子。然而,在患病状态下,FAPs是肌内脂肪和纤维化瘢痕组织的细胞起源。这种脂肪纤维化是肌肉减少症和神经肌肉疾病(如杜氏肌营养不良症)的标志。确定FAPs分化为肌内脂肪的原因和方式的一个重要障碍是脂肪细胞的有效保存和随后的可视化,特别是在冷冻组织切片中。传统的骨骼肌组织处理方法,如速冻,不能正确保存单个脂肪细胞的形态,从而阻止准确的可视化和定量。为了克服这一障碍,开发了一种严格的方案,保留了骨骼肌切片中的脂肪细胞形态,允许肌肉内脂肪的可视化,成像和定量。该协议还概述了如何处理RT-qPCR的肌肉组织的一部分,使用户能够通过观察脂肪生成基因表达的差异来确认观察到的脂肪形成变化。此外,它可以通过肌肉样品的全载免疫荧光来可视化脂肪细胞。最后,该协议概述了如何对表达 Pdgfrα的FAPs进行遗传谱系示踪,以研究FAPs的脂肪转化。该协议始终如一地产生脂肪细胞的高分辨率和形态学上准确的免疫荧光图像,以及RT-qPCR的确认,允许对肌内脂肪进行稳健,严格和可重复的可视化和定量。总之,这里描述的分析管道是提高我们对FAPs如何分化成肌内脂肪的理解的第一步,并为验证防止脂肪形成的新干预措施提供了一个框架。
Introduction
脂肪纤维化对健康肌肉组织的浸润是杜氏肌营养不良症(DMD)和其他神经肌肉疾病,以及肌肉减少症,肥胖症和糖尿病的显着特征1,2,3,4,5,6,7,8,9,10.虽然在这些情况下脂肪浸润增加与肌肉功能下降密切相关,但我们对肌内脂肪形成的原因和方式的了解仍然有限。FAPs是一种多能的间充质基质细胞群,存在于大多数成人器官中,包括骨骼肌11,12。然而,随着年龄的增长和慢性疾病,FAPs产生纤维化瘢痕组织并分化成脂肪细胞,其位于单个肌纤维之间并形成肌内脂肪13,14,15,16,17,18,19,20。
为了开始对抗肌内脂肪的形成,需要定义FAPs如何变成脂肪细胞的机制。PDGFRα是该领域识别肌肉内FAPs的“金标准”标记13,16,17,18,20,21,22,23,24,25,26,27。结果,在Pdgfrα前驱体的控制下,已经产生了几种小鼠他莫昔芬诱导的Cre系,允许使用Cre-LoxP系统27,28,29在体内遗传操纵FAPs。例如,通过将这种可诱导的Cre谱系与遗传报告基因相结合,可以执行FAPs的谱系追踪,我们已经成功地将这种策略应用于肌肉和白色脂肪组织中的命运映射FAPs20,30。除了谱系追踪之外,这些Cre品系还为研究FAP到脂肪的转化提供了有价值的工具。
定义FAPs脂肪转化为肌内脂肪的机制的一个主要障碍是能够严格和可重复地量化在不同条件下形成的肌内脂肪量。关键是平衡肌肉和脂肪组织的保存,并将其与可用的染色方法相匹配,以可视化脂肪细胞。例如,骨骼肌通常在没有事先固定的情况下被速冻,保留肌纤维但破坏脂肪细胞形态(图1)。相反,固定后进行石蜡包埋,同时显示最佳的组织组织学,包括脂肪细胞,去除所有脂质,从而使大多数亲脂性染料(例如常用的染料油红O)无法使用。
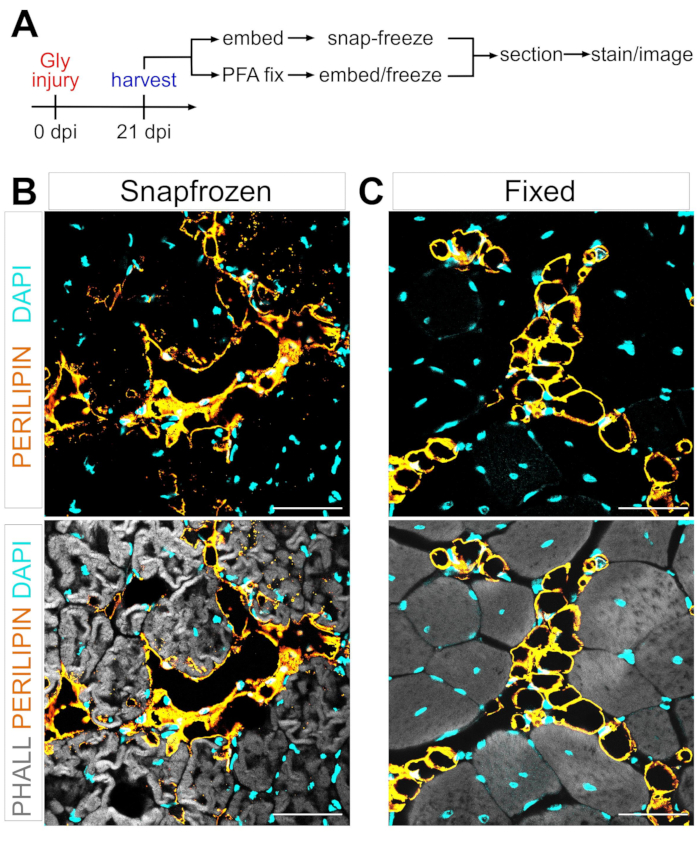
图1:速冻肌肉组织中肌内脂肪与固定肌肉组织中的代表性图像。免疫荧光图像显示甘油损伤后21天(B)速冻和(C)固定TA内的脂肪细胞(黄色),肌纤维(灰色)和细胞核(青色)。比例尺:50 μm.请点击此处查看此图的大图。
这里描述的方案保留了肌纤维和脂肪细胞的形态,并允许可视化和分析多种细胞类型。该方法基于多聚甲醛(PFA)固定肌肉组织中脂肪细胞的免疫荧光染色,允许与多种抗体共染色。它还可以很容易地适应于使用全安装成像在完整组织中的空间显示肌内脂肪,从而提供有关肌肉内脂肪的细胞微环境的信息。此外,该协议可以与我们最近发表的方法相结合,以确定固定肌肉组织31中肌纤维的横截面积,这是评估肌肉健康的重要测量。本文还概述了将这种方法与遗传谱系追踪相结合,以绘制FAPs分化为脂肪细胞的命运图谱。因此,这里描述的多功能方案能够对FAPs及其在组织切片和完整组织中分化为肌内脂肪进行严格且可重复的评估。
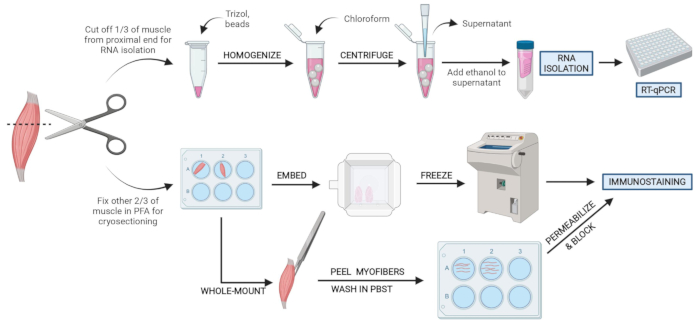
图 2:协议概述示意图。 组织处理的示意图概述,其中三分之一的TA被去除,快速冷冻和匀浆,以便 随后通过 RT-qPCR进行RNA分离和转录分析。另外三分之二的TA是PFA固定的,并经过加工,用于对冷冻切片或全纤维进行免疫染色。 请点击此处查看此图的大图。
Protocol
Representative Results
Discussion
该协议概述了一个广泛而详细的协议,允许对肌内脂肪进行有效的可视化和严格的定量。通过将同一肌肉分成两部分,一部分用于免疫荧光,另一部分用于RT-qPCR分析,该方案也非常通用。它还可以与FAPs的遗传谱系追踪相结合,以研究它们在某些条件下转化为脂肪细胞,并且高度适应于标记和成像多种其他细胞类型。
可视化肌内脂肪最常用的方法是石蜡切片,然后是苏木精和曙红染色或冷冻切片,用于亲脂性染料(如油红O(ORO))。然而,虽然石蜡处理的组织保持最佳的组织学,但相同的过程也提取所有脂质,防止使用亲脂性染料。虽然亲脂染色方法对PFA固定和未固定的组织切片都有效,但脂质液滴很容易通过对盖玻片施加压力而移位,从而扭曲肌内脂肪的空间分布。为了避免这种情况,最近的一项研究建立了一个严格的方案,使用全装载方法可视化ORO +脂肪细胞。为此,作者将TA去细胞化,以可视化整个TA41中肌内脂肪的空间分布。尽管这种技术功能强大,但它也可以防止使用其他共染色剂来标记其他细胞结构。这里介绍的整个免疫荧光方法可用于将脂肪细胞与各种标记物共染色,从而可以精细地映射细胞环境。然而,一个主要挑战是抗体的组织渗透。保持在一起的纤维越多,抗体就越难以均匀地穿透和结合所有可用的抗原。因此,当观察小组纤维时,这种方法是最有效的。同时,这也是一个限制,因为当只关注小的,剥离的纤维束时,肌内脂肪的整体解剖位置正在丢失。然而,随着目前新型组织清除方法和新的成像技术的发展,在未来42,43,44中将有可能实现更大的组织渗透和可视化。
虽然先前的肌肉组织固定保留了脂肪细胞的形态,但它也给评估肌纤维的大小带来了挑战,肌纤维是肌肉健康的重要衡量标准。肌纤维大小是通过测量肌纤维的横截面积来确定的。我们之前已经报道过,先前对肌肉组织的固定将导致大多数可用于轮廓肌纤维的标志物失效31。为了克服这一障碍,我们开发了一种新颖的图像分割管道,即使在固定的肌肉部分31中也可以测量肌纤维的大小。因此,我们已经建立了一个强大而高效的组织处理管道,与该协议相结合,克服了先前固定肌肉组织引起的大多数缺点。
这种方法的另一个主要优点是多功能性。通过将TA分成两部分,可以从一块肌肉获得的信息量最大化。这不仅减少了动物数量,而且还通过基因表达确认组织学,从而增加了额外的控制层,反之亦然。此外,除了脂肪生成基因之外,还可以检查许多不同的基因。分离的RNA也可用于整个肌肉RNA酶实验。最后,速冻肌肉块也可用于蛋白质工作。该协议的一个限制是损伤在整个TA长度上不一致的可能性。这可能导致两个肌肉部位在它们所含的肌内脂肪量上出现差异的情况,并且可能需要从任何下游分析中排除此类样本。因此,建议不要简单地依靠RT-qPCR来得出关于肌内脂肪量的主要结论,而是作为组织学定量的支持数据。
总之,该协议概述了一个强大,高效和严格的组织处理管道,该管道将允许肌内脂肪的可视化和定量,这是开发对抗脂肪纤维化的新治疗方案的第一步。同时,它是多功能的,可以适应肌肉内的许多不同细胞类型以及其他组织中的脂肪细胞。
Disclosures
The authors have nothing to disclose.
Acknowledgements
我们感谢Kopinke实验室的成员帮助收集数据并批判性地阅读手稿。我们还感谢佛罗里达大学真菌研究所的成员对手稿的宝贵意见。这项工作得到了NIH拨款1R01AR079449的支持。图 2 是使用生物渲染器创建的。
Materials
| 16% PFA (Pack of 12, 10 mL bottles) | Electron Miscroscopy Sciences | 15710 | |
| 2.0 mL Microcentrifuge Tubes | Fisher Scientific | 05-408-138 | microcentrifuge tubes for snapfreezing/bead beating |
| 2-Methylbutane (4 L) | Fisher Chemical | O3551-4 | isopentane |
| Absolute Ethanol (200 proof) | ThermoFisher Scientific | BP2818100 | |
| AffiniPure Fab fragment donkey anti-mouse | Jackson ImmunoResearch | 715-007-003 | mouse-on-mouse blocking |
| Alexa Fluor 488 donkey anti-chicken secondary antibody | Jackson ImmunoResearch | 703-545-155 | |
| Alexa Fluor 488 donkey anti-mouse secondary antibody | Invitrogen | A21202 | |
| Alexa Fluor 488 donkey anti-rabbit secondary antibody | Invitrogen | A21206 | |
| Alexa Fluor 568 donkey anti-goat secondary antibody | Invitrogen | A11057 | |
| Alexa Fluor 568 donkey anti-rabbit secondary antibody | Invitrogen | A11037 | |
| Alexa Fluor 568 Phalloidin antibody | Invitrogen | A12380 | Dissolved in 1.5 mL methanol (~66 µM working solution) |
| BioLite 24-well Multidishes | ThermoFisher Scientific | 930186 | 24 well plate for PFA tissue incubation |
| Biometra TOne | analytikjena | 8462070301 | Thermal cycler |
| Chicken anti-GFP antibody | Aves Labs | GFP-1020 | |
| Chloroform/isoamyl alcohol 24:1(v/v) for molecular biology, DNAse, RNAse, and Protease free | ThermoFisher Scientific | AC327155000 | |
| Corn oil | Sigma Aldrich | C8267 | |
| DAPI stain | Invitrogen | D1306 | 150 µM working solution in dH2O |
| Donkey Serum (100 mL) | Millipore Sigma | 5058837 | for blocking solution |
| Dumont #5 Forceps | Fine Science Tools | 11251-20 | sharp-tipped tweezers |
| Fine Scissors Straight 9 cm | Fine Science Tools | 14060-09 | |
| Fluoromount-G | SouthernBiotech | 0100-01 | mounting medium |
| Glycerol, 99.5%, for molecular biology (500 mL) | Acros Organics | 327255000 | |
| Goat anti-PDGFRα antibody | R&D | AF1062 | |
| Hybridization Oven | VWR | 230301V | for Tamoxifen incubation |
| ImmEdge Hydrophobic Barrier PAP Pen | Vector Laboratories | H-4000 | hydrophobic pen |
| Insulin Syringe with Micro-Fine IV needle (28 G) | BD | 329461 | |
| Insulin Syringe with Slip Tip, 1 mL | BD | 329654 | Insulin syringe without needle, for oral gavaging |
| iScript cDNA Synthesis Kit | Bio-Rad | 1708890 | |
| Isoflurane | Patterson Veterinary | 78938441 | |
| Leica DMi8 inverted microscope | Leica | micrscope used for widefield IF and confocal imaging | |
| Micro Slides | VWR | 48311-703 | positively charged microscope slides |
| mouse anti-MYOD antibody | Invitrogen | MA1-41017 | |
| mouse anti-PAX7 antibody (supernatant) | DSHB | AB 428528 | |
| MX35 Premier+ Microtome blades | ThermoFisher Scientific | 3052835 | microtome blades |
| NanoDrop 2000 Spectrophotometer | ThermoFisher Scientific | ND2000 | spectrophotometer for RNA yield |
| Play-Doh | Hasbro | modeling compound | |
| PowerUp SYBR Green Master Mix | ThermoFisher Scientific | A25742 | green dye PCR master mix |
| Puralube Vet Ointment | Puralube | 17033-211-38 | vet ophthalmic ointment |
| QuantStudi 6 Flex Real-Time 384-well PCR System | Applied Biosystems | 4485694 | qPCR machine |
| Rabbit anti-perilipin antibody | Cell Signaling Technology | 9349S | |
| Red-Rotor Shaker | Hoefer Scientific | PR70-115V | shaker for IF staining |
| Richard-Allan Scientific Slip-Rite Cover Glass | ThermoFisher Scientific | 152460 | coverslips |
| RNeasy Mini Kit | QIAGEN | 74106 | contains mini spin columns |
| Safe-Lock Tubes 1.5 ml, natural | Eppendorf | 22363204 | |
| Sample Tubes RB (2 mL) | QIAGEN | 990381 | |
| Sodium azide | Alfa Aesar | 14314 | |
| Stainless Steel Beads, 2.8 mm | Precellys | KT03961-1-101.BK | small beads |
| Stainless Steel Beads, 5 mm | QIAGEN | 69989 | medium beads |
| Stainless Steel Beads, 7 mm | QIAGEN | 69990 | large beads |
| Stainless Steel Disposable Scalpels | Miltex | 327-4102 | scalpel |
| Stainless steel feeding tube, 20 G x 38 mm, straight | Instech Laboratories | FTSS-20S-3 | gavage needle |
| Tamoxifen | Toronto Research Chemicals | T006000 | |
| Tissue Plus O.C.T. Compound | Fisher HealthCare | 4585 | embedding medium |
| TissueLyser LT | QIAGEN | 85600 | bead beater |
| TissueLyser LT Adapter, 12-Tube | QIAGEN | 69980 | |
| Tissue-Tek Cryomold | Sakura | 4566 | specimen molds |
| Triton X-100 | Alfa Aesar | A16046 | |
| TRIzol Reagent | ThermoFisher Scientific | 15596026 | guanidium thiocyanate |
| Tween20 (500 mL) | Fisher BioReagents | BP337-500 | |
| VWR Micro Slides – Superfrost Plus | VWR | 48311703 | |
| Wheaton Coplin staining jars | Millipore Sigma | S6016 | Coplin jar |
References
- Milad, N., et al. Increased plasma lipid levels exacerbate muscle pathology in the mdx mouse model of Duchenne muscular dystrophy. Skeletal Muscle. 7 (1), 19 (2017).
- Goodpaster, B. H., et al. Obesity, regional body fat distribution, and the metabolic syndrome in older men and women. Archives of Internal Medicine. 165 (7), 777-783 (2005).
- Goodpaster, B. H., et al. Association between regional adipose tissue distribution and both type 2 diabetes and impaired glucose tolerance in elderly men and women. Diabetes Care. 26 (2), 372-379 (2003).
- Goodpaster, B. H., et al. The loss of skeletal muscle strength, mass, and quality in older adults: the health, aging and body composition study. The Journals of Gerontology, Series A: Biological Sciences and Medical Sciences. 61 (10), 1059-1064 (2006).
- Goodpaster, B. H., Thaete, F. L., Kelley, D. E. Thigh adipose tissue distribution is associated with insulin resistance in obesity and in type 2 diabetes mellitus. American Journal of Clinical Nutrition. 71 (4), 885-892 (2000).
- Goodpaster, B. H., Theriault, R., Watkins, S. C., Kelley, D. E. Intramuscular lipid content is increased in obesity and decreased by weight loss. Metabolism. 49 (4), 467-472 (2000).
- Burakiewicz, J., et al. Quantifying fat replacement of muscle by quantitative MRI in muscular dystrophy. Journal of Neurology. 264 (10), 2053-2067 (2017).
- Murphy, W. A., Totty, W. G., Carroll, J. E. MRI of normal and pathologic skeletal muscle. American Journal of Roentgenology. 146 (3), 565-574 (1986).
- Willcocks, R. J., et al. Multicenter prospective longitudinal study of magnetic resonance biomarkers in a large duchenne muscular dystrophy cohort. Annals of Neurology. 79 (4), 535-547 (2016).
- Wokke, B. H., et al. Quantitative MRI and strength measurements in the assessment of muscle quality in Duchenne muscular dystrophy. Neuromuscular Disorders. 24 (5), 409-416 (2014).
- Contreras, O., Rossi, F. M. V., Theret, M. Origins, potency, and heterogeneity of skeletal muscle fibro-adipogenic progenitors-time for new definitions. Skeletal Muscle. 11 (1), 16 (2021).
- El Agha, E., et al. Mesenchymal stem cells in fibrotic disease. Cell Stem Cell. 21 (2), 166-177 (2017).
- Joe, A. W., et al. Muscle injury activates resident fibro/adipogenic progenitors that facilitate myogenesis. Nature Cell Biology. 12 (2), 153-163 (2010).
- Liu, W., Liu, Y., Lai, X., Kuang, S. Intramuscular adipose is derived from a non-Pax3 lineage and required for efficient regeneration of skeletal muscles. Developmental Biology. 361 (1), 27-38 (2012).
- Scott, R. W., Arostegui, M., Schweitzer, R., Rossi, F. M. V., Underhill, T. M. Hic1 defines quiescent mesenchymal progenitor subpopulations with distinct functions and fates in skeletal muscle regeneration. Cell Stem Cell. 25 (6), 797-813 (2019).
- Uezumi, A., et al. Fibrosis and adipogenesis originate from a common mesenchymal progenitor in skeletal muscle. Journal of Cell Science. 124, 3654-3664 (2011).
- Hogarth, M. W., et al. Fibroadipogenic progenitors are responsible for muscle loss in limb girdle muscular dystrophy 2B. Nature Communications. 10 (1), 2430 (2019).
- Uezumi, A., Fukada, S., Yamamoto, N., Takeda, S., Tsuchida, K. Mesenchymal progenitors distinct from satellite cells contribute to ectopic fat cell formation in skeletal muscle. Nature Cell Biology. 12 (2), 143-152 (2010).
- Stumm, J., et al. Odd skipped-related 1 (Osr1) identifies muscle-interstitial fibro-adipogenic progenitors (FAPs) activated by acute injury. Stem Cell Research. 32, 8-16 (2018).
- Kopinke, D., Roberson, E. C., Reiter, J. F. Ciliary Hedgehog signaling restricts injury-induced adipogenesis. Cell. 170 (2), 340-351 (2017).
- Huang, Y., Das, A. K., Yang, Q. Y., Zhu, M. J., Du, M. Zfp423 promotes adipogenic differentiation of bovine stromal vascular cells. PLoS One. 7 (10), 47496 (2012).
- Sun, Y. -. M., et al. PDGFRα regulated by miR-34a and FoxO1 promotes adipogenesis in porcine intramuscular preadipocytes through Erk signaling pathway. International Journal of Molecular Sciences. 18 (11), 2424 (2017).
- Te, L. J. I., Doherty, C., Correa, J., Batt, J. Identification, isolation, and characterization of fibro-adipogenic progenitors (FAPs) and myogenic progenitors (MPs) in skeletal muscle in the rat. Journal of Visualized Experiments: JoVE. (172), e61750 (2021).
- Lukjanenko, L., et al. Aging disrupts muscle stem cell function by impairing matricellular WISP1 secretion from fibro-adipogenic progenitors. Cell Stem Cell. 24 (3), 433-446 (2019).
- Santini, M. P., et al. Tissue-resident PDGFRalpha(+) progenitor cells contribute to fibrosis versus healing in a context- and spatiotemporally dependent manner. Cell Reports. 30 (2), 555-570 (2020).
- Uezumi, A., et al. Identification and characterization of PDGFRalpha+ mesenchymal progenitors in human skeletal muscle. Cell Death & Disease. 5, 1186 (2014).
- Wosczyna, M. N., et al. Mesenchymal stromal cells are required for regeneration and homeostatic maintenance of skeletal muscle. Cell Reports. 27 (7), 2029-2035 (2019).
- Soliman, H., et al. Pathogenic potential of Hic1-expressing cardiac stromal progenitors. Cell Stem Cell. 26 (2), 205-220 (2020).
- Chung, M. I., Bujnis, M., Barkauskas, C. E., Kobayashi, Y., Hogan, B. L. M. Niche-mediated BMP/SMAD signaling regulates lung alveolar stem cell proliferation and differentiation. Development. 145 (9), (2018).
- Hilgendorf, K. I., et al. Omega-3 fatty acids activate ciliary FFAR4 to control adipogenesis. Cell. 179 (6), 1289-1305 (2019).
- Waisman, A., Norris, A. M., Elías Costa, M., Kopinke, D. Automatic and unbiased segmentation and quantification of myofibers in skeletal muscle. Scientific Reports. 11 (1), 11793 (2021).
- Kang, S. H., Fukaya, M., Yang, J. K., Rothstein, J. D., Bergles, D. E. NG2+ CNS glial progenitors remain committed to the oligodendrocyte lineage in postnatal life and following neurodegeneration. Neuron. 68 (4), 668-681 (2010).
- Srinivas, S., et al. Cre reporter strains produced by targeted insertion of EYFP and ECFP into the ROSA26 locus. BMC Developmental Biology. 1, 4 (2001).
- Lukjanenko, L., Brachat, S., Pierrel, E., Lach-Trifilieff, E., Feige, J. N. Genomic profiling reveals that transient adipogenic activation is a hallmark of mouse models of skeletal muscle regeneration. PLoS One. 8 (8), 71084 (2013).
- Mahdy, M. A., Lei, H. Y., Wakamatsu, J., Hosaka, Y. Z., Nishimura, T. Comparative study of muscle regeneration following cardiotoxin and glycerol injury. Annals of Anatomy = Anatomischer Anzeiger: Official Organ of the Anatomische Gesellscaft. 202, 18-27 (2015).
- Pisani, D. F., Bottema, C. D., Butori, C., Dani, C., Dechesne, C. A. Mouse model of skeletal muscle adiposity: a glycerol treatment approach. Biochemical and Biophysical Research Communications. 396 (3), 767-773 (2010).
- Kawai, H., et al. Experimental glycerol myopathy: a histological study. Acta Neuropathologica. 80 (2), 192-197 (1990).
- Schmittgen, T. D., Livak, K. J. Analyzing real-time PCR data by the comparative CT method. Nature Protocols. 3 (6), 1101-1108 (2008).
- Uezumi, A., et al. Mesenchymal Bmp3b expression maintains skeletal muscle integrity and decreases in age-related sarcopenia. The Journal of Clinical Investigation. 131 (1), 139617 (2021).
- Biferali, B., et al. Prdm16-mediated H3K9 methylation controls fibro-adipogenic progenitors identity during skeletal muscle repair. Science Advances. 7 (23), 9371 (2021).
- Biltz, N. K., Meyer, G. A. A novel method for the quantification of fatty infiltration in skeletal muscle. Skeletal Muscle. 7 (1), (2017).
- Vieites-Prado, A., Renier, N. Tissue clearing and 3D imaging in developmental biology. Development. 148 (18), (2021).
- Gómez-Gaviro, M. V., Sanderson, D., Ripoll, J., Desco, M. Biomedical applications of tissue clearing and three-dimensional imaging in health and disease. iScience. 23 (8), 101432 (2020).
- Ueda, H. R., et al. Tissue clearing and its applications in neuroscience. Nature Reviews Neuroscience. 21 (2), 61-79 (2020).

