Polysomrensning fra sojabønne symbiotiske knuder
Summary
Denne protokol beskriver en metode til eukaryot polysomrensning fra intakte sojabønneknuder. Efter sekventering kan standardrørledninger til genekspressionsanalyse bruges til at identificere differentielt udtrykte gener på transkriptom- og translatomniveauerne.
Abstract
Formålet med denne protokol er at tilvejebringe en strategi til undersøgelse af det eukaryote translatom af sojabønnen (Glycine max) symbiotisk knude. Dette papir beskriver metoder optimeret til at isolere planteafledte polyribosomer og deres tilknyttede mRNA’er, der skal analyseres ved hjælp af RNA-sekventering. For det første opnås cytoplasmatiske lysater gennem homogenisering i polysom- og RNA-bevarende betingelser fra hele, frosne sojabønneknuder. Derefter ryddes lysater ved centrifugering ved lav hastighed, og 15% af supernatanten anvendes til total RNA (TOTAL) isolering. Det resterende ryddede lysat bruges til at isolere polysomer ved ultracentrifugering gennem en tolags saccharosepude (12% og 33,5%). Polysom-associeret mRNA (PAR) renses fra polysomale pellets efter resuspension. Både TOTAL og PAR evalueres ved meget følsom kapillærelektroforese for at opfylde kvalitetsstandarderne for sekventeringsbiblioteker for RNA-seq. Som et eksempel på en downstream-applikation kan standardrørledninger til genekspressionsanalyse efter sekventering anvendes til at opnå differentielt udtrykte gener på transkriptom- og translatomniveauerne. Sammenfattende tillader denne metode i kombination med RNA-seq undersøgelsen af den translationelle regulering af eukaryote mRNA’er i et komplekst væv såsom den symbiotiske knude.
Introduction
Bælgplanter, såsom sojabønner (Glycine max), kan etablere symbiose med specifikke jordbakterier kaldet rhizobia. Dette mutualistiske forhold fremkalder dannelsen af nye organer, de symbiotiske knuder, på plantens rødder. Knuderne er planteorganerne, der er vært for bakterierne og består af værtsceller, hvis cytoplasma koloniseres med en specialiseret form for rhizobia kaldet bakterioider. Disse bakteroider katalyserer reduktionen af atmosfærisk nitrogen (N2) til ammoniak, som overføres til planten til gengæld for kulhydrater 1,2.
Selvom denne kvælstoffikserende symbiose er en af de mest velundersøgte plantemikrobesymbioser, er der stadig mange aspekter, der skal forstås bedre, såsom hvordan planter, der udsættes for forskellige abiotiske stressforhold, modulerer deres interaktion med deres symbiotiske partner, og hvordan dette påvirker knudemetabolismen. Disse processer kunne bedre forstås ved at analysere knudetranslatomet (dvs. delmængden af messenger RNA’er [mRNA’er] aktivt oversat). Polyribosomer eller polysomer er komplekser af flere ribosomer forbundet med mRNA, der almindeligvis anvendes til at studere oversættelse3. Polysomprofileringsmetoden består af analysen af mRNA’erne forbundet med polysomer og er med succes blevet brugt til at studere de posttranskriptionelle mekanismer, der styrer genekspression, der forekommer i forskellige biologiske processer 4,5.
Historisk set har genomekspressionsanalyse primært fokuseret på at bestemme mRNA-overflod 6,7,8,9. Der er imidlertid en mangel på korrelation mellem transkription og proteinniveauer på grund af de forskellige stadier af posttranskriptionel regulering af genekspression, især translation10,11,12. Desuden er der ikke observeret nogen afhængighed mellem ændringerne på transkriptomniveauet og dem, der forekommer på translatomniveau13. Den direkte analyse af det sæt mRNA’er, der oversættes, muliggør en mere nøjagtig og komplet måling af cellegenekspressionen (hvis endepunkt er proteinoverflod) end den, der opnås, når kun mRNA-niveauer analyseres14,15,16.
Denne protokol beskriver, hvordan planteafledte polysomer renses fra intakte sojabønneknuder ved differentiel centrifugering gennem en tolags saccharosepude (figur 1). Men da bakteroidafledte ribosomer også er til stede i knuderne, renses en blanding af ribosomer og RNA-arter, selvom de eukaryote repræsenterer hovedfraktionen (90% -95%). Den efterfølgende RNA-isolering, kvantificering og kvalitetskontrol er også beskrevet (figur 1). Denne protokol skal i kombination med RNA-seq give eksperimentelle resultater om translationel regulering af eukaryote mRNA’er i et komplekst væv, såsom den symbiotiske knude.
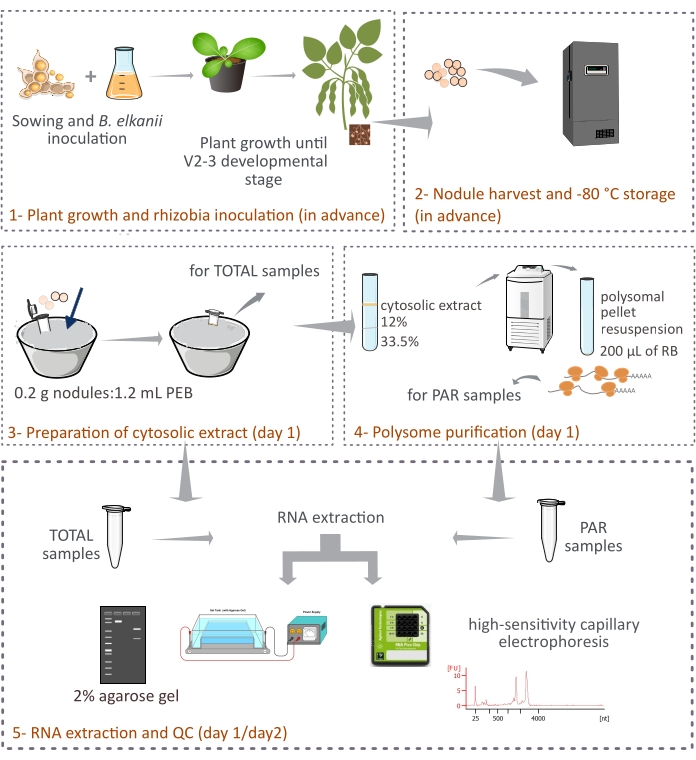
Figur 1: Skematisk oversigt over den foreslåede metode til eukaryot polysomrensning fra symbiotiske knuder. Ordningen giver et overblik over de trin, der følges i protokollen fra (1) plantevækst og (2) knudehøst til (3) forberedelse af cytosolekstrakter, (3) opnåelse af TOTAL-prøver og (4) PAR-prøver og (5) RNA-ekstraktion og kvalitetskontrol. Forkortelser: PEB = polysomekstraktionsbuffer; RB = resuspension buffer; TOTAL = total RNA; PAR = polysom-associeret mRNA. Klik her for at se en større version af denne figur.
Protocol
Representative Results
Discussion
At studere genekspressionsregulering på translationelt niveau er afgørende for bedre at forstå forskellige biologiske processer, da endepunktet for cellegenekspression er proteinmængde13,14. Dette kan vurderes ved at analysere translatomet af vævet eller organismen af interesse, for hvilket den polysomale fraktion skal oprenses, og dets associerede mRNA’er analyseres 3,4,34,35,36.<sup clas…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Denne forskning blev finansieret af CSIC I + D 2020-bevilling nr. 282, FVF 2017-bevilling nr. 210 og PEDECIBA (María Martha Sainz).
Materials
| Plant growth and rhizobia inoculation | |||
| Orbital shaker | Daihan Scientific | Model SHO-1D | |
| YEM-medium | Amresco | J850 (yeast extract) 0122 (mannitol) | |
| Water deficit treatment | |||
| KNO3 | Merck | 221295 | |
| Porometer | Decagon Device | Model SC-1 | |
| Scalpel | |||
| Preparation of cytosolic extracts | |||
| Brij L23 | Sigma-Aldrich | P1254 | |
| Centrifuge | Sigma | Model 2K15 | |
| Chloranphenicol | Sigma-Aldrich | C0378 | |
| Cycloheximide | Sigma-Aldrich | C7698 | |
| DOC | Sigma-Aldrich | 30970 | |
| DTT | Sigma-Aldrich | D9779 | |
| EGTA | Sigma-Aldrich | E3889 | |
| Igepal CA 360 | Sigma-Aldrich | I8896 | |
| KCl | Merck | 1.04936 | |
| MgCl2 | Sigma-Aldrich | M8266 | |
| Plastic tissue grinder | Fisher Scientific | 12649595 | |
| PMSF | Sigma-Aldrich | P7626 | |
| PTE | Sigma-Aldrich | P2393 | |
| Tris | Invitrogen | 15504-020 | |
| Triton X-100 | Sigma-Aldrich | T8787 | |
| Tween 20 | Sigma-Aldrich | P1379 | |
| Weighing dish | Deltalab | 1911103 | |
| Preparation of sucrose cushions | |||
| Sucrose | Invitrogen | 15503022 | |
| SW 40 Ti rotor | Beckman-Coulter | ||
| Ultracentrifuge | Beckman-Coulter | Optima L-100K | |
| Ultracentrifuge tubes | Beckman-Coulter | 344059 | 13.2 mL tubes |
| RNA extraction and quality control | |||
| Agarose | Thermo scientific | R0492 | |
| Bioanalyzer | Agilent | Model 2100. Eukaryote total RNA nano assay | |
| Chloroform | DI | 41191 | |
| Ethanol | Dorwil | UN1170 | |
| Isopropanol | Mallinckrodt | 3032-06 | |
| Glycogen | Sigma | 10814-010 | |
| TRIzol LS | Ambion | 102960028 | |
| Miscellaneous | |||
| Falcon tubes 15 mL | Biologix | 10-0152 | |
| Filter tips 10 µL | BioPointe Scientific | 321-4050 | |
| Filter tips 1000 µL | BioPointe Scientific | 361-1050 | |
| Filter tips 20 µL | BioPointe Scientific | 341-4050 | |
| Filter tips 200 µL | Tarsons | 528104 | |
| Microcentrifuge tubes 1.5 mL | Tarsons | 500010-N | |
| Microcentrifuge tubes 2.0 mL | Tarsons | 500020-N | |
| Sequencing company | Macrogen | ||
| Sterile 250 mL flask | Marienfeld | 4110207 |
References
- Limpens, E., et al. Cell- and tissue-specific transcriptome analyses of Medicago truncatula root nodules. PLoS ONE. 8 (5), 64377 (2013).
- Masson-Boivin, C., Giraud, E., Perret, X., Batut, J. Establishing nitrogen-fixing symbiosis with legumes: How many rhizobium recipes. Trends in Microbiology. 17 (10), 458-466 (2009).
- King, H. A., Gerber, A. P. Translatome profiling: Methods for genome-scale analysis of mRNA translation. Briefings in Functional Genomics. 15 (1), 22-31 (2014).
- Chassé, H., Boulben, S., Costache, V., Cormier, P., Morales, J. Analysis of translation using polysome profiling. Nucleic Acids Research. 45 (3), 15 (2017).
- Yángüez, E., Castro-Sanz, A. B., Fernández-Bautista, N., Oliveros, J. C., Castellano, M. M. Analysis of genome-wide changes in the translatome of Arabidopsis seedlings subjected to heat stress. PloS ONE. 8 (8), 71425 (2013).
- Brown, P. O., Botstein, D. Exploring the new world of the genome with DNA microarrays. Nature Genetics. 21, 33-37 (1999).
- Krishnamurthy, A., Ferl, R. J., Paul, A. -. L. Comparing RNA-Seq and microarray gene expression data in two zones of the Arabidopsis root apex relevant to spaceflight. Applications in Plant Sciences. 6 (11), 1197 (2018).
- Shulse, C. N., et al. High-throughput single-cell transcriptome profiling of plant cell types. Cell Reports. 27 (7), 2241-2247 (2019).
- Wang, Z., Gerstein, M., Snyder, M. RNA-Seq: A revolutionary tool for transcriptomics. Nature Reviews Genetics. 10 (1), 57-63 (2009).
- Kawaguchi, R., Girke, T., Bray, E. A., Bailey-Serres, J. Differential mRNA translation contributes to gene regulation under non-stress and dehydration stress conditions in Arabidopsis thaliana. The Plant Journal. 38 (5), 823-839 (2004).
- Larsson, O., Tian, B., Sonenberg, N. Toward a genome-wide landscape of translational control. Cold Spring Harbor Perspectives in Biology. 5 (1), 012302 (2013).
- Vogel, C., Marcotte, E. M. Insights into regulation of protein abundance from proteomics and transcriptomis analyses. Nature Reviews Genetics. 13 (4), 227-232 (2013).
- Tebaldi, T., et al. Widespread uncoupling between transcriptome and translatome variations after a stimulus in mammalian cells. BMC Genomics. 13 (220), 1-15 (2012).
- Wang, T., et al. Translating mRNAs strongly correlate to proteins in a multivariate manner and their translation ratios are phenotype specific. Nucleic Acids Research. 41 (9), 4743-4754 (2013).
- Ingolia, N. T. Ribosome profiling: New views of translation, from single codons to genome scale. Nature Reviews Genetics. 15 (3), 205-213 (2014).
- Lukoszek, R., Feist, P., Ignatova, Z. Insights into the adaptive response of Arabidopsis thaliana to prolonged thermal stress by ribosomal profiling and RNA-Seq. BMC Plant Biology. 16 (1), 1-13 (2016).
- Broughton, W. J., Dilworth, M. J. Control of leghaemoglobin synthesis in snake beans. Biochemical Journal. 125 (4), 1075-1080 (1971).
- Schroeder, A., et al. The RIN: An RNA integrity number for assigning integrity values to RNA measurements. BMC Molecular Biology. 7, 1-14 (2006).
- Rio, D. C., Ares, M., Hannon, G. J., Nilsen, T. W. Nondenaturing agarose gel electrophoresis of RNA nondenaturing agarose gel electrophoresis of RNA. Cold Spring Harbor Protocols. 2010 (6), 1-3 (2012).
- Ewels, P., Magnusson, M., Lundin, S., Käller, M. MultiQC: Summarize analysis results for multiple tools and samples in a single report. Bioinformatics. 32 (19), 3047-3048 (2016).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: A flexible trimmer for Illumina sequence data. Bioinformatics. 30 (15), 2114-2120 (2014).
- Marcel, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet.journal. 17 (1), 10-12 (2011).
- . Sickle: A sliding-window, adaptive, quality-based trimming tool for FastQ files (Version 1.33) Available from: https://github.com/najoshi/sickle (2011)
- Langmead, B., Salzberg, S. L. Fast gapped-read alignment with Bowtie 2. Nature Methods. 9 (4), 357-359 (2012).
- Kim, D., et al. TopHat2: Accurate alignment of transcriptomes in the presence of insertions, deletions and gene fusions. Genome Biology. 14 (4), 1-13 (2013).
- Patro, R., Duggal, G., Love, M. I., Irizarry, R. A., Kingsford, C. Salmon provides fast and bias-aware quantification of transcript expression. Nature Methods. 14 (4), 417-419 (2017).
- Liao, Y., Smyth, G. K., Shi, W. featureCounts: An efficient general purpose program for assigning sequence reads to genomic features. Bioinformatics. 30 (7), 923-930 (2013).
- Robinson, M. D., McCarthy, D. J., Smyth, G. K. edgeR: A Bioconductor package for differential expression analysis of digital gene expression data. Bioinformatics. 26 (1), 139-140 (2010).
- Love, M. I., Huber, W., Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biology. 15 (550), 1-21 (2014).
- Ritchie, M. E., et al. limma powers differential expression analyses for RNA-sequencing and microarray studies. Nucleic Acids Research. 43 (7), 47 (2015).
- Afgan, E., et al. The Galaxy platform for accessible, reproducible and collaborative biomedical analyses: 2018 update. Nucleic Acids Research. 46, 537-544 (2018).
- Hunt, G. P., et al. GEOexplorer: A webserver for gene expression analysis and visualisation. Nucleic Acids Research. , (2022).
- Imbeaud, S., et al. Towards standardization of RNA quality assessment using user-independent classifiers of microcapillary electrophoresis traces. Nucleic Acids Research. 33 (6), 1-12 (2005).
- Di Paolo, A., et al. PDCD4 regulates axonal growth by translational repression of neurite growth-related genes and is modulated during nerve injury responses. RNA. 26 (11), 1637-1653 (2020).
- Smircich, P., et al. Ribosome profiling reveals translation control as a key mechanism generating differential gene expression in Trypanosoma cruzi. BMC Genomics. 16 (1), 443 (2015).
- Eastman, G., Sharlow, E. R., Lazo, J. S., Bloom, G. S., Sotelo-Silveira, J. R. Transcriptome and translatome regulation of pathogenesis in Alzheimer’s disease model mice. Journal of Alzheimer’s Disease. 86 (1), 365-386 (2022).
- Zanetti, M. E., Chang, I. -. F., Gong, F., Galbraith, D. W., Bailey-Serres, J. Immunopurification of polyribosomal complexes of Arabidopsis for global analysis of gene expression. Plant physiology. 138 (2), 624-635 (2005).
- Mustroph, A., et al. Profiling translatomes of discrete cell populations resolves altered cellular priorities during hypoxia in Arabidopsis. Proceedings of the National Academy of Sciences of the United States of America. 106 (44), 18843-11848 (2009).
- Ingolia, N. T., Ghaemmaghami, S., Newman, J. R. S., Weissman, J. S. Genome-wide analysis in vivo of translation with nucleotide resolution using ribosome profiling. Science. 324 (5924), 218-223 (2009).
- Brar, G. A., Weissman, J. S. Ribosome profiling reveals the what, when, where and how of protein synthesis. Nature Reviews. Molecular Cell Biology. 16 (11), 651-664 (2015).
- Eastman, G., Smircich, P., Sotelo-Silveira, J. R. Following ribosome footprints to understand translation at a genome wide level. Computational and Structural Biotechnology Journal. 16, 167-176 (2018).
- Westermann, A. J., Gorski, S. A., Vogel, J. Dual RNA-seq of pathogen and host. Nature Reviews Microbiology. 10 (9), 618-630 (2012).

