Soya Fasulyesi Simbiyotik Nodüllerinden Polizom Saflaştırma
Summary
Bu protokol, bozulmamış soya fasulyesi nodüllerinden ökaryotik polizom saflaştırma için bir yöntemi açıklar. Dizilemeden sonra, transkriptom ve translatom seviyelerinde diferansiyel olarak eksprese edilen genleri tanımlamak için gen ekspresyon analizi için standart boru hatları kullanılabilir.
Abstract
Bu protokolün amacı, soya fasulyesi (Glycine max) simbiyotik nodülün ökaryotik translatomunu incelemek için bir strateji sağlamaktır. Bu yazıda, RNA dizilimi kullanılarak analiz edilecek bitki kaynaklı poliribozomları ve bunlarla ilişkili mRNA’ları izole etmek için optimize edilmiş yöntemler açıklanmaktadır. İlk olarak, sitoplazmik lizatlar, bütün, dondurulmuş soya fasulyesi nodüllerinden polizom ve RNA koruyucu koşullarda homojenizasyon yoluyla elde edilir. Daha sonra, lizatlar düşük hızlı santrifüjleme ile temizlenir ve süpernatantın% 15’i toplam RNA (TOTAL) izolasyonu için kullanılır. Kalan temizlenmiş lizat, iki katmanlı bir sakkaroz yastığı (% 12 ve% 33.5) aracılığıyla ultrasantrifüjleme yoluyla polizomları izole etmek için kullanılır. Polizom ile ilişkili mRNA (PAR), yeniden süspansiyondan sonra polisomal peletlerden saflaştırılır. Hem TOTAL hem de PAR, RNA-seks için dizileme kütüphanelerinin kalite standartlarını karşılamak için oldukça hassas kılcal elektroforez ile değerlendirilir. Bir aşağı akış uygulamasına örnek olarak, dizilemeden sonra, transkriptom ve translatom seviyelerinde diferansiyel olarak eksprese edilen genleri elde etmek için gen ekspresyon analizi için standart boru hatları kullanılabilir. Özetle, bu yöntem, RNA-sek ile kombinasyon halinde, simbiyotik nodül gibi karmaşık bir dokuda ökaryotik mRNA’ların translasyonel regülasyonunun incelenmesine izin verir.
Introduction
Soya fasulyesi (Glycine max) gibi baklagil bitkileri, rizobia adı verilen spesifik toprak bakterileri ile simbiyoz oluşturabilir. Bu karşılıklı ilişki, bitki kökleri üzerinde yeni organların, simbiyotik nodüllerin oluşumunu ortaya çıkarır. Nodüller, bakterileri barındıran bitki organlarıdır ve sitoplazması bakterioidler adı verilen özel bir rizobya formu ile kolonize edilen konakçı hücrelerden oluşur. Bu bakterioidler, atmosferik azotun (N2) amonyağa indirgenmesini katalize eder ve bu da karbonhidratlar karşılığında bitkiye aktarılır 1,2.
Bu azot sabitleyici simbiyoz, en iyi çalışılmış bitki-mikrop simbiyozlarından biri olmasına rağmen, farklı abiyotik stres koşullarına maruz kalan bitkilerin simbiyotik partnerleriyle etkileşimlerini nasıl modüle ettikleri ve bunun nodül metabolizmasını nasıl etkilediği gibi birçok yönün daha iyi anlaşılması gerekmektedir. Bu süreçler, nodül translatomunu (yani, aktif olarak çevrilen mesajcı RNA’ların [mRNA’lar] alt kümesini) analiz ederek daha iyi anlaşılabilir. Poliribozomlar veya polizomlar, translasyon3’ü incelemek için yaygın olarak kullanılan, mRNA ile ilişkili çoklu ribozomların kompleksleridir. Polizom profilleme yöntemi, polizomlarla ilişkili mRNA’ların analizinden oluşur ve çeşitli biyolojik süreçlerde meydana gelen gen ekspresyonunu kontrol eden posttranskripsiyonel mekanizmaları incelemek için başarıyla kullanılmıştır 4,5.
Tarihsel olarak, genom ekspresyon analizi öncelikle mRNA bolluğunu belirlemeye odaklanmıştır 6,7,8,9. Bununla birlikte, gen ekspresyonunun posttranskripsiyonel düzenlemesinin farklı aşamaları, özellikle de translasyon10,11,12 nedeniyle transkript ve protein seviyeleri arasında korelasyon eksikliği vardır. Ayrıca, transkriptom seviyesindeki değişiklikler ile translatom13 seviyesinde meydana gelenler arasında herhangi bir bağımlılık gözlenmemiştir. Çevrilmekte olan mRNA setinin doğrudan analizi, hücre gen ekspresyonunun (son noktası protein bolluğu olan) sadece mRNA seviyeleri analiz edildiğinde elde edilenden daha doğru ve eksiksiz bir ölçümünü sağlar14,15,16.
Bu protokol, bitki kaynaklı polizomların, iki katmanlı bir sakkaroz yastığı aracılığıyla diferansiyel santrifüjleme ile bozulmamış soya fasulyesi nodüllerinden nasıl saflaştırıldığını açıklar (Şekil 1). Bununla birlikte, nodüllerde bakterioid kaynaklı ribozomlar da bulunduğundan, ökaryotik olanlar ana fraksiyonu (% 90 -% 95) temsil etse de, ribozomların ve RNA türlerinin bir karışımı saflaştırılır. Sonraki RNA izolasyonu, nicelleştirme ve kalite kontrolü de tanımlanmıştır (Şekil 1). Bu protokol, RNA-sek ile kombinasyon halinde, simbiyotik nodül gibi karmaşık bir dokudaki ökaryotik mRNA’ların translasyonel regülasyonu hakkında deneysel sonuçlar sağlamalıdır.
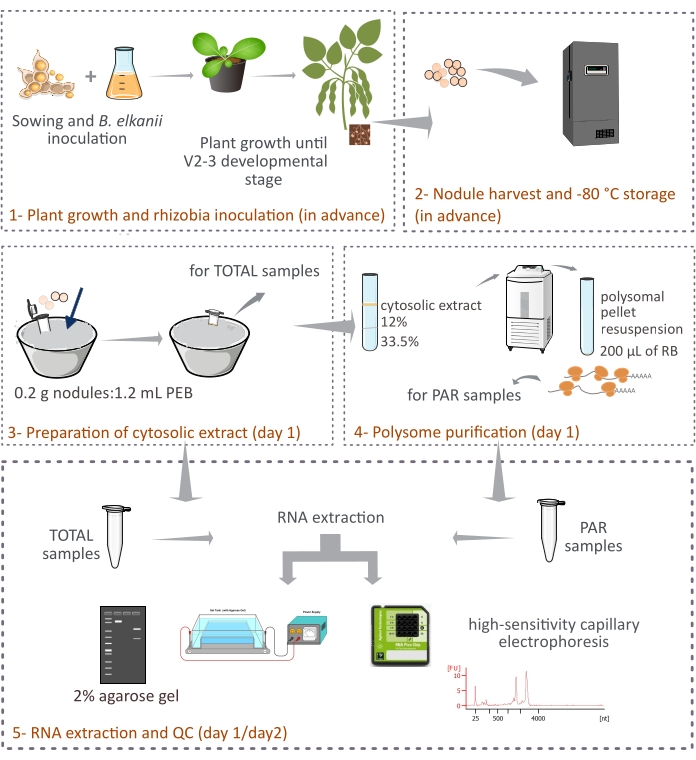
Şekil 1: Simbiyotik nodüllerden ökaryotik polizom saflaştırma için önerilen metodolojiye şematik genel bakış. Şema, (1) bitki büyümesi ve (2) nodül hasadından (3) sitozolik ekstraktların hazırlanmasına, (3) TOTAL numunelerinin ve (4) PAR numunelerinin elde edilmesine ve (5) RNA ekstraksiyonu ve kalite kontrolüne kadar protokolde izlenen adımlara genel bir bakış sunmaktadır. Kısaltmalar: PEB = polizom ekstraksiyon tamponu; RB = süspansiyon tamponu; TOPLAM = toplam RNA; PAR = polizomla ilişkili mRNA. Bu şeklin daha büyük bir versiyonunu görüntülemek için lütfen buraya tıklayın.
Protocol
Representative Results
Discussion
Gen ekspresyonu regülasyonunu translasyonel düzeyde incelemek, farklı biyolojik süreçleri daha iyi anlamak için kritik öneme sahiptir, çünkü hücre gen ekspresyonunun son noktası protein bolluğudur13,14. Bu, polisomal fraksiyonun saflaştırılması gereken ilgili doku veya organizmanın translatomunu analiz ederek ve ilişkili mRNA’larıanaliz ederek değerlendirilebilir 3,4,34,35,36.</s…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Bu araştırma CSIC I + D 2020 hibe No. 282, FVF 2017 hibe No. 210 ve PEDECIBA (María Martha Sainz) tarafından finanse edilmiştir.
Materials
| Plant growth and rhizobia inoculation | |||
| Orbital shaker | Daihan Scientific | Model SHO-1D | |
| YEM-medium | Amresco | J850 (yeast extract) 0122 (mannitol) | |
| Water deficit treatment | |||
| KNO3 | Merck | 221295 | |
| Porometer | Decagon Device | Model SC-1 | |
| Scalpel | |||
| Preparation of cytosolic extracts | |||
| Brij L23 | Sigma-Aldrich | P1254 | |
| Centrifuge | Sigma | Model 2K15 | |
| Chloranphenicol | Sigma-Aldrich | C0378 | |
| Cycloheximide | Sigma-Aldrich | C7698 | |
| DOC | Sigma-Aldrich | 30970 | |
| DTT | Sigma-Aldrich | D9779 | |
| EGTA | Sigma-Aldrich | E3889 | |
| Igepal CA 360 | Sigma-Aldrich | I8896 | |
| KCl | Merck | 1.04936 | |
| MgCl2 | Sigma-Aldrich | M8266 | |
| Plastic tissue grinder | Fisher Scientific | 12649595 | |
| PMSF | Sigma-Aldrich | P7626 | |
| PTE | Sigma-Aldrich | P2393 | |
| Tris | Invitrogen | 15504-020 | |
| Triton X-100 | Sigma-Aldrich | T8787 | |
| Tween 20 | Sigma-Aldrich | P1379 | |
| Weighing dish | Deltalab | 1911103 | |
| Preparation of sucrose cushions | |||
| Sucrose | Invitrogen | 15503022 | |
| SW 40 Ti rotor | Beckman-Coulter | ||
| Ultracentrifuge | Beckman-Coulter | Optima L-100K | |
| Ultracentrifuge tubes | Beckman-Coulter | 344059 | 13.2 mL tubes |
| RNA extraction and quality control | |||
| Agarose | Thermo scientific | R0492 | |
| Bioanalyzer | Agilent | Model 2100. Eukaryote total RNA nano assay | |
| Chloroform | DI | 41191 | |
| Ethanol | Dorwil | UN1170 | |
| Isopropanol | Mallinckrodt | 3032-06 | |
| Glycogen | Sigma | 10814-010 | |
| TRIzol LS | Ambion | 102960028 | |
| Miscellaneous | |||
| Falcon tubes 15 mL | Biologix | 10-0152 | |
| Filter tips 10 µL | BioPointe Scientific | 321-4050 | |
| Filter tips 1000 µL | BioPointe Scientific | 361-1050 | |
| Filter tips 20 µL | BioPointe Scientific | 341-4050 | |
| Filter tips 200 µL | Tarsons | 528104 | |
| Microcentrifuge tubes 1.5 mL | Tarsons | 500010-N | |
| Microcentrifuge tubes 2.0 mL | Tarsons | 500020-N | |
| Sequencing company | Macrogen | ||
| Sterile 250 mL flask | Marienfeld | 4110207 |
References
- Limpens, E., et al. Cell- and tissue-specific transcriptome analyses of Medicago truncatula root nodules. PLoS ONE. 8 (5), 64377 (2013).
- Masson-Boivin, C., Giraud, E., Perret, X., Batut, J. Establishing nitrogen-fixing symbiosis with legumes: How many rhizobium recipes. Trends in Microbiology. 17 (10), 458-466 (2009).
- King, H. A., Gerber, A. P. Translatome profiling: Methods for genome-scale analysis of mRNA translation. Briefings in Functional Genomics. 15 (1), 22-31 (2014).
- Chassé, H., Boulben, S., Costache, V., Cormier, P., Morales, J. Analysis of translation using polysome profiling. Nucleic Acids Research. 45 (3), 15 (2017).
- Yángüez, E., Castro-Sanz, A. B., Fernández-Bautista, N., Oliveros, J. C., Castellano, M. M. Analysis of genome-wide changes in the translatome of Arabidopsis seedlings subjected to heat stress. PloS ONE. 8 (8), 71425 (2013).
- Brown, P. O., Botstein, D. Exploring the new world of the genome with DNA microarrays. Nature Genetics. 21, 33-37 (1999).
- Krishnamurthy, A., Ferl, R. J., Paul, A. -. L. Comparing RNA-Seq and microarray gene expression data in two zones of the Arabidopsis root apex relevant to spaceflight. Applications in Plant Sciences. 6 (11), 1197 (2018).
- Shulse, C. N., et al. High-throughput single-cell transcriptome profiling of plant cell types. Cell Reports. 27 (7), 2241-2247 (2019).
- Wang, Z., Gerstein, M., Snyder, M. RNA-Seq: A revolutionary tool for transcriptomics. Nature Reviews Genetics. 10 (1), 57-63 (2009).
- Kawaguchi, R., Girke, T., Bray, E. A., Bailey-Serres, J. Differential mRNA translation contributes to gene regulation under non-stress and dehydration stress conditions in Arabidopsis thaliana. The Plant Journal. 38 (5), 823-839 (2004).
- Larsson, O., Tian, B., Sonenberg, N. Toward a genome-wide landscape of translational control. Cold Spring Harbor Perspectives in Biology. 5 (1), 012302 (2013).
- Vogel, C., Marcotte, E. M. Insights into regulation of protein abundance from proteomics and transcriptomis analyses. Nature Reviews Genetics. 13 (4), 227-232 (2013).
- Tebaldi, T., et al. Widespread uncoupling between transcriptome and translatome variations after a stimulus in mammalian cells. BMC Genomics. 13 (220), 1-15 (2012).
- Wang, T., et al. Translating mRNAs strongly correlate to proteins in a multivariate manner and their translation ratios are phenotype specific. Nucleic Acids Research. 41 (9), 4743-4754 (2013).
- Ingolia, N. T. Ribosome profiling: New views of translation, from single codons to genome scale. Nature Reviews Genetics. 15 (3), 205-213 (2014).
- Lukoszek, R., Feist, P., Ignatova, Z. Insights into the adaptive response of Arabidopsis thaliana to prolonged thermal stress by ribosomal profiling and RNA-Seq. BMC Plant Biology. 16 (1), 1-13 (2016).
- Broughton, W. J., Dilworth, M. J. Control of leghaemoglobin synthesis in snake beans. Biochemical Journal. 125 (4), 1075-1080 (1971).
- Schroeder, A., et al. The RIN: An RNA integrity number for assigning integrity values to RNA measurements. BMC Molecular Biology. 7, 1-14 (2006).
- Rio, D. C., Ares, M., Hannon, G. J., Nilsen, T. W. Nondenaturing agarose gel electrophoresis of RNA nondenaturing agarose gel electrophoresis of RNA. Cold Spring Harbor Protocols. 2010 (6), 1-3 (2012).
- Ewels, P., Magnusson, M., Lundin, S., Käller, M. MultiQC: Summarize analysis results for multiple tools and samples in a single report. Bioinformatics. 32 (19), 3047-3048 (2016).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: A flexible trimmer for Illumina sequence data. Bioinformatics. 30 (15), 2114-2120 (2014).
- Marcel, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet.journal. 17 (1), 10-12 (2011).
- . Sickle: A sliding-window, adaptive, quality-based trimming tool for FastQ files (Version 1.33) Available from: https://github.com/najoshi/sickle (2011)
- Langmead, B., Salzberg, S. L. Fast gapped-read alignment with Bowtie 2. Nature Methods. 9 (4), 357-359 (2012).
- Kim, D., et al. TopHat2: Accurate alignment of transcriptomes in the presence of insertions, deletions and gene fusions. Genome Biology. 14 (4), 1-13 (2013).
- Patro, R., Duggal, G., Love, M. I., Irizarry, R. A., Kingsford, C. Salmon provides fast and bias-aware quantification of transcript expression. Nature Methods. 14 (4), 417-419 (2017).
- Liao, Y., Smyth, G. K., Shi, W. featureCounts: An efficient general purpose program for assigning sequence reads to genomic features. Bioinformatics. 30 (7), 923-930 (2013).
- Robinson, M. D., McCarthy, D. J., Smyth, G. K. edgeR: A Bioconductor package for differential expression analysis of digital gene expression data. Bioinformatics. 26 (1), 139-140 (2010).
- Love, M. I., Huber, W., Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biology. 15 (550), 1-21 (2014).
- Ritchie, M. E., et al. limma powers differential expression analyses for RNA-sequencing and microarray studies. Nucleic Acids Research. 43 (7), 47 (2015).
- Afgan, E., et al. The Galaxy platform for accessible, reproducible and collaborative biomedical analyses: 2018 update. Nucleic Acids Research. 46, 537-544 (2018).
- Hunt, G. P., et al. GEOexplorer: A webserver for gene expression analysis and visualisation. Nucleic Acids Research. , (2022).
- Imbeaud, S., et al. Towards standardization of RNA quality assessment using user-independent classifiers of microcapillary electrophoresis traces. Nucleic Acids Research. 33 (6), 1-12 (2005).
- Di Paolo, A., et al. PDCD4 regulates axonal growth by translational repression of neurite growth-related genes and is modulated during nerve injury responses. RNA. 26 (11), 1637-1653 (2020).
- Smircich, P., et al. Ribosome profiling reveals translation control as a key mechanism generating differential gene expression in Trypanosoma cruzi. BMC Genomics. 16 (1), 443 (2015).
- Eastman, G., Sharlow, E. R., Lazo, J. S., Bloom, G. S., Sotelo-Silveira, J. R. Transcriptome and translatome regulation of pathogenesis in Alzheimer’s disease model mice. Journal of Alzheimer’s Disease. 86 (1), 365-386 (2022).
- Zanetti, M. E., Chang, I. -. F., Gong, F., Galbraith, D. W., Bailey-Serres, J. Immunopurification of polyribosomal complexes of Arabidopsis for global analysis of gene expression. Plant physiology. 138 (2), 624-635 (2005).
- Mustroph, A., et al. Profiling translatomes of discrete cell populations resolves altered cellular priorities during hypoxia in Arabidopsis. Proceedings of the National Academy of Sciences of the United States of America. 106 (44), 18843-11848 (2009).
- Ingolia, N. T., Ghaemmaghami, S., Newman, J. R. S., Weissman, J. S. Genome-wide analysis in vivo of translation with nucleotide resolution using ribosome profiling. Science. 324 (5924), 218-223 (2009).
- Brar, G. A., Weissman, J. S. Ribosome profiling reveals the what, when, where and how of protein synthesis. Nature Reviews. Molecular Cell Biology. 16 (11), 651-664 (2015).
- Eastman, G., Smircich, P., Sotelo-Silveira, J. R. Following ribosome footprints to understand translation at a genome wide level. Computational and Structural Biotechnology Journal. 16, 167-176 (2018).
- Westermann, A. J., Gorski, S. A., Vogel, J. Dual RNA-seq of pathogen and host. Nature Reviews Microbiology. 10 (9), 618-630 (2012).

