대두 공생 결절에서 폴리솜 정제
Summary
이 프로토콜은 온전한 대두 결절로부터 진핵 폴리솜 정제를 위한 방법을 기술한다. 시퀀싱 후, 유전자 발현 분석을 위한 표준 파이프라인을 사용하여 전사체 및 트랜스라톰 수준에서 차등적으로 발현된 유전자를 식별할 수 있습니다.
Abstract
이 프로토콜의 목적은 대두의 진핵생물 번역(Glycine max) 공생 결절을 연구하기 위한 전략을 제공하는 것입니다. 이 논문은 RNA 시퀀싱을 사용하여 분석할 식물 유래 폴리리보솜 및 관련 mRNA를 분리하는 데 최적화된 방법을 설명합니다. 첫째, 세포질 용해물은 전체 냉동 대두 결절로부터 폴리솜 및 RNA 보존 조건에서 균질화를 통해 얻어진다. 그런 다음 저속 원심분리에 의해 용해물을 제거하고 상청액의 15%를 총 RNA(TOTAL) 분리에 사용합니다. 나머지 투명화된 용해물은 2층 슈크로스 쿠션(12% 및 33.5%)을 통한 초원심분리에 의해 폴리좀을 분리하는 데 사용됩니다. 폴리솜 관련 mRNA (PAR)는 재현탁 후 폴리솜 펠릿으로부터 정제된다. TOTAL과 PAR은 모두 RNA-seq에 대한 시퀀싱 라이브러리의 품질 표준을 충족하기 위해 고감도 모세관 전기영동으로 평가됩니다. 다운스트림 적용의 예로서, 시퀀싱 후, 유전자 발현 분석을 위한 표준 파이프라인을 사용하여 전사체 및 트랜스플라톰 수준에서 차등적으로 발현된 유전자를 수득할 수 있다. 요약하면,이 방법은 RNA-seq와 함께 공생 결절과 같은 복잡한 조직에서 진핵 mRNA의 번역 조절을 연구 할 수있게합니다.
Introduction
콩 (Glycine max)과 같은 콩과 식물은 뿌리 줄기라고하는 특정 토양 박테리아와 공생 할 수 있습니다. 이 상호 주의적 관계는 식물 뿌리에 새로운 기관인 공생 결절의 형성을 유도합니다. 결절은 박테리아를 숙주하는 식물 기관이며 세포질이 박테로이드라는 특수한 형태의 뿌리 줄기로 식민지화 된 숙주 세포로 구성됩니다. 이 박테로이드는 대기 질소 (N 2)의 암모니아로의 환원을 촉매하며, 암모니아는 탄수화물 1,2에 대한 대가로 식물로 옮겨집니다.
이 질소 고정 공생은 가장 잘 연구 된 식물-미생물 공생 중 하나이지만, 다양한 비 생물 적 스트레스 조건을받는 식물이 공생 파트너와의 상호 작용을 조절하는 방법과 이것이 결절 대사에 미치는 영향과 같은 많은 측면이 더 잘 이해되어야합니다. 이러한 과정은 결절 번역체 (즉, 활발히 번역 된 메신저 RNA [mRNA]의 하위 집합)를 분석함으로써 더 잘 이해 될 수있다. 폴리리보솜 또는 폴리솜은 mRNA와 관련된 여러 리보솜의 복합체로, 일반적으로 번역3을 연구하는 데 사용됩니다. 폴리솜 프로파일링 방법은 폴리솜과 관련된 mRNA의 분석으로 구성되며 다양한생물학적 과정에서 발생하는 유전자 발현을 제어하는 전사 후 메커니즘을 연구하는 데 성공적으로 사용되었습니다4,5.
역사적으로 게놈 발현 분석은 주로 mRNA 풍부도 6,7,8,9를 결정하는 데 중점을 두었습니다. 그러나, 유전자 발현, 특히 번역10,11,12의 전사 후 조절의 상이한 단계로 인해 전사체와 단백질 수준 사이의 상관 관계가 부족하다. 더욱이, 전사체 수준에서의 변화와 트랜스 라톰13의 수준에서 발생하는 변화 사이에는 의존성이 관찰되지 않았다. 번역되는 mRNA 세트의 직접 분석을 통해 mRNA 수준 만 분석 할 때 얻은 것보다 세포 유전자 발현 (종말점이 단백질 풍부도)을보다 정확하고 완벽하게 측정 할 수 있습니다 14,15,16.
이 프로토콜은 식물 유래 폴리솜이 2층 자당 쿠션을 통한 차등 원심분리를 통해 온전한 대두 결절에서 정제되는 방법을 설명합니다(그림 1). 그러나 박테로이드 유래 리보솜도 결절에 존재하기 때문에 진핵 생물이 주요 분획 (90 % -95 %)을 나타내더라도 리보솜과 RNA 종의 혼합이 정제됩니다. 후속 RNA 분리, 정량화 및 품질 관리도 설명합니다(그림 1). 이 프로토콜은 RNA-seq와 함께 공생 결절과 같은 복잡한 조직에서 진핵 mRNA의 번역 조절에 대한 실험 결과를 제공해야합니다.
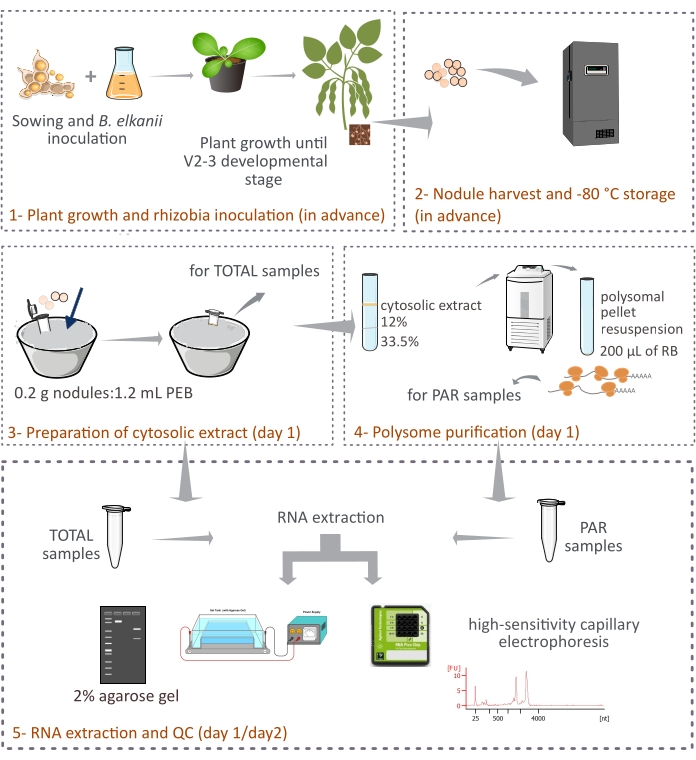
그림 1: 공생 결절로부터 진핵 폴리솜 정제를 위해 제안된 방법론의 개략적인 개요. 이 계획은 (1) 식물 성장 및 (2) 결절 수확에서 (3) 세포질 추출물의 준비, (3) TOTAL 샘플 및 (4) PAR 샘플 획득, (5) RNA 추출 및 품질 관리에 이르기까지 프로토콜에서 따르는 단계에 대한 개요를 제공합니다. 약어: PEB = 폴리솜 추출 완충액; RB = 재현탁 완충액; 합계 = 총 RNA; PAR = 폴리솜 관련 mRNA. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
Protocol
Representative Results
Discussion
번역 수준에서 유전자 발현 조절을 연구하는 것은 세포 유전자 발현의 종말점이 단백질 풍부도이기 때문에 다양한 생물학적 과정을 더 잘 이해하는 데 중요합니다13,14. 이것은 다염색체 분획을 정제해야하는 관심 조직 또는 유기체의 트랜스 라톰을 분석하고 관련 mRNA를 분석 한 3,4,34,35,36을 분석하여 평가할 수 있습니다.<sup cla…
Disclosures
The authors have nothing to disclose.
Acknowledgements
이 연구는 CSIC I+D 2020 보조금 번호 282, FVF 2017 보조금 번호 210 및 PEDECIBA(마리아 마사 사인즈)의 자금 지원을 받았습니다.
Materials
| Plant growth and rhizobia inoculation | |||
| Orbital shaker | Daihan Scientific | Model SHO-1D | |
| YEM-medium | Amresco | J850 (yeast extract) 0122 (mannitol) | |
| Water deficit treatment | |||
| KNO3 | Merck | 221295 | |
| Porometer | Decagon Device | Model SC-1 | |
| Scalpel | |||
| Preparation of cytosolic extracts | |||
| Brij L23 | Sigma-Aldrich | P1254 | |
| Centrifuge | Sigma | Model 2K15 | |
| Chloranphenicol | Sigma-Aldrich | C0378 | |
| Cycloheximide | Sigma-Aldrich | C7698 | |
| DOC | Sigma-Aldrich | 30970 | |
| DTT | Sigma-Aldrich | D9779 | |
| EGTA | Sigma-Aldrich | E3889 | |
| Igepal CA 360 | Sigma-Aldrich | I8896 | |
| KCl | Merck | 1.04936 | |
| MgCl2 | Sigma-Aldrich | M8266 | |
| Plastic tissue grinder | Fisher Scientific | 12649595 | |
| PMSF | Sigma-Aldrich | P7626 | |
| PTE | Sigma-Aldrich | P2393 | |
| Tris | Invitrogen | 15504-020 | |
| Triton X-100 | Sigma-Aldrich | T8787 | |
| Tween 20 | Sigma-Aldrich | P1379 | |
| Weighing dish | Deltalab | 1911103 | |
| Preparation of sucrose cushions | |||
| Sucrose | Invitrogen | 15503022 | |
| SW 40 Ti rotor | Beckman-Coulter | ||
| Ultracentrifuge | Beckman-Coulter | Optima L-100K | |
| Ultracentrifuge tubes | Beckman-Coulter | 344059 | 13.2 mL tubes |
| RNA extraction and quality control | |||
| Agarose | Thermo scientific | R0492 | |
| Bioanalyzer | Agilent | Model 2100. Eukaryote total RNA nano assay | |
| Chloroform | DI | 41191 | |
| Ethanol | Dorwil | UN1170 | |
| Isopropanol | Mallinckrodt | 3032-06 | |
| Glycogen | Sigma | 10814-010 | |
| TRIzol LS | Ambion | 102960028 | |
| Miscellaneous | |||
| Falcon tubes 15 mL | Biologix | 10-0152 | |
| Filter tips 10 µL | BioPointe Scientific | 321-4050 | |
| Filter tips 1000 µL | BioPointe Scientific | 361-1050 | |
| Filter tips 20 µL | BioPointe Scientific | 341-4050 | |
| Filter tips 200 µL | Tarsons | 528104 | |
| Microcentrifuge tubes 1.5 mL | Tarsons | 500010-N | |
| Microcentrifuge tubes 2.0 mL | Tarsons | 500020-N | |
| Sequencing company | Macrogen | ||
| Sterile 250 mL flask | Marienfeld | 4110207 |
References
- Limpens, E., et al. Cell- and tissue-specific transcriptome analyses of Medicago truncatula root nodules. PLoS ONE. 8 (5), 64377 (2013).
- Masson-Boivin, C., Giraud, E., Perret, X., Batut, J. Establishing nitrogen-fixing symbiosis with legumes: How many rhizobium recipes. Trends in Microbiology. 17 (10), 458-466 (2009).
- King, H. A., Gerber, A. P. Translatome profiling: Methods for genome-scale analysis of mRNA translation. Briefings in Functional Genomics. 15 (1), 22-31 (2014).
- Chassé, H., Boulben, S., Costache, V., Cormier, P., Morales, J. Analysis of translation using polysome profiling. Nucleic Acids Research. 45 (3), 15 (2017).
- Yángüez, E., Castro-Sanz, A. B., Fernández-Bautista, N., Oliveros, J. C., Castellano, M. M. Analysis of genome-wide changes in the translatome of Arabidopsis seedlings subjected to heat stress. PloS ONE. 8 (8), 71425 (2013).
- Brown, P. O., Botstein, D. Exploring the new world of the genome with DNA microarrays. Nature Genetics. 21, 33-37 (1999).
- Krishnamurthy, A., Ferl, R. J., Paul, A. -. L. Comparing RNA-Seq and microarray gene expression data in two zones of the Arabidopsis root apex relevant to spaceflight. Applications in Plant Sciences. 6 (11), 1197 (2018).
- Shulse, C. N., et al. High-throughput single-cell transcriptome profiling of plant cell types. Cell Reports. 27 (7), 2241-2247 (2019).
- Wang, Z., Gerstein, M., Snyder, M. RNA-Seq: A revolutionary tool for transcriptomics. Nature Reviews Genetics. 10 (1), 57-63 (2009).
- Kawaguchi, R., Girke, T., Bray, E. A., Bailey-Serres, J. Differential mRNA translation contributes to gene regulation under non-stress and dehydration stress conditions in Arabidopsis thaliana. The Plant Journal. 38 (5), 823-839 (2004).
- Larsson, O., Tian, B., Sonenberg, N. Toward a genome-wide landscape of translational control. Cold Spring Harbor Perspectives in Biology. 5 (1), 012302 (2013).
- Vogel, C., Marcotte, E. M. Insights into regulation of protein abundance from proteomics and transcriptomis analyses. Nature Reviews Genetics. 13 (4), 227-232 (2013).
- Tebaldi, T., et al. Widespread uncoupling between transcriptome and translatome variations after a stimulus in mammalian cells. BMC Genomics. 13 (220), 1-15 (2012).
- Wang, T., et al. Translating mRNAs strongly correlate to proteins in a multivariate manner and their translation ratios are phenotype specific. Nucleic Acids Research. 41 (9), 4743-4754 (2013).
- Ingolia, N. T. Ribosome profiling: New views of translation, from single codons to genome scale. Nature Reviews Genetics. 15 (3), 205-213 (2014).
- Lukoszek, R., Feist, P., Ignatova, Z. Insights into the adaptive response of Arabidopsis thaliana to prolonged thermal stress by ribosomal profiling and RNA-Seq. BMC Plant Biology. 16 (1), 1-13 (2016).
- Broughton, W. J., Dilworth, M. J. Control of leghaemoglobin synthesis in snake beans. Biochemical Journal. 125 (4), 1075-1080 (1971).
- Schroeder, A., et al. The RIN: An RNA integrity number for assigning integrity values to RNA measurements. BMC Molecular Biology. 7, 1-14 (2006).
- Rio, D. C., Ares, M., Hannon, G. J., Nilsen, T. W. Nondenaturing agarose gel electrophoresis of RNA nondenaturing agarose gel electrophoresis of RNA. Cold Spring Harbor Protocols. 2010 (6), 1-3 (2012).
- Ewels, P., Magnusson, M., Lundin, S., Käller, M. MultiQC: Summarize analysis results for multiple tools and samples in a single report. Bioinformatics. 32 (19), 3047-3048 (2016).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: A flexible trimmer for Illumina sequence data. Bioinformatics. 30 (15), 2114-2120 (2014).
- Marcel, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet.journal. 17 (1), 10-12 (2011).
- . Sickle: A sliding-window, adaptive, quality-based trimming tool for FastQ files (Version 1.33) Available from: https://github.com/najoshi/sickle (2011)
- Langmead, B., Salzberg, S. L. Fast gapped-read alignment with Bowtie 2. Nature Methods. 9 (4), 357-359 (2012).
- Kim, D., et al. TopHat2: Accurate alignment of transcriptomes in the presence of insertions, deletions and gene fusions. Genome Biology. 14 (4), 1-13 (2013).
- Patro, R., Duggal, G., Love, M. I., Irizarry, R. A., Kingsford, C. Salmon provides fast and bias-aware quantification of transcript expression. Nature Methods. 14 (4), 417-419 (2017).
- Liao, Y., Smyth, G. K., Shi, W. featureCounts: An efficient general purpose program for assigning sequence reads to genomic features. Bioinformatics. 30 (7), 923-930 (2013).
- Robinson, M. D., McCarthy, D. J., Smyth, G. K. edgeR: A Bioconductor package for differential expression analysis of digital gene expression data. Bioinformatics. 26 (1), 139-140 (2010).
- Love, M. I., Huber, W., Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biology. 15 (550), 1-21 (2014).
- Ritchie, M. E., et al. limma powers differential expression analyses for RNA-sequencing and microarray studies. Nucleic Acids Research. 43 (7), 47 (2015).
- Afgan, E., et al. The Galaxy platform for accessible, reproducible and collaborative biomedical analyses: 2018 update. Nucleic Acids Research. 46, 537-544 (2018).
- Hunt, G. P., et al. GEOexplorer: A webserver for gene expression analysis and visualisation. Nucleic Acids Research. , (2022).
- Imbeaud, S., et al. Towards standardization of RNA quality assessment using user-independent classifiers of microcapillary electrophoresis traces. Nucleic Acids Research. 33 (6), 1-12 (2005).
- Di Paolo, A., et al. PDCD4 regulates axonal growth by translational repression of neurite growth-related genes and is modulated during nerve injury responses. RNA. 26 (11), 1637-1653 (2020).
- Smircich, P., et al. Ribosome profiling reveals translation control as a key mechanism generating differential gene expression in Trypanosoma cruzi. BMC Genomics. 16 (1), 443 (2015).
- Eastman, G., Sharlow, E. R., Lazo, J. S., Bloom, G. S., Sotelo-Silveira, J. R. Transcriptome and translatome regulation of pathogenesis in Alzheimer’s disease model mice. Journal of Alzheimer’s Disease. 86 (1), 365-386 (2022).
- Zanetti, M. E., Chang, I. -. F., Gong, F., Galbraith, D. W., Bailey-Serres, J. Immunopurification of polyribosomal complexes of Arabidopsis for global analysis of gene expression. Plant physiology. 138 (2), 624-635 (2005).
- Mustroph, A., et al. Profiling translatomes of discrete cell populations resolves altered cellular priorities during hypoxia in Arabidopsis. Proceedings of the National Academy of Sciences of the United States of America. 106 (44), 18843-11848 (2009).
- Ingolia, N. T., Ghaemmaghami, S., Newman, J. R. S., Weissman, J. S. Genome-wide analysis in vivo of translation with nucleotide resolution using ribosome profiling. Science. 324 (5924), 218-223 (2009).
- Brar, G. A., Weissman, J. S. Ribosome profiling reveals the what, when, where and how of protein synthesis. Nature Reviews. Molecular Cell Biology. 16 (11), 651-664 (2015).
- Eastman, G., Smircich, P., Sotelo-Silveira, J. R. Following ribosome footprints to understand translation at a genome wide level. Computational and Structural Biotechnology Journal. 16, 167-176 (2018).
- Westermann, A. J., Gorski, S. A., Vogel, J. Dual RNA-seq of pathogen and host. Nature Reviews Microbiology. 10 (9), 618-630 (2012).

