Polysom rensing fra soyabønner Symbiotic Nodules
Summary
Denne protokollen beskriver en metode for eukaryotisk polysomrensing fra intakte soyabønneknuter. Etter sekvensering kan standard rørledninger for genuttrykksanalyse brukes til å identifisere differensielt uttrykte gener på transkriptom- og translatomnivå.
Abstract
Målet med denne protokollen er å gi en strategi for å studere eukaryotisk translatom av soyabønner (Glycine max) symbiotisk knute. Dette papiret beskriver metoder optimalisert for å isolere planteavledede polyribosomer og deres tilhørende mRNA som skal analyseres ved hjelp av RNA-sekvensering. Først oppnås cytoplasmatiske lysater gjennom homogenisering i polysom- og RNA-bevarende forhold fra hele, frosne soyabønneknuter. Deretter fjernes lysater ved lavhastighets sentrifugering, og 15% av supernatanten brukes til total RNA (TOTAL) isolasjon. Det gjenværende klarerte lysatet brukes til å isolere polysomer ved ultrasentrifugering gjennom en tolags sukrosepute (12% og 33,5%). Polysomassosiert mRNA (PAR) renses fra polysomale pellets etter resuspendering. Både TOTAL og PAR evalueres ved høysensitiv kapillærelektroforese for å oppfylle kvalitetsstandardene for sekvenseringsbiblioteker for RNA-seq. Som et eksempel på en nedstrøms applikasjon, etter sekvensering, kan standard rørledninger for genuttrykksanalyse brukes til å oppnå differensielt uttrykte gener på transkriptom- og translatomnivå. Oppsummert tillater denne metoden, i kombinasjon med RNA-seq, studiet av translasjonsreguleringen av eukaryote mRNA i et komplekst vev som den symbiotiske knuten.
Introduction
Leguminøse planter, som soyabønne (Glycine max), kan etablere symbiose med spesifikke jordbakterier kalt rhizobia. Dette mutualistiske forholdet fremkaller dannelsen av nye organer, de symbiotiske knutene, på planterøttene. Knutene er planteorganene som er vert for bakteriene og består av vertsceller hvis cytoplasma er kolonisert med en spesialisert form for rhizobia kalt bakterier. Disse bakterieoidene katalyserer reduksjonen av atmosfærisk nitrogen (N 2) til ammoniakk, som overføres til planten i retur for karbohydrater 1,2.
Selv om denne nitrogenfikserende symbiosen er en av de mest studerte plantemikrobesymbiosene, gjenstår mange aspekter å bli bedre forstått, for eksempel hvordan planter utsatt for forskjellige abiotiske stressforhold modulerer samspillet med deres symbiotiske partner og hvordan dette påvirker knutemetabolismen. Disse prosessene kan forstås bedre ved å analysere knutetranslatomet (dvs. delmengden av messenger-RNA [mRNA] aktivt oversatt). Polyribosomer eller polysomer er komplekser av flere ribosomer assosiert med mRNA, som vanligvis brukes til å studere oversettelse3. Polysomprofileringsmetoden består av analysen av mRNA assosiert med polysomer og har blitt brukt til å studere posttranskripsjonsmekanismer som styrer genuttrykk som forekommer i ulike biologiske prosesser 4,5.
Historisk sett har genomuttrykksanalyse primært fokusert på å bestemme mRNA-overflod 6,7,8,9. Imidlertid er det mangel på korrelasjon mellom transkripsjon og proteinnivåer på grunn av de forskjellige stadiene av posttranskripsjonell regulering av genuttrykk, spesielt oversettelse10,11,12. Videre er det ikke observert avhengighet mellom endringene på transkriptomnivået og de som forekommer på translatomnivået13. Den direkte analysen av settet av mRNA som oversettes, tillater en mer nøyaktig og fullstendig måling av cellegenuttrykket (hvis endepunkt er proteinoverflod) enn det som oppnås når bare mRNA-nivåer analyseres14,15,16.
Denne protokollen beskriver hvordan planteavledede polysomer renses fra intakte soyabønneknuter ved differensiell sentrifugering gjennom en tolags sukrosepute (figur 1). Men siden bakterieformede ribosomer også er tilstede i knutene, blir en blanding av ribosomer og RNA-arter renset, selv om de eukaryote representerer hovedfraksjonen (90% -95%). Den påfølgende RNA-isolasjonen, kvantifiseringen og kvalitetskontrollen er også beskrevet (figur 1). Denne protokollen, i kombinasjon med RNA-seq, skal gi eksperimentelle resultater på translasjonsregulering av eukaryote mRNA i et komplekst vev som den symbiotiske knuten.
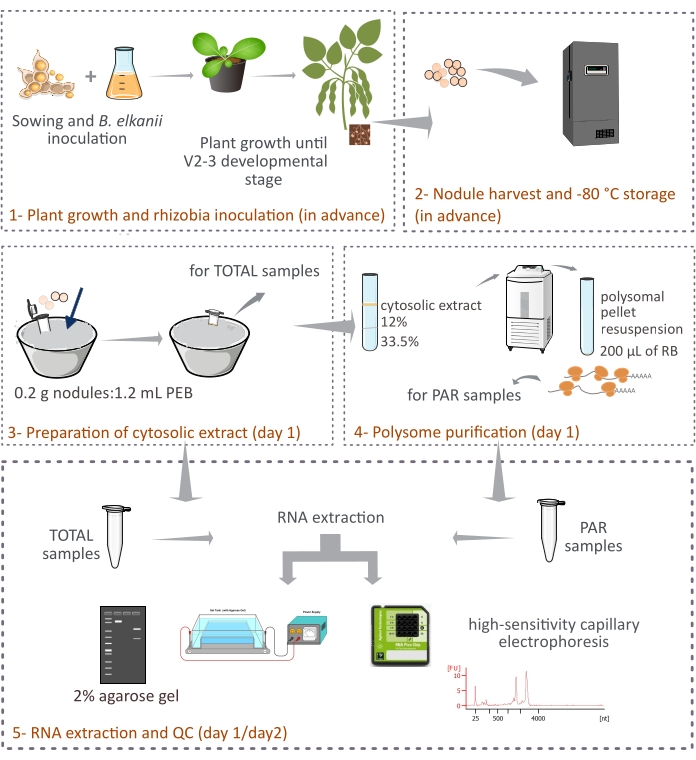
Figur 1: Skjematisk oversikt over foreslått metodikk for eukaryotisk polysomrensing fra symbiotiske knuter. Ordningen gir en oversikt over trinnene som følges i protokollen fra (1) plantevekst og (2) knutehøsting til (3) fremstilling av cytosoliske ekstrakter, (3) innhenting av TOTAL-prøver og (4) PAR-prøver, og (5) RNA-ekstraksjon og kvalitetskontroll. Forkortelser: PEB = polysomekstraksjonsbuffer; RB = resuspension buffer; TOTALT = totalt RNA; PAR = polysomassosiert mRNA. Klikk her for å se en større versjon av dette tallet.
Protocol
Representative Results
Discussion
Å studere regulering av genuttrykk på translasjonsnivå er avgjørende for å bedre forstå forskjellige biologiske prosesser siden endepunktet for cellegenuttrykk er proteinoverflod13,14. Dette kan vurderes ved å analysere translatomet av vevet eller organismen av interesse som den polysomale fraksjonen skal renses for og tilhørende mRNA analyseres 3,4,34,35,36.<sup…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Denne forskningen ble finansiert av CSIC I + D 2020-stipend nr. 282, FVF 2017-stipend nr. 210 og PEDECIBA (María Martha Sainz).
Materials
| Plant growth and rhizobia inoculation | |||
| Orbital shaker | Daihan Scientific | Model SHO-1D | |
| YEM-medium | Amresco | J850 (yeast extract) 0122 (mannitol) | |
| Water deficit treatment | |||
| KNO3 | Merck | 221295 | |
| Porometer | Decagon Device | Model SC-1 | |
| Scalpel | |||
| Preparation of cytosolic extracts | |||
| Brij L23 | Sigma-Aldrich | P1254 | |
| Centrifuge | Sigma | Model 2K15 | |
| Chloranphenicol | Sigma-Aldrich | C0378 | |
| Cycloheximide | Sigma-Aldrich | C7698 | |
| DOC | Sigma-Aldrich | 30970 | |
| DTT | Sigma-Aldrich | D9779 | |
| EGTA | Sigma-Aldrich | E3889 | |
| Igepal CA 360 | Sigma-Aldrich | I8896 | |
| KCl | Merck | 1.04936 | |
| MgCl2 | Sigma-Aldrich | M8266 | |
| Plastic tissue grinder | Fisher Scientific | 12649595 | |
| PMSF | Sigma-Aldrich | P7626 | |
| PTE | Sigma-Aldrich | P2393 | |
| Tris | Invitrogen | 15504-020 | |
| Triton X-100 | Sigma-Aldrich | T8787 | |
| Tween 20 | Sigma-Aldrich | P1379 | |
| Weighing dish | Deltalab | 1911103 | |
| Preparation of sucrose cushions | |||
| Sucrose | Invitrogen | 15503022 | |
| SW 40 Ti rotor | Beckman-Coulter | ||
| Ultracentrifuge | Beckman-Coulter | Optima L-100K | |
| Ultracentrifuge tubes | Beckman-Coulter | 344059 | 13.2 mL tubes |
| RNA extraction and quality control | |||
| Agarose | Thermo scientific | R0492 | |
| Bioanalyzer | Agilent | Model 2100. Eukaryote total RNA nano assay | |
| Chloroform | DI | 41191 | |
| Ethanol | Dorwil | UN1170 | |
| Isopropanol | Mallinckrodt | 3032-06 | |
| Glycogen | Sigma | 10814-010 | |
| TRIzol LS | Ambion | 102960028 | |
| Miscellaneous | |||
| Falcon tubes 15 mL | Biologix | 10-0152 | |
| Filter tips 10 µL | BioPointe Scientific | 321-4050 | |
| Filter tips 1000 µL | BioPointe Scientific | 361-1050 | |
| Filter tips 20 µL | BioPointe Scientific | 341-4050 | |
| Filter tips 200 µL | Tarsons | 528104 | |
| Microcentrifuge tubes 1.5 mL | Tarsons | 500010-N | |
| Microcentrifuge tubes 2.0 mL | Tarsons | 500020-N | |
| Sequencing company | Macrogen | ||
| Sterile 250 mL flask | Marienfeld | 4110207 |
References
- Limpens, E., et al. Cell- and tissue-specific transcriptome analyses of Medicago truncatula root nodules. PLoS ONE. 8 (5), 64377 (2013).
- Masson-Boivin, C., Giraud, E., Perret, X., Batut, J. Establishing nitrogen-fixing symbiosis with legumes: How many rhizobium recipes. Trends in Microbiology. 17 (10), 458-466 (2009).
- King, H. A., Gerber, A. P. Translatome profiling: Methods for genome-scale analysis of mRNA translation. Briefings in Functional Genomics. 15 (1), 22-31 (2014).
- Chassé, H., Boulben, S., Costache, V., Cormier, P., Morales, J. Analysis of translation using polysome profiling. Nucleic Acids Research. 45 (3), 15 (2017).
- Yángüez, E., Castro-Sanz, A. B., Fernández-Bautista, N., Oliveros, J. C., Castellano, M. M. Analysis of genome-wide changes in the translatome of Arabidopsis seedlings subjected to heat stress. PloS ONE. 8 (8), 71425 (2013).
- Brown, P. O., Botstein, D. Exploring the new world of the genome with DNA microarrays. Nature Genetics. 21, 33-37 (1999).
- Krishnamurthy, A., Ferl, R. J., Paul, A. -. L. Comparing RNA-Seq and microarray gene expression data in two zones of the Arabidopsis root apex relevant to spaceflight. Applications in Plant Sciences. 6 (11), 1197 (2018).
- Shulse, C. N., et al. High-throughput single-cell transcriptome profiling of plant cell types. Cell Reports. 27 (7), 2241-2247 (2019).
- Wang, Z., Gerstein, M., Snyder, M. RNA-Seq: A revolutionary tool for transcriptomics. Nature Reviews Genetics. 10 (1), 57-63 (2009).
- Kawaguchi, R., Girke, T., Bray, E. A., Bailey-Serres, J. Differential mRNA translation contributes to gene regulation under non-stress and dehydration stress conditions in Arabidopsis thaliana. The Plant Journal. 38 (5), 823-839 (2004).
- Larsson, O., Tian, B., Sonenberg, N. Toward a genome-wide landscape of translational control. Cold Spring Harbor Perspectives in Biology. 5 (1), 012302 (2013).
- Vogel, C., Marcotte, E. M. Insights into regulation of protein abundance from proteomics and transcriptomis analyses. Nature Reviews Genetics. 13 (4), 227-232 (2013).
- Tebaldi, T., et al. Widespread uncoupling between transcriptome and translatome variations after a stimulus in mammalian cells. BMC Genomics. 13 (220), 1-15 (2012).
- Wang, T., et al. Translating mRNAs strongly correlate to proteins in a multivariate manner and their translation ratios are phenotype specific. Nucleic Acids Research. 41 (9), 4743-4754 (2013).
- Ingolia, N. T. Ribosome profiling: New views of translation, from single codons to genome scale. Nature Reviews Genetics. 15 (3), 205-213 (2014).
- Lukoszek, R., Feist, P., Ignatova, Z. Insights into the adaptive response of Arabidopsis thaliana to prolonged thermal stress by ribosomal profiling and RNA-Seq. BMC Plant Biology. 16 (1), 1-13 (2016).
- Broughton, W. J., Dilworth, M. J. Control of leghaemoglobin synthesis in snake beans. Biochemical Journal. 125 (4), 1075-1080 (1971).
- Schroeder, A., et al. The RIN: An RNA integrity number for assigning integrity values to RNA measurements. BMC Molecular Biology. 7, 1-14 (2006).
- Rio, D. C., Ares, M., Hannon, G. J., Nilsen, T. W. Nondenaturing agarose gel electrophoresis of RNA nondenaturing agarose gel electrophoresis of RNA. Cold Spring Harbor Protocols. 2010 (6), 1-3 (2012).
- Ewels, P., Magnusson, M., Lundin, S., Käller, M. MultiQC: Summarize analysis results for multiple tools and samples in a single report. Bioinformatics. 32 (19), 3047-3048 (2016).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: A flexible trimmer for Illumina sequence data. Bioinformatics. 30 (15), 2114-2120 (2014).
- Marcel, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet.journal. 17 (1), 10-12 (2011).
- . Sickle: A sliding-window, adaptive, quality-based trimming tool for FastQ files (Version 1.33) Available from: https://github.com/najoshi/sickle (2011)
- Langmead, B., Salzberg, S. L. Fast gapped-read alignment with Bowtie 2. Nature Methods. 9 (4), 357-359 (2012).
- Kim, D., et al. TopHat2: Accurate alignment of transcriptomes in the presence of insertions, deletions and gene fusions. Genome Biology. 14 (4), 1-13 (2013).
- Patro, R., Duggal, G., Love, M. I., Irizarry, R. A., Kingsford, C. Salmon provides fast and bias-aware quantification of transcript expression. Nature Methods. 14 (4), 417-419 (2017).
- Liao, Y., Smyth, G. K., Shi, W. featureCounts: An efficient general purpose program for assigning sequence reads to genomic features. Bioinformatics. 30 (7), 923-930 (2013).
- Robinson, M. D., McCarthy, D. J., Smyth, G. K. edgeR: A Bioconductor package for differential expression analysis of digital gene expression data. Bioinformatics. 26 (1), 139-140 (2010).
- Love, M. I., Huber, W., Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biology. 15 (550), 1-21 (2014).
- Ritchie, M. E., et al. limma powers differential expression analyses for RNA-sequencing and microarray studies. Nucleic Acids Research. 43 (7), 47 (2015).
- Afgan, E., et al. The Galaxy platform for accessible, reproducible and collaborative biomedical analyses: 2018 update. Nucleic Acids Research. 46, 537-544 (2018).
- Hunt, G. P., et al. GEOexplorer: A webserver for gene expression analysis and visualisation. Nucleic Acids Research. , (2022).
- Imbeaud, S., et al. Towards standardization of RNA quality assessment using user-independent classifiers of microcapillary electrophoresis traces. Nucleic Acids Research. 33 (6), 1-12 (2005).
- Di Paolo, A., et al. PDCD4 regulates axonal growth by translational repression of neurite growth-related genes and is modulated during nerve injury responses. RNA. 26 (11), 1637-1653 (2020).
- Smircich, P., et al. Ribosome profiling reveals translation control as a key mechanism generating differential gene expression in Trypanosoma cruzi. BMC Genomics. 16 (1), 443 (2015).
- Eastman, G., Sharlow, E. R., Lazo, J. S., Bloom, G. S., Sotelo-Silveira, J. R. Transcriptome and translatome regulation of pathogenesis in Alzheimer’s disease model mice. Journal of Alzheimer’s Disease. 86 (1), 365-386 (2022).
- Zanetti, M. E., Chang, I. -. F., Gong, F., Galbraith, D. W., Bailey-Serres, J. Immunopurification of polyribosomal complexes of Arabidopsis for global analysis of gene expression. Plant physiology. 138 (2), 624-635 (2005).
- Mustroph, A., et al. Profiling translatomes of discrete cell populations resolves altered cellular priorities during hypoxia in Arabidopsis. Proceedings of the National Academy of Sciences of the United States of America. 106 (44), 18843-11848 (2009).
- Ingolia, N. T., Ghaemmaghami, S., Newman, J. R. S., Weissman, J. S. Genome-wide analysis in vivo of translation with nucleotide resolution using ribosome profiling. Science. 324 (5924), 218-223 (2009).
- Brar, G. A., Weissman, J. S. Ribosome profiling reveals the what, when, where and how of protein synthesis. Nature Reviews. Molecular Cell Biology. 16 (11), 651-664 (2015).
- Eastman, G., Smircich, P., Sotelo-Silveira, J. R. Following ribosome footprints to understand translation at a genome wide level. Computational and Structural Biotechnology Journal. 16, 167-176 (2018).
- Westermann, A. J., Gorski, S. A., Vogel, J. Dual RNA-seq of pathogen and host. Nature Reviews Microbiology. 10 (9), 618-630 (2012).

