Polysomrening från sojabönor Symbiotic Nodules
Summary
Detta protokoll beskriver en metod för eukaryot polysomrening från intakta sojabönor. Efter sekvensering kan standardpipelines för genuttrycksanalys användas för att identifiera differentiellt uttryckta gener på transkriptom- och translatomnivåerna.
Abstract
Syftet med detta protokoll är att tillhandahålla en strategi för att studera det eukaryota överlatomet av sojabönan (Glycine max) symbiotisk knöl. Detta dokument beskriver metoder optimerade för att isolera växtbaserade polyribosomer och deras associerade mRNA som ska analyseras med RNA-sekvensering. Först erhålls cytoplasmatiska lysater genom homogenisering i polysom- och RNA-bevarande förhållanden från hela, frysta sojabönknutor. Därefter rensas lysater genom låghastighetscentrifugering, och 15% av supernatanten används för total RNA (TOTAL) isolering. Det återstående rensade lysatet används för att isolera polysomer genom ultracentrifugering genom en tvåskikts sackaroskudde (12% och 33,5%). Polysomassocierat mRNA (PAR) renas från polysomala pellets efter resuspension. Både TOTAL och PAR utvärderas av mycket känslig kapillärelektrofores för att uppfylla kvalitetsstandarderna för sekvenseringsbibliotek för RNA-seq. Som ett exempel på en nedströms applikation, efter sekvensering, kan standardrörledningar för genuttrycksanalys användas för att erhålla differentiellt uttryckta gener på transkriptom- och translatomnivåerna. Sammanfattningsvis möjliggör denna metod, i kombination med RNA-seq, studier av translationell reglering av eukaryota mRNA i en komplex vävnad såsom den symbiotiska knölen.
Introduction
Baljväxter, såsom sojabönor (Glycine max), kan etablera symbios med specifika jordbakterier som kallas rhizobia. Detta mutualistiska förhållande framkallar bildandet av nya organ, de symbiotiska knölarna, på växtrötterna. Knölarna är växtorganen som är värd för bakterierna och består av värdceller vars cytoplasma koloniseras med en specialiserad form av rhizobia som kallas bakterioider. Dessa bakterioider katalyserar reduktionen av atmosfäriskt kväve (N2) till ammoniak, som överförs till växten i utbyte mot kolhydrater 1,2.
Även om denna kvävefixerande symbios är en av de mest välstuderade växt-mikrobsymbioserna, återstår många aspekter att förstå bättre, till exempel hur växter som utsätts för olika abiotiska stressförhållanden modulerar deras interaktion med sin symbiotiska partner och hur detta påverkar knölmetabolismen. Dessa processer kan förstås bättre genom att analysera knöltranslatomet (dvs. delmängden av budbärar-RNA [mRNA] som aktivt översätts). Polyribosomer eller polysomer är komplex av flera ribosomer associerade med mRNA, som vanligtvis används för att studera översättning3. Polysomprofileringsmetoden består av analys av mRNA associerade med polysomer och har framgångsrikt använts för att studera de posttranskriptionella mekanismerna som styr genuttryck som förekommer i olika biologiska processer 4,5.
Historiskt sett har genomuttrycksanalys främst fokuserat på att bestämma mRNA-överflöd 6,7,8,9. Det finns emellertid en brist på korrelation mellan transkript- och proteinnivåer på grund av de olika stadierna av posttranskriptionell reglering av genuttryck, särskilt översättning10,11,12. Dessutom har inget beroende observerats mellan förändringarna på transkriptomets nivå och de som uppträder vid translatomets nivå13. Den direkta analysen av den uppsättning mRNA som översätts möjliggör en mer exakt och fullständig mätning av cellgenuttrycket (vars slutpunkt är proteinmängd) än den som erhålls när endast mRNA-nivåer analyseras14,15,16.
Detta protokoll beskriver hur växtbaserade polysomer renas från intakta sojabönknutor genom differentiell centrifugering genom en tvåskikts sackaroskudde (figur 1). Men eftersom bakterioid-härledda ribosomer också finns i knölarna, renas en blandning av ribosomer och RNA-arter, även om de eukaryota representerar huvudfraktionen (90% -95%). Den efterföljande RNA-isoleringen, kvantifieringen och kvalitetskontrollen beskrivs också (figur 1). Detta protokoll, i kombination med RNA-seq, bör ge experimentella resultat om translationell reglering av eukaryota mRNA i en komplex vävnad såsom den symbiotiska knölen.
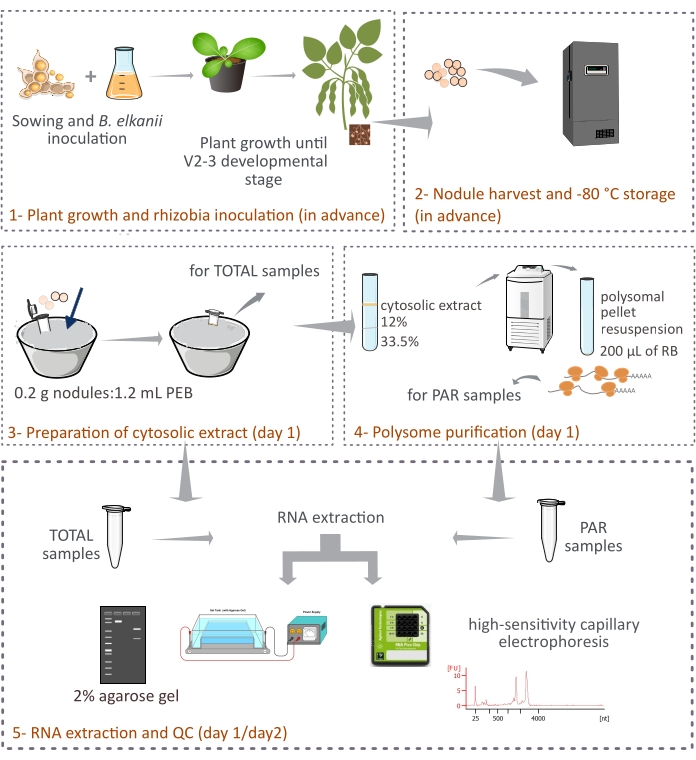
Figur 1: Schematisk översikt över den föreslagna metoden för eukaryot polysomrening från symbiotiska knölar. Schemat ger en översikt över de steg som följs i protokollet från (1) växttillväxt och (2) knölskörd till (3) beredning av cytosolextrakten, (3) erhållande av TOTAL-prover och (4) PAR-prover och (5) RNA-extraktion och kvalitetskontroll. Förkortningar: PEB = polysomextraktionsbuffert; RB = resuspensionsbuffert; TOTALT = totalt RNA; PAR = polysomassocierat mRNA. Klicka här för att se en större version av denna siffra.
Protocol
Representative Results
Discussion
Att studera genuttrycksreglering på translationell nivå är avgörande för att bättre förstå olika biologiska processer eftersom slutpunkten för cellgenuttryck är proteinförekomst13,14. Detta kan bedömas genom att analysera translatomet hos vävnaden eller organismen av intresse för vilken den polysomala fraktionen ska renas och dess associerade mRNA analyseras 3,4,34,35,36.<sup clas…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Denna forskning finansierades av CSIC I + D 2020 bidrag nr 282, FVF 2017 bidrag nr 210 och PEDECIBA (María Martha Sainz).
Materials
| Plant growth and rhizobia inoculation | |||
| Orbital shaker | Daihan Scientific | Model SHO-1D | |
| YEM-medium | Amresco | J850 (yeast extract) 0122 (mannitol) | |
| Water deficit treatment | |||
| KNO3 | Merck | 221295 | |
| Porometer | Decagon Device | Model SC-1 | |
| Scalpel | |||
| Preparation of cytosolic extracts | |||
| Brij L23 | Sigma-Aldrich | P1254 | |
| Centrifuge | Sigma | Model 2K15 | |
| Chloranphenicol | Sigma-Aldrich | C0378 | |
| Cycloheximide | Sigma-Aldrich | C7698 | |
| DOC | Sigma-Aldrich | 30970 | |
| DTT | Sigma-Aldrich | D9779 | |
| EGTA | Sigma-Aldrich | E3889 | |
| Igepal CA 360 | Sigma-Aldrich | I8896 | |
| KCl | Merck | 1.04936 | |
| MgCl2 | Sigma-Aldrich | M8266 | |
| Plastic tissue grinder | Fisher Scientific | 12649595 | |
| PMSF | Sigma-Aldrich | P7626 | |
| PTE | Sigma-Aldrich | P2393 | |
| Tris | Invitrogen | 15504-020 | |
| Triton X-100 | Sigma-Aldrich | T8787 | |
| Tween 20 | Sigma-Aldrich | P1379 | |
| Weighing dish | Deltalab | 1911103 | |
| Preparation of sucrose cushions | |||
| Sucrose | Invitrogen | 15503022 | |
| SW 40 Ti rotor | Beckman-Coulter | ||
| Ultracentrifuge | Beckman-Coulter | Optima L-100K | |
| Ultracentrifuge tubes | Beckman-Coulter | 344059 | 13.2 mL tubes |
| RNA extraction and quality control | |||
| Agarose | Thermo scientific | R0492 | |
| Bioanalyzer | Agilent | Model 2100. Eukaryote total RNA nano assay | |
| Chloroform | DI | 41191 | |
| Ethanol | Dorwil | UN1170 | |
| Isopropanol | Mallinckrodt | 3032-06 | |
| Glycogen | Sigma | 10814-010 | |
| TRIzol LS | Ambion | 102960028 | |
| Miscellaneous | |||
| Falcon tubes 15 mL | Biologix | 10-0152 | |
| Filter tips 10 µL | BioPointe Scientific | 321-4050 | |
| Filter tips 1000 µL | BioPointe Scientific | 361-1050 | |
| Filter tips 20 µL | BioPointe Scientific | 341-4050 | |
| Filter tips 200 µL | Tarsons | 528104 | |
| Microcentrifuge tubes 1.5 mL | Tarsons | 500010-N | |
| Microcentrifuge tubes 2.0 mL | Tarsons | 500020-N | |
| Sequencing company | Macrogen | ||
| Sterile 250 mL flask | Marienfeld | 4110207 |
References
- Limpens, E., et al. Cell- and tissue-specific transcriptome analyses of Medicago truncatula root nodules. PLoS ONE. 8 (5), 64377 (2013).
- Masson-Boivin, C., Giraud, E., Perret, X., Batut, J. Establishing nitrogen-fixing symbiosis with legumes: How many rhizobium recipes. Trends in Microbiology. 17 (10), 458-466 (2009).
- King, H. A., Gerber, A. P. Translatome profiling: Methods for genome-scale analysis of mRNA translation. Briefings in Functional Genomics. 15 (1), 22-31 (2014).
- Chassé, H., Boulben, S., Costache, V., Cormier, P., Morales, J. Analysis of translation using polysome profiling. Nucleic Acids Research. 45 (3), 15 (2017).
- Yángüez, E., Castro-Sanz, A. B., Fernández-Bautista, N., Oliveros, J. C., Castellano, M. M. Analysis of genome-wide changes in the translatome of Arabidopsis seedlings subjected to heat stress. PloS ONE. 8 (8), 71425 (2013).
- Brown, P. O., Botstein, D. Exploring the new world of the genome with DNA microarrays. Nature Genetics. 21, 33-37 (1999).
- Krishnamurthy, A., Ferl, R. J., Paul, A. -. L. Comparing RNA-Seq and microarray gene expression data in two zones of the Arabidopsis root apex relevant to spaceflight. Applications in Plant Sciences. 6 (11), 1197 (2018).
- Shulse, C. N., et al. High-throughput single-cell transcriptome profiling of plant cell types. Cell Reports. 27 (7), 2241-2247 (2019).
- Wang, Z., Gerstein, M., Snyder, M. RNA-Seq: A revolutionary tool for transcriptomics. Nature Reviews Genetics. 10 (1), 57-63 (2009).
- Kawaguchi, R., Girke, T., Bray, E. A., Bailey-Serres, J. Differential mRNA translation contributes to gene regulation under non-stress and dehydration stress conditions in Arabidopsis thaliana. The Plant Journal. 38 (5), 823-839 (2004).
- Larsson, O., Tian, B., Sonenberg, N. Toward a genome-wide landscape of translational control. Cold Spring Harbor Perspectives in Biology. 5 (1), 012302 (2013).
- Vogel, C., Marcotte, E. M. Insights into regulation of protein abundance from proteomics and transcriptomis analyses. Nature Reviews Genetics. 13 (4), 227-232 (2013).
- Tebaldi, T., et al. Widespread uncoupling between transcriptome and translatome variations after a stimulus in mammalian cells. BMC Genomics. 13 (220), 1-15 (2012).
- Wang, T., et al. Translating mRNAs strongly correlate to proteins in a multivariate manner and their translation ratios are phenotype specific. Nucleic Acids Research. 41 (9), 4743-4754 (2013).
- Ingolia, N. T. Ribosome profiling: New views of translation, from single codons to genome scale. Nature Reviews Genetics. 15 (3), 205-213 (2014).
- Lukoszek, R., Feist, P., Ignatova, Z. Insights into the adaptive response of Arabidopsis thaliana to prolonged thermal stress by ribosomal profiling and RNA-Seq. BMC Plant Biology. 16 (1), 1-13 (2016).
- Broughton, W. J., Dilworth, M. J. Control of leghaemoglobin synthesis in snake beans. Biochemical Journal. 125 (4), 1075-1080 (1971).
- Schroeder, A., et al. The RIN: An RNA integrity number for assigning integrity values to RNA measurements. BMC Molecular Biology. 7, 1-14 (2006).
- Rio, D. C., Ares, M., Hannon, G. J., Nilsen, T. W. Nondenaturing agarose gel electrophoresis of RNA nondenaturing agarose gel electrophoresis of RNA. Cold Spring Harbor Protocols. 2010 (6), 1-3 (2012).
- Ewels, P., Magnusson, M., Lundin, S., Käller, M. MultiQC: Summarize analysis results for multiple tools and samples in a single report. Bioinformatics. 32 (19), 3047-3048 (2016).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: A flexible trimmer for Illumina sequence data. Bioinformatics. 30 (15), 2114-2120 (2014).
- Marcel, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet.journal. 17 (1), 10-12 (2011).
- . Sickle: A sliding-window, adaptive, quality-based trimming tool for FastQ files (Version 1.33) Available from: https://github.com/najoshi/sickle (2011)
- Langmead, B., Salzberg, S. L. Fast gapped-read alignment with Bowtie 2. Nature Methods. 9 (4), 357-359 (2012).
- Kim, D., et al. TopHat2: Accurate alignment of transcriptomes in the presence of insertions, deletions and gene fusions. Genome Biology. 14 (4), 1-13 (2013).
- Patro, R., Duggal, G., Love, M. I., Irizarry, R. A., Kingsford, C. Salmon provides fast and bias-aware quantification of transcript expression. Nature Methods. 14 (4), 417-419 (2017).
- Liao, Y., Smyth, G. K., Shi, W. featureCounts: An efficient general purpose program for assigning sequence reads to genomic features. Bioinformatics. 30 (7), 923-930 (2013).
- Robinson, M. D., McCarthy, D. J., Smyth, G. K. edgeR: A Bioconductor package for differential expression analysis of digital gene expression data. Bioinformatics. 26 (1), 139-140 (2010).
- Love, M. I., Huber, W., Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biology. 15 (550), 1-21 (2014).
- Ritchie, M. E., et al. limma powers differential expression analyses for RNA-sequencing and microarray studies. Nucleic Acids Research. 43 (7), 47 (2015).
- Afgan, E., et al. The Galaxy platform for accessible, reproducible and collaborative biomedical analyses: 2018 update. Nucleic Acids Research. 46, 537-544 (2018).
- Hunt, G. P., et al. GEOexplorer: A webserver for gene expression analysis and visualisation. Nucleic Acids Research. , (2022).
- Imbeaud, S., et al. Towards standardization of RNA quality assessment using user-independent classifiers of microcapillary electrophoresis traces. Nucleic Acids Research. 33 (6), 1-12 (2005).
- Di Paolo, A., et al. PDCD4 regulates axonal growth by translational repression of neurite growth-related genes and is modulated during nerve injury responses. RNA. 26 (11), 1637-1653 (2020).
- Smircich, P., et al. Ribosome profiling reveals translation control as a key mechanism generating differential gene expression in Trypanosoma cruzi. BMC Genomics. 16 (1), 443 (2015).
- Eastman, G., Sharlow, E. R., Lazo, J. S., Bloom, G. S., Sotelo-Silveira, J. R. Transcriptome and translatome regulation of pathogenesis in Alzheimer’s disease model mice. Journal of Alzheimer’s Disease. 86 (1), 365-386 (2022).
- Zanetti, M. E., Chang, I. -. F., Gong, F., Galbraith, D. W., Bailey-Serres, J. Immunopurification of polyribosomal complexes of Arabidopsis for global analysis of gene expression. Plant physiology. 138 (2), 624-635 (2005).
- Mustroph, A., et al. Profiling translatomes of discrete cell populations resolves altered cellular priorities during hypoxia in Arabidopsis. Proceedings of the National Academy of Sciences of the United States of America. 106 (44), 18843-11848 (2009).
- Ingolia, N. T., Ghaemmaghami, S., Newman, J. R. S., Weissman, J. S. Genome-wide analysis in vivo of translation with nucleotide resolution using ribosome profiling. Science. 324 (5924), 218-223 (2009).
- Brar, G. A., Weissman, J. S. Ribosome profiling reveals the what, when, where and how of protein synthesis. Nature Reviews. Molecular Cell Biology. 16 (11), 651-664 (2015).
- Eastman, G., Smircich, P., Sotelo-Silveira, J. R. Following ribosome footprints to understand translation at a genome wide level. Computational and Structural Biotechnology Journal. 16, 167-176 (2018).
- Westermann, A. J., Gorski, S. A., Vogel, J. Dual RNA-seq of pathogen and host. Nature Reviews Microbiology. 10 (9), 618-630 (2012).

