Constructie van metabolische netwerken uit evenwicht in blaasjes ter grootte van nano- en micrometer
Summary
We presenteren een protocol voor het reconstitueren van membraaneiwitten en het inkapselen van enzymen en andere in water oplosbare componenten in lipideblaasjes van submicrometer en micrometer grootte.
Abstract
We presenteren een methode om complexe eiwitnetwerken in blaasjes op te nemen, met integrale membraaneiwitten, enzymen en op fluorescentie gebaseerde sensoren, met behulp van gezuiverde componenten. Deze methode is relevant voor het ontwerp en de bouw van bioreactoren en de studie van complexe metabole reactienetwerken die uit evenwicht zijn. We beginnen met het reconstrueren van (meerdere) membraaneiwitten tot grote unilamellaire blaasjes (LUV’s) volgens een eerder ontwikkeld protocol. Vervolgens kapselen we een mengsel van gezuiverde enzymen, metabolieten en op fluorescentie gebaseerde sensoren (fluorescerende eiwitten of kleurstoffen) in via vries-dooi-extrusie en verwijderen we niet-opgenomen componenten door centrifugatie en/of grootte-uitsluitingschromatografie. De prestaties van de metabole netwerken worden in realtime gemeten door de ATP/ADP-verhouding, metabolietconcentratie, interne pH of andere parameters te bewaken door middel van fluorescentie-uitlezing. Onze membraaneiwitbevattende blaasjes met een diameter van 100-400 nm kunnen worden omgezet in reuzen-unilamellaire blaasjes (GUV’s), met behulp van bestaande maar geoptimaliseerde procedures. De aanpak maakt het mogelijk om oplosbare componenten (enzymen, metabolieten, sensoren) op te nemen in blaasjes ter grootte van een micrometer, waardoor het volume van de bioreactoren met ordes van grootte wordt opgeschaald. Het metabolische netwerk dat GUV’s bevat, zit gevangen in microfluïdische apparaten voor analyse door optische microscopie.
Introduction
Het vakgebied van bottom-up synthetische biologie richt zich op het bouwen van (minimale) cellen 1,2 en metabole bioreactoren voor biotechnologische 3,4 of biomedische doeleinden 5,6,7,8. De constructie van synthetische cellen biedt een uniek platform dat onderzoekers in staat stelt om (membraan)eiwitten te bestuderen in goed gedefinieerde omstandigheden die die van inheemse omgevingen nabootsen, waardoor opkomende eigenschappen en verborgen biochemische functies van eiwitten en reactienetwerken kunnen worden ontdekt9. Als tussenstap naar een autonoom functionerende synthetische cel worden modules ontwikkeld die essentiële kenmerken van levende cellen vastleggen, zoals metabolisch energiebehoud, eiwit- en lipidensynthese en homeostase. Dergelijke modules vergroten niet alleen ons begrip van het leven, maar hebben ook potentiële toepassingen op het gebied van geneeskunde8 en biotechnologie10.
Transmembraaneiwitten vormen de kern van vrijwel elk metabolisch netwerk, omdat ze moleculen in of uit de cel transporteren, signalen geven en reageren op de kwaliteit van de omgeving, en tal van biosynthetische rollen spelen. De engineering van metabole modules in synthetische cellen vereist dus in de meeste gevallen de reconstitutie van integrale en/of perifere membraaneiwitten tot een membraandubbellaag die is samengesteld uit specifieke lipiden en een hoge integriteit (lage permeabiliteit). De omgang met deze membraaneiwitten is uitdagend en vereist specifieke kennis en experimentele vaardigheden.
Er zijn verschillende methoden ontwikkeld om membraaneiwitten in fosfolipideblaasjes te reconstrueren, meestal met als doel de functie11,12, regulatie13, kinetische eigenschappen14,15, lipideafhankelijkheid15,16 en/of stabiliteit17 van een specifiek eiwit te bestuderen. Deze methoden omvatten de snelle verdunning van detergent-oplosbaar eiwit in waterige media in aanwezigheid van lipiden18, de verwijdering van detergenten door het incuberen van detergent-solubel eiwit met detergent-gedestabiliseerde lipideblaasjes en absorptie van het detergent(en) op polystyreenkorrels19, of het verwijderen van detergenten door dialyse of grootte-uitsluitingschromatografie20. Organische oplosmiddelen zijn gebruikt om lipideblaasjes te vormen, bijvoorbeeld via de vorming van olie-water-interfasen21, maar de meeste integrale membraaneiwitten worden geïnactiveerd wanneer ze worden blootgesteld aan dergelijke oplosmiddelen.
In ons laboratorium reconstrueren we meestal membraaneiwitten door middel van de detergent-absorptiemethode om grote unilamellaire blaasjes (LUV’s) te vormen19. Deze methode maakt de co-reconstitutie van meerdere membraaneiwitten en de inkapseling in het blaasjeslumen van enzymen, metabolieten en sondes mogelijk22,23. De membraaneiwitbevattende LUV’s kunnen worden omgezet in reusachtige-unilamellaire blaasjes (GUV’s) met/zonder inkapseling van in water oplosbare componenten, met behulp van elektroformatie24 of gelondersteunde zwelling25 en specifieke omstandigheden om de integriteit van de membraaneiwitten te behouden26.
Dit artikel presenteert een protocol voor de reconstitutie in LUV’s van een uit evenwicht zijnd metabolisch netwerk dat ATP regenereert door de afbraak van L-arginine in L-ornithine27. De vorming van ATP gaat gepaard met de productie van glycerol-3-fosfaat (G3P), een belangrijke bouwsteen voor de fosfolipidensynthese22,28. De metabole route bestaat uit twee integrale membraaneiwitten, een arginine/ornithine (ArcD) en een G3P/Pi-antiporter (GlpT). Daarnaast zijn drie oplosbare enzymen (ArcA, ArcB, ArcC) nodig voor de recycling van ATP, en GlpK wordt gebruikt om glycerol om te zetten in glycerol 3-fosfaat, waarbij gebruik wordt gemaakt van het ATP uit de afbraak van L-arginine, zie figuur 1 voor een schematisch overzicht van de route. Dit protocol vormt een goed uitgangspunt voor de toekomstige constructie van nog complexere reactienetwerken – voor de synthese van lipiden of eiwitten of de deling van cellen. De lipidensamenstelling van de blaasjes ondersteunt de activiteit van een breed scala aan integrale membraaneiwitten en is geoptimaliseerd voor het transport van diverse moleculen in of uit de blaasjes 27,29,30.
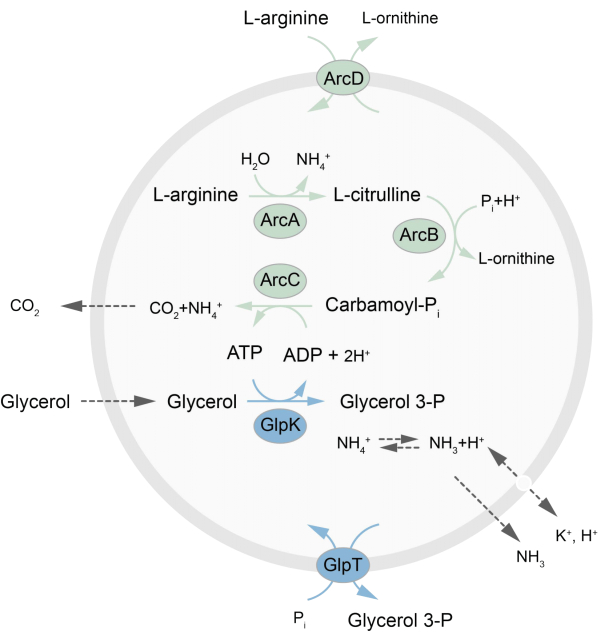
Figuur 1: Overzicht van de route voor ATP-productie en glycerol-3-fosfaatsynthese en -uitscheiding. Klik hier om een grotere versie van deze figuur te bekijken.
Kortom, gezuiverde membraaneiwitten (opgelost in dodecyl-β-D-maltoside, DDM) worden toegevoegd aan voorgevormde lipideblaasjes die zijn gedestabiliseerd met Triton X-100, waardoor de eiwitten in het membraan kunnen worden ingebracht. De wasmiddelmoleculen worden vervolgens (langzaam) verwijderd door de toevoeging van geactiveerde polystyreenkorrels, wat resulteert in de vorming van goed afgesloten proteoliposomen. Oplosbare componenten kunnen vervolgens aan de blaasjes worden toegevoegd en worden ingekapseld via vries-dooicycli, waardoor de moleculen worden opgesloten in het proces van membraanfusie. De verkregen blaasjes zijn zeer heterogeen en veel zijn multilamellair. Ze worden vervolgens geëxtrudeerd door een polycarbonaatfilter met een poriegrootte van 400, 200 of 100 nm, wat blaasjes van meer uniforme grootte oplevert; Hoe kleiner de poriegrootte, hoe homogener en unilamellair de blaasjes, maar tegen de prijs van een kleiner inwendig volume. Niet-opgenomen eiwitten en kleine moleculen worden uit de externe oplossing verwijderd door middel van grootte-uitsluitingschromatografie. De proteoLUV’s kunnen worden omgezet in blaasjes ter grootte van een micrometer door middel van gel-geassisteerde zwelling, en deze proteoGUV’s worden vervolgens verzameld en gevangen in een microfluïdische chip voor microscopische karakterisering en manipulatie. Figuur 2 geeft een schematisch overzicht van het volledige protocol.
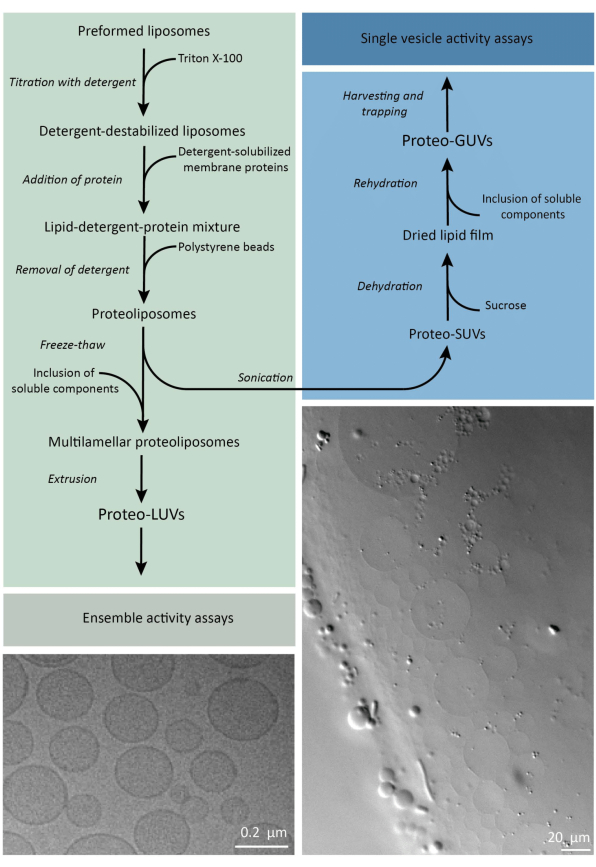
Figuur 2: Overzicht van het protocol voor het reconstitueren van membraaneiwitten en het inkapselen van enzymen en wateroplosbare componenten in lipideblaasjes met een submicrometer (LUV’s) en micrometergrootte (GUV’s). Klik hier om een grotere versie van deze figuur te bekijken.
De reconstitutie- en inkapselingsprotocollen werken goed en de functionaliteit van de eiwitten blijft behouden, maar de proteoLUV’s en proteoGUV’s zijn heterogeen van grootte. Microfluïdische benaderingen31,32 maken de vorming van blaasjes ter grootte van een micrometer mogelijk die homogener van grootte zijn, maar functionele reconstitutie van membraaneiwitten is over het algemeen niet mogelijk omdat resterend oplosmiddel in de dubbellaag de eiwitten inactiveert. De proteoLUV’s variëren in grootte van 100 tot 400 nm, en bij lage concentraties enzymen kan de inkapseling leiden tot blaasjes met onvolledige metabole routes (stochastische effecten; zie figuur 3). LUV’s zijn ideaal voor het bouwen van specifieke metabolische modules, zoals hier te zien is voor de productie van ATP en bouwstenen zoals G3P. Dergelijke proteoLUV’s kunnen mogelijk worden ingekapseld in GUV’s en dienen als organelachtige compartimenten voor de gastheerblaasjes.
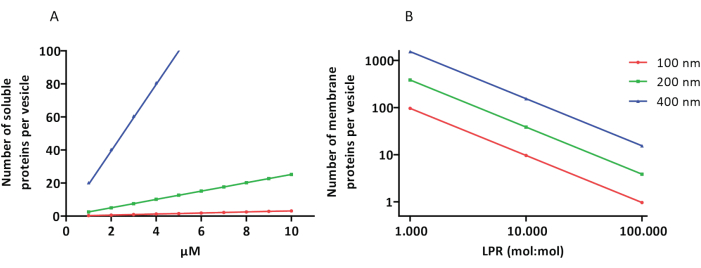
Figuur 3: Aantal moleculen per blaasje met een diameter van 100, 200 of 400 nm. (A) Wanneer de ingekapselde eiwitten (enzymen, sondes) zich in het bereik van 1-10 μM bevinden. (B) De reconstitutie wordt gedaan bij 1 tot 1.000, 1 tot 10.000 en 1 tot 100.000 membraaneiwitten per lipide (mol/mol). We gaan ervan uit dat moleculen in de aangegeven concentraties worden ingekapseld en in het membraan worden opgenomen in deze eiwit-lipideverhoudingen. Voor sommige enzymen hebben we gezien dat ze zich binden aan membranen, waardoor hun schijnbare concentratie in de blaasjes kan toenemen. Afkorting: LPR = Lipid-Protein-Ratio Klik hier om een grotere versie van deze figuur te bekijken.
Protocol
Representative Results
Discussion
We presenteren een protocol voor de synthese van (membraan)eiwit met lipideblaasjes van minder dan een micrometer grootte (proteoLUV’s), en de omzetting van proteoLUV’s in reus-unilamellaire blaasjes (proteoGUV’s). Het protocol moet van toepassing zijn op de reconstitutie van andere membraaneiwitten 13,19,30,40 en de inkapseling van andere metabole netwerken dan de hier gepresenteerde L-arginineafbraak- en glycerol-3-fosfaatsyntheseroutes.<sup class="x…
Disclosures
The authors have nothing to disclose.
Acknowledgements
De auteurs bedanken Aditya Iyer voor het klonen van het pBAD-PercevalHR-gen en Gea Schuurman-Wolters voor haar hulp bij de productie en zuivering van eiwitten. Het onderzoek werd gefinancierd door het NWO Zwaartekracht programma “Building a Synthetic Cell” (BaSyC).
Materials
| Agarose | Sigma Aldrich | A9414-25g | |
| Amicon cut-off filter | Sigma Aldrich | Milipore centrifugal filter units Amicon Ultra | |
| BioBeads | BioRad | 152-3920 | |
| CHCl3 | Macron Fine Chemicals | MFCD00000826 | |
| D(+)-Glucose | Formedium | – | |
| D(+)-Sucrose | Formedium | – | |
| DDM | Glycon | D97002 -C | |
| Diethyl Ether | Biosolve | 52805 | |
| DMSO | Sigma-Aldrich | 276855-100ml | |
| DOPC | Avanti | 850375P-1g | |
| DOPE | Avanti | 850725P-1g | |
| DOPG | Avanti | 840475P-1g | |
| DTT | Formedium | DTT005 | |
| EtOH | J.T.Baker Avantor | MFCD00003568 | |
| Extruder | Avestin Inc | LF-1 | |
| Fluorimeter | Jasco | Spectrofluorometer FP-8300 | |
| Glycerol | BOOM | 51171608 | |
| Gravity flow column | Bio-Rad | 732-1010 | |
| Hamilton syringe 100 µL | Hamilton | 7656-01 | |
| Hamilton syringe 1000 µL | Hamilton | 81320 | |
| Handheld LCP dispenser | Art Robbins Instruments | 620-411-00 | |
| Handheld Sonicator | Hielscher Ultrasound Technology | UP50H | |
| HCl | BOOM | x76021889.1000 | |
| Imidazole | Roth | X998.4-250g | |
| K2HPO4 | Supelco | 1.05099.1000 | |
| KCl | BOOM | 76028270.1 | |
| KH2PO4 | Supelco | 1.04873.1000 | |
| Kimwipe | Kimtech Science | 7552 | |
| Large Falcon tube centrifuge | Eppendorf | Centrifuge 5810 R | |
| L-Arginine | Sigma-Aldrich | A5006-100G | |
| Light microscope | Leica | DM LS2 | |
| L-Ornithine | Roth | T204.1 | |
| LSM Laser Scanning Confocal Microscope | Zeiss | LSM 710 ConfoCor 3 | |
| MgCl2 | Sigma-Aldrich | M2670-1KG | |
| Microfluidic chip | Homemade | PDMS based | DOI: https://doi.org/10.1039/C8LC01275J |
| Na-ADP | Sigma-Aldrich | A2754-1G | |
| NaCl | Supelco | 1.06404.1000 | |
| Nanodrop Spectrometer | Isogen Life Science | ND-1000 spectrophotometer NanoDrop | |
| NaOH | Supelco | 1.06498.1000 | |
| Needles for GUVs | Henke-Ject | 14-14575 | 27 G x 3/4'' 0.4 x 20 mm |
| Needles for microfluidics | Henke-Ject | 14-15538 | 18 G x 1 1/2'' 1.2 x 40 mm |
| Ni2+ Sepharose | Cytiva | 17526802 | |
| Nigericin | Sigma-Aldrich | N7143-5MG | |
| Nutator | VWR | 83007-210 | |
| Osmolality meter | Gonotec Salmenkipp | Osmomat 3000 basic freezing point osmometer | |
| Plasmacleaner | Plasma Etch | PE-Avenger | |
| Polycarbonate filter | Cytiva Whatman | Nuclepor Track-Etch Membrane Product: 10417104 | 0.4 µm |
| Polycarbonate ultracentrifuge tube | Beckman Coulter | 355647 | |
| Pyranine | Acros Organics | H1529-1G | |
| Quartz cuvette (black) | Hellma Analytics | 108B-10-40 | |
| Sephadex G-75 resin | GE Healthcare | 17-0050-01 | |
| Sonicator | Sonics Sonics & Materials INC | Sonics vibra cell | |
| Syringe filter | Sarstedt | Filtropur S plus 0.2 | 0.2 µm |
| Syringe pump | Harvard Apparatus | A-42467 | |
| Tabletop centrifuge | Eppendorf | centrifuge 5418 | |
| Teflon spacer | Homemade | Teflon based | 45 x 26 x 1.5 or 45 x 26 x 3 or 20 x 20 x 3 mm |
| Tris | PanReac AppliChem | A1086.1000 | |
| Triton X-100 | Sigma Aldrich | T8787-100 ml | |
| Ultracentrifuge | Beckman Coulter | Optima Max-E | |
| UV lamp | Spectroline | ENB-280C/FE | |
| UV/VIS Spectrometer | Jasco | V730 spectrophotometer | |
| Valinomycin | Sigma-Aldrich | V0627-10MG | |
| Widefield fluorescence microscope | Zeiss | AxioObserver | |
| β-Casein | Sigma Aldrich | C5890-500g |
References
- Hirschi, S., Ward, T. R., Meier, W. P., Müller, D. J., Fotiadis, D. Synthetic biology: bottom-up assembly of molecular systems. Chem Rev. 122 (21), 16294-16328 (2022).
- Ivanov, I., et al. Bottom-up synthesis of artificial cells: recent highlights and future challenges. Annu Rev Chem Biomol. Eng. 12 (1), 287-308 (2021).
- Clomburg, J. M., Crumbley, A. M., Gonzalez, R. Industrial biomanufacturing: The future of chemical production. Science. 355 (6320), (2017).
- Shi, T., Han, P., You, C., Zhang, Y. -. H. P. J. An in vitro synthetic biology platform for emerging industrial biomanufacturing: Bottom-up pathway design. Synth Syst Biotechnol. 3 (3), 186-195 (2018).
- Wang, A., et al. Liver-target and glucose-responsive polymersomes toward mimicking endogenous insulin secretion with improved hepatic glucose utilization. Adv Funct Mater. 30 (13), 1910168 (2020).
- Kanter, G., et al. Cell-free production of scFv fusion proteins: an efficient approach for personalized lymphoma vaccines. Blood. 109 (8), 3393-3399 (2007).
- Zeltins, A. Construction and characterization of virus-like particles: a review. Mol Biotechnol. 53 (1), 92-107 (2013).
- Jain, K. K. Synthetic biology and personalized medicine. Med Princ Pract. 22 (3), 209-219 (2013).
- Schwille, P., Frohn, B. P. Hidden protein functions and what they may teach us. Trends Cell Biol. 32 (2), 102-109 (2022).
- Sachsenmeier, P. Industry 5.0-The relevance and implications of bionics and synthetic biology. Engineering. 2 (2), 225-229 (2016).
- Schmidt, D., Jiang, Q. -. X., MacKinnon, R. Phospholipids and the origin of cationic gating charges in voltage sensors. Nature. 444 (7120), 775-779 (2006).
- Godoy-Hernandez, A., et al. Rapid and highly stable membrane reconstitution by LAiR enables the study of physiological integral membrane protein functions. ACS Cent Sci. 9 (3), 494-507 (2023).
- Sikkema, H. R., et al. Gating by ionic strength and safety check by cyclic-di-AMP in the ABC transporter OpuA. Sci Adv. 6 (47), 7697 (2020).
- Foucaud, C., Poolman, B. Lactose transport system of Streptococcus thermophilus. Functional reconstitution of the protein and characterization of the kinetic mechanism of transport. J Biol Chem. 267 (31), 22087-22094 (1992).
- Yoneda, J. S., Sebinelli, H. G., Itri, R., Ciancaglini, P. Overview on solubilization and lipid reconstitution of Na,K-ATPase: enzyme kinetic and biophysical characterization. Biophys Rev. 12 (1), 49-64 (2020).
- Simidjiev, I., et al. Self-assembly of large, ordered lamellae from non-bilayer lipids and integral membrane proteins in vitro. Proc Natl Acad Sci. 97 (4), 1473-1476 (2000).
- Harris, N. J., Booth, P. J. Folding and stability of membrane transport proteins in vitro. Biochim Biophys Acta BBA – Biomembr. 1818 (4), 1055-1066 (2012).
- Jackson, M. L., Litman, B. J. Rhodopsin-egg phosphatidylcholine reconstitution by an octyl glucoside dilution procedure. Biochim Biophys Acta BBA – Biomembr. 812 (2), 369-376 (1985).
- Geertsma, E. R., Nik Mahmood, N. A. B., Schuurman-Wolters, G. K., Poolman, B. Membrane reconstitution of ABC transporters and assays of translocator function. Nat Protoc. 3 (2), 256-266 (2008).
- Rigaud, J. -. L., Pitard, B., Levy, D. Reconstitution of membrane proteins into liposomes: application to energy-transducing membrane proteins. Biochim Biophys Acta BBA – Bioenerg. 1231 (3), 223-246 (1995).
- Szoka, F., Papahadjopoulos, D. Procedure for preparation of liposomes with large internal aqueous space and high capture by reverse-phase evaporation. Proc Natl Acad Sci. 75 (9), 4194-4198 (1978).
- . Synthetic Organelles for Energy Conservation and Delivery of Building Blocks for Lipid Biosynthesis Available from: https://www.researchsquare.com/article/rs-3385355/v1 (2023)
- Lee, K. Y., et al. Photosynthetic artificial organelles sustain and control ATP-dependent reactions in a protocellular system. Nat Biotechnol. 36 (6), 530-535 (2018).
- Méléard, P., Bagatolli, L. A., Pott, T. Giant unilamellar vesicle electroformation. Methods in Enzymology. , 161-176 (2009).
- Garten, M., Aimon, S., Bassereau, P., Toombes, G. E. S. Reconstitution of a transmembrane protein, the voltage-gated ion channel, KvAP, into giant unilamellar vesicles for microscopy and patch clamp studies. J. Vis. Exp. (95), e52281 (2015).
- Doeven, M. K., et al. lateral mobility and function of membrane proteins incorporated into giant unilamellar vesicles. Biophys J. 88 (2), 1134-1142 (2005).
- Pols, T., et al. A synthetic metabolic network for physicochemical homeostasis. Nat Commun. 10 (1), 4239 (2019).
- Bailoni, E., Poolman, B. ATP recycling fuels sustainable glycerol 3-phosphate formation in synthetic cells fed by dynamic dialysis. ACS Synth Biol. 11 (7), 2348-2360 (2022).
- Van Der Heide, T. On the osmotic signal and osmosensing mechanism of an ABC transport system for glycine betaine. EMBO J. 20 (24), 7022-7032 (2001).
- Van’T Klooster, J. S., et al. Membrane lipid requirements of the lysine transporter Lyp1 from Saccharomyces cerevisiae. J Mol Biol. 432 (14), 4023-4031 (2020).
- Lou, G., Anderluzzi, G., Woods, S., Roberts, C. W., Perrie, Y. A novel microfluidic-based approach to formulate size-tuneable large unilamellar cationic liposomes: Formulation, cellular uptake and biodistribution investigations. Eur J Pharm Biopharm. 143, 51-60 (2019).
- Weiss, M., et al. Sequential bottom-up assembly of mechanically stabilized synthetic cells by microfluidics. Nat Mater. 17 (1), 89-96 (2018).
- Pols, T., Singh, S., Deelman-Driessen, C., Gaastra, B. F., Poolman, B. Enzymology of the pathway for ATP production by arginine breakdown. FEBS J. 288 (1), 293-309 (2021).
- Yandrapalli, N., Robinson, T. Ultra-high capacity microfluidic trapping of giant vesicles for high-throughput membrane studies. Lab Chip. 19 (4), 626-633 (2019).
- Elias, M., et al. Microfluidic characterization of biomimetic membrane mechanics with an on-chip micropipette. Micro Nano Eng. 8, 100064 (2020).
- Robinson, T., Kuhn, P., Eyer, K., Dittrich, P. S. Microfluidic trapping of giant unilamellar vesicles to study transport through a membrane pore. Biomicrofluidics. 7 (4), 044105 (2013).
- Cooper, A., Girish, V., Subramaniam, A. B. Osmotic Pressure Enables High-Yield Assembly of Giant Vesicles in Solutions of Physiological Ionic Strengths. Langmuir. 39 (15), 5579-5590 (2023).
- Tantama, M., Martínez-François, J. R., Mongeon, R., Yellen, G. Imaging energy status in live cells with a fluorescent biosensor of the intracellular ATP-to-ADP ratio. Nat Commun. 4 (1), 2550 (2013).
- Setyawati, I., et al. In vitro reconstitution of dynamically interacting integral membrane subunits of energy-coupling factor transporters. eLife. 9, e64389 (2020).
- Oropeza-Guzman, E., Ríos-Ramírez, M., Ruiz-Suárez, J. C. Leveraging the coffee ring effect for a defect-free electroformation of giant unilamellar vesicles. Langmuir. 35 (50), 16528-16535 (2019).
- Estes, D. J., Mayer, M. Electroformation of giant liposomes from spin-coated films of lipids. Colloids Surf B Biointerfaces. 42 (2), 115-123 (2005).

.
