Построение неравновесных метаболических сетей в нано- и микрометровых везикулах
Summary
Представлен протокол восстановления мембранных белков и инкапсуляции ферментов и других водорастворимых компонентов в липидных везикулах субмикронного и микрометрового размера.
Abstract
Мы представляем метод встраивания в везикулы сложных белковых сетей, включающих интегральные мембранные белки, ферменты и флуоресцентные сенсоры, с использованием очищенных компонентов. Этот метод актуален для проектирования и строительства биореакторов и исследования сложных неравновесных сетей метаболических реакций. Мы начинаем с восстановления (множественных) мембранных белков в большие униламеллярные везикулы (LUV) в соответствии с ранее разработанным протоколом. Затем мы инкапсулируем смесь очищенных ферментов, метаболитов и флуоресцентных сенсоров (флуоресцентных белков или красителей) с помощью замораживания-размораживания-экструзии и удаляем неинкорпорированные компоненты с помощью центрифугирования и/или эксклюзионной хроматографии. Производительность метаболических сетей измеряется в режиме реального времени путем мониторинга соотношения АТФ/АДФ, концентрации метаболитов, внутреннего pH или других параметров с помощью флуоресцентного считывания. Наши мембранные белкисодержащие везикулы диаметром 100-400 нм могут быть преобразованы в гигантские униламеллярные везикулы (GUV) с использованием существующих, но оптимизированных процедур. Этот подход позволяет включать растворимые компоненты (ферменты, метаболиты, сенсоры) в везикулы микрометрового размера, тем самым увеличивая объем биореакторов на порядки. Метаболическая сеть, содержащая GUV, захватывается микрофлюидными устройствами для анализа с помощью оптической микроскопии.
Introduction
Область синтетической биологии снизу вверх фокусируется на конструировании (минимальных) клеток 1,2 и метаболических биореакторов для биотехнологических 3,4 или биомедицинских целей 5,6,7,8. Конструирование синтетических клеток обеспечивает уникальную платформу, которая позволяет исследователям изучать (мембранные) белки в четко определенных условиях, имитирующих условия нативной среды, что позволяет обнаружить эмерджентные свойства и скрытые биохимические функции белков и реакционных сетей9. В качестве промежуточного шага на пути к автономно функционирующей синтетической клетке разрабатываются модули, которые улавливают основные характеристики живых клеток, такие как сохранение метаболической энергии, синтез белков и липидов, а также гомеостаз. Такие модули не только улучшают наше понимание жизни,но и имеют потенциальное применение в области медицины и биотехнологии10.
Трансмембранные белки лежат в основе практически любой метаболической сети, поскольку они транспортируют молекулы внутрь или из клетки, сигнализируют и реагируют на качество окружающей среды, а также играют многочисленные биосинтетические роли. Таким образом, инженерия метаболических модулей в синтетических клетках в большинстве случаев требует восстановления интегральных и/или периферических мембранных белков в мембранный бислой, состоящий из специфических липидов и обладающих высокой целостностью (низкой проницаемостью). Работа с этими мембранными белками сложна и требует специальных знаний и экспериментальных навыков.
Было разработано несколько методов для восстановления мембранных белков в фосфолипидных везикулах, чаще всего с целью изучения функции11,12, регуляции13, кинетических свойств14,15, липидной зависимости 15,16 и/или стабильности17 конкретного белка. Эти методы включают быстрое разведение растворимого в детергентах белка в водную среду в присутствии липидов18, удаление детергентов путем инкубации растворимого в моющем средстве белка с дестабилизированными липидными везикулами и абсорбцию детергента(ов) на гранулах полистирола19 или удаление детергентов с помощью диализа или эксклюзионной хроматографии20. Органические растворители использовались для образования липидных везикул, например, путем образования интерфаз21 нефть-вода, но большинство интегральных мембранных белков инактивируются при воздействии таких растворителей.
В нашей лаборатории мы в основном восстанавливаем мембранные белки методом детергент-абсорбции с образованием крупных униламеллярных везикул (LUV)19. Этот метод позволяет осуществлять совместное воссоздание множественных мембранных белков и инкапсуляцию в просвет везикулы ферментов, метаболитов и зондов22,23. Мембранные белки, содержащие LUV, могут быть преобразованы в гигантские униламеллярные везикулы (GUV) с инкапсуляцией водорастворимых компонентов или без нее, используя либо электрообразование24, либо гелеобразное набухание25 и специальные условия для сохранения целостности мембранных белков26.
В данной статье представлен протокол восстановления в LUV неравновесной метаболической сети, которая регенерирует АТФ путем распада L-аргинина на L-орнитин27. Образование АТФ связано с образованием глицерол-3-фосфата (G3P), важного строительного блока для синтеза фосфолипидов22,28. Метаболический путь состоит из двух интегральных мембранных белков: аргинина/орнитина (ArcD) и антипорта G3P/Pi (GlpT). Кроме того, три растворимых фермента (ArcA, ArcB, ArcC) необходимы для рециркуляции АТФ, а GlpK используется для превращения глицерина в глицерин-3-фосфат с использованием АТФ из распада L-аргинина, см. Рисунок 1 для схематического обзора пути. Этот протокол представляет собой хорошую отправную точку для будущего построения еще более сложных реакционных сетей — для синтеза липидов или белков или для деления клеток. Липидный состав везикул поддерживает активность широкого спектра интегральных мембранных белков и был оптимизирован для транспортировки различных молекул внутрь или из везикул 27,29,30.
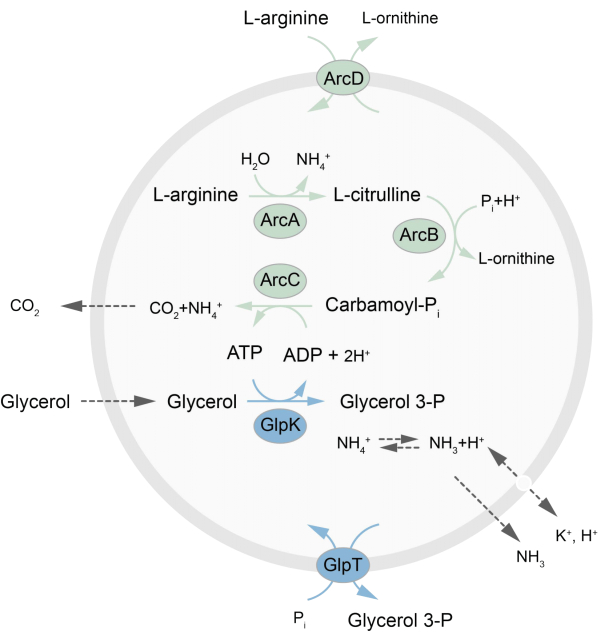
Рисунок 1: Обзор пути производства АТФ, синтеза и выведения глицерин-3-фосфата. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
Короче говоря, очищенные мембранные белки (солюбилизированные в додецил-β-D-мальтозиде, ДДМ) добавляются к предварительно сформированным липидным везикулам, которые были дестабилизированы с помощью Triton X-100, что позволяет встраивать белки в мембрану. Молекулы детергента впоследствии (медленно) удаляются путем добавления активированных гранул полистирола, что приводит к образованию хорошо запечатанных протеолипосом. Растворимые компоненты затем могут быть добавлены в везикулы и инкапсулированы с помощью циклов замораживания-оттаивания, что улавливает молекулы в процессе слияния мембран. Полученные везикулы очень неоднородны и многие из них являются многопластинчатыми. Затем они выдавливаются через поликарбонатный фильтр с размером пор 400, 200 или 100 нм, что дает пузырьки более равномерного размера; Чем меньше размер пор, тем более однородными и одноламеллярными являются везикулы, но ценой меньшего внутреннего объема. Неинкорпорированные белки и малые молекулы удаляются из наружного раствора с помощью эксклюзионной хроматографии. ПротеоЛУВ могут быть преобразованы в микрометровые везикулы путем гелеобразного набухания, а затем эти протеоГУВ собираются и удерживаются в микрофлюидном чипе для микроскопической характеристики и манипуляций. На рисунке 2 показан схематический обзор полного протокола.
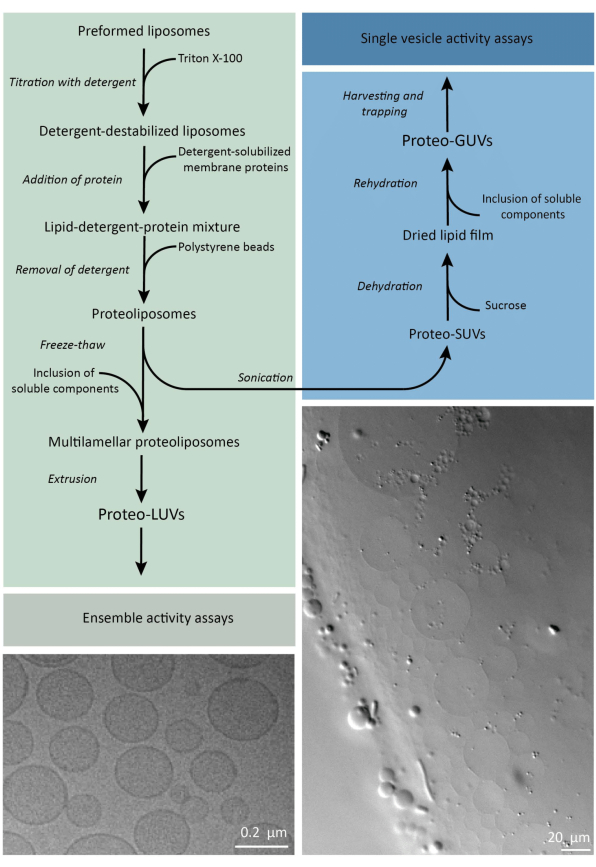
Рисунок 2: Обзор протокола восстановления мембранных белков и инкапсуляции ферментов и водорастворимых компонентов в липидных везикулах субмикронного (LUV) и микрометрового размера (GUVs). Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
Протоколы восстановления и инкапсуляции работают хорошо, и функциональность белков сохраняется, но proteoLUV и proteoGUVs неоднородны по размеру. Микрофлюидные подходы31,32 позволяют формировать везикулы микрометрового размера, которые являются более однородными по размеру, но функциональное восстановление мембранных белков, как правило, невозможно, поскольку остаточный растворитель в бислое инактивирует белки. Размер протеоЛУВ колеблется от 100 до 400 нм, и при низких концентрациях ферментов инкапсуляция может привести к образованию везикул с неполными метаболическими путями (стохастические эффекты; см. рис. 3). LUV идеально подходят для создания определенных метаболических модулей, как показано здесь для производства АТФ и строительных блоков, таких как G3P. Такие proteoLUV потенциально могут быть инкапсулированы в GUV и служить органеллоподобными компартментами для везикул хозяина.
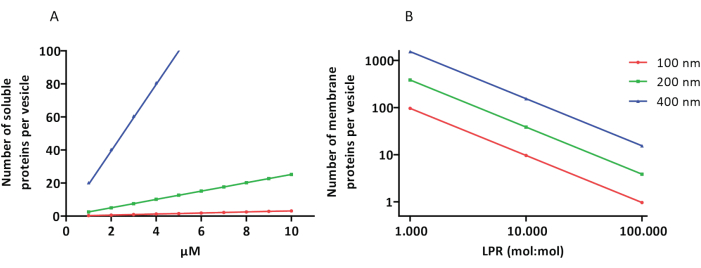
Рисунок 3: Количество молекул в везикуле диаметром 100, 200 или 400 нм. (А) Когда инкапсулированные белки (ферменты, зонды) находятся в диапазоне от 1 до 10 мкМ. (В) Восстановление осуществляется при концентрации от 1 до 1 000, от 1 до 10 000 и от 1 до 100 000 мембранных белков на липид (моль/моль). Мы исходим из предположения, что молекулы инкапсулируются в указанных концентрациях и встраиваются в мембрану при этих соотношении белка к липидам. Что касается некоторых ферментов, то мы видели, что они связываются с мембранами, что может увеличивать их видимую концентрацию в везикулах. Аббревиатура: LPR = Lipid-Protein-Ratio Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
Protocol
Representative Results
Discussion
Мы представляем протокол синтеза (мембранного) белка, содержащего липидные везикулы субмикронного размера (proteoLUVs), и превращения proteoLUV в гигантско-униламеллярные везикулы (proteoGUVs). Протокол должен быть применим для восстановления других мембранных белков 13,19,30,40 и инкапсуляции метаболи?…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Авторы благодарят Адитью Айер за клонирование гена pBAD-PercevalHR и Геа Шуурман-Уолтерс за помощь в производстве и очистке белка. Исследование финансировалось в рамках программы NWO Gravitation «Построение синтетической клетки» (BaSyC).
Materials
| Agarose | Sigma Aldrich | A9414-25g | |
| Amicon cut-off filter | Sigma Aldrich | Milipore centrifugal filter units Amicon Ultra | |
| BioBeads | BioRad | 152-3920 | |
| CHCl3 | Macron Fine Chemicals | MFCD00000826 | |
| D(+)-Glucose | Formedium | – | |
| D(+)-Sucrose | Formedium | – | |
| DDM | Glycon | D97002 -C | |
| Diethyl Ether | Biosolve | 52805 | |
| DMSO | Sigma-Aldrich | 276855-100ml | |
| DOPC | Avanti | 850375P-1g | |
| DOPE | Avanti | 850725P-1g | |
| DOPG | Avanti | 840475P-1g | |
| DTT | Formedium | DTT005 | |
| EtOH | J.T.Baker Avantor | MFCD00003568 | |
| Extruder | Avestin Inc | LF-1 | |
| Fluorimeter | Jasco | Spectrofluorometer FP-8300 | |
| Glycerol | BOOM | 51171608 | |
| Gravity flow column | Bio-Rad | 732-1010 | |
| Hamilton syringe 100 µL | Hamilton | 7656-01 | |
| Hamilton syringe 1000 µL | Hamilton | 81320 | |
| Handheld LCP dispenser | Art Robbins Instruments | 620-411-00 | |
| Handheld Sonicator | Hielscher Ultrasound Technology | UP50H | |
| HCl | BOOM | x76021889.1000 | |
| Imidazole | Roth | X998.4-250g | |
| K2HPO4 | Supelco | 1.05099.1000 | |
| KCl | BOOM | 76028270.1 | |
| KH2PO4 | Supelco | 1.04873.1000 | |
| Kimwipe | Kimtech Science | 7552 | |
| Large Falcon tube centrifuge | Eppendorf | Centrifuge 5810 R | |
| L-Arginine | Sigma-Aldrich | A5006-100G | |
| Light microscope | Leica | DM LS2 | |
| L-Ornithine | Roth | T204.1 | |
| LSM Laser Scanning Confocal Microscope | Zeiss | LSM 710 ConfoCor 3 | |
| MgCl2 | Sigma-Aldrich | M2670-1KG | |
| Microfluidic chip | Homemade | PDMS based | DOI: https://doi.org/10.1039/C8LC01275J |
| Na-ADP | Sigma-Aldrich | A2754-1G | |
| NaCl | Supelco | 1.06404.1000 | |
| Nanodrop Spectrometer | Isogen Life Science | ND-1000 spectrophotometer NanoDrop | |
| NaOH | Supelco | 1.06498.1000 | |
| Needles for GUVs | Henke-Ject | 14-14575 | 27 G x 3/4'' 0.4 x 20 mm |
| Needles for microfluidics | Henke-Ject | 14-15538 | 18 G x 1 1/2'' 1.2 x 40 mm |
| Ni2+ Sepharose | Cytiva | 17526802 | |
| Nigericin | Sigma-Aldrich | N7143-5MG | |
| Nutator | VWR | 83007-210 | |
| Osmolality meter | Gonotec Salmenkipp | Osmomat 3000 basic freezing point osmometer | |
| Plasmacleaner | Plasma Etch | PE-Avenger | |
| Polycarbonate filter | Cytiva Whatman | Nuclepor Track-Etch Membrane Product: 10417104 | 0.4 µm |
| Polycarbonate ultracentrifuge tube | Beckman Coulter | 355647 | |
| Pyranine | Acros Organics | H1529-1G | |
| Quartz cuvette (black) | Hellma Analytics | 108B-10-40 | |
| Sephadex G-75 resin | GE Healthcare | 17-0050-01 | |
| Sonicator | Sonics Sonics & Materials INC | Sonics vibra cell | |
| Syringe filter | Sarstedt | Filtropur S plus 0.2 | 0.2 µm |
| Syringe pump | Harvard Apparatus | A-42467 | |
| Tabletop centrifuge | Eppendorf | centrifuge 5418 | |
| Teflon spacer | Homemade | Teflon based | 45 x 26 x 1.5 or 45 x 26 x 3 or 20 x 20 x 3 mm |
| Tris | PanReac AppliChem | A1086.1000 | |
| Triton X-100 | Sigma Aldrich | T8787-100 ml | |
| Ultracentrifuge | Beckman Coulter | Optima Max-E | |
| UV lamp | Spectroline | ENB-280C/FE | |
| UV/VIS Spectrometer | Jasco | V730 spectrophotometer | |
| Valinomycin | Sigma-Aldrich | V0627-10MG | |
| Widefield fluorescence microscope | Zeiss | AxioObserver | |
| β-Casein | Sigma Aldrich | C5890-500g |
References
- Hirschi, S., Ward, T. R., Meier, W. P., Müller, D. J., Fotiadis, D. Synthetic biology: bottom-up assembly of molecular systems. Chem Rev. 122 (21), 16294-16328 (2022).
- Ivanov, I., et al. Bottom-up synthesis of artificial cells: recent highlights and future challenges. Annu Rev Chem Biomol. Eng. 12 (1), 287-308 (2021).
- Clomburg, J. M., Crumbley, A. M., Gonzalez, R. Industrial biomanufacturing: The future of chemical production. Science. 355 (6320), (2017).
- Shi, T., Han, P., You, C., Zhang, Y. -. H. P. J. An in vitro synthetic biology platform for emerging industrial biomanufacturing: Bottom-up pathway design. Synth Syst Biotechnol. 3 (3), 186-195 (2018).
- Wang, A., et al. Liver-target and glucose-responsive polymersomes toward mimicking endogenous insulin secretion with improved hepatic glucose utilization. Adv Funct Mater. 30 (13), 1910168 (2020).
- Kanter, G., et al. Cell-free production of scFv fusion proteins: an efficient approach for personalized lymphoma vaccines. Blood. 109 (8), 3393-3399 (2007).
- Zeltins, A. Construction and characterization of virus-like particles: a review. Mol Biotechnol. 53 (1), 92-107 (2013).
- Jain, K. K. Synthetic biology and personalized medicine. Med Princ Pract. 22 (3), 209-219 (2013).
- Schwille, P., Frohn, B. P. Hidden protein functions and what they may teach us. Trends Cell Biol. 32 (2), 102-109 (2022).
- Sachsenmeier, P. Industry 5.0-The relevance and implications of bionics and synthetic biology. Engineering. 2 (2), 225-229 (2016).
- Schmidt, D., Jiang, Q. -. X., MacKinnon, R. Phospholipids and the origin of cationic gating charges in voltage sensors. Nature. 444 (7120), 775-779 (2006).
- Godoy-Hernandez, A., et al. Rapid and highly stable membrane reconstitution by LAiR enables the study of physiological integral membrane protein functions. ACS Cent Sci. 9 (3), 494-507 (2023).
- Sikkema, H. R., et al. Gating by ionic strength and safety check by cyclic-di-AMP in the ABC transporter OpuA. Sci Adv. 6 (47), 7697 (2020).
- Foucaud, C., Poolman, B. Lactose transport system of Streptococcus thermophilus. Functional reconstitution of the protein and characterization of the kinetic mechanism of transport. J Biol Chem. 267 (31), 22087-22094 (1992).
- Yoneda, J. S., Sebinelli, H. G., Itri, R., Ciancaglini, P. Overview on solubilization and lipid reconstitution of Na,K-ATPase: enzyme kinetic and biophysical characterization. Biophys Rev. 12 (1), 49-64 (2020).
- Simidjiev, I., et al. Self-assembly of large, ordered lamellae from non-bilayer lipids and integral membrane proteins in vitro. Proc Natl Acad Sci. 97 (4), 1473-1476 (2000).
- Harris, N. J., Booth, P. J. Folding and stability of membrane transport proteins in vitro. Biochim Biophys Acta BBA – Biomembr. 1818 (4), 1055-1066 (2012).
- Jackson, M. L., Litman, B. J. Rhodopsin-egg phosphatidylcholine reconstitution by an octyl glucoside dilution procedure. Biochim Biophys Acta BBA – Biomembr. 812 (2), 369-376 (1985).
- Geertsma, E. R., Nik Mahmood, N. A. B., Schuurman-Wolters, G. K., Poolman, B. Membrane reconstitution of ABC transporters and assays of translocator function. Nat Protoc. 3 (2), 256-266 (2008).
- Rigaud, J. -. L., Pitard, B., Levy, D. Reconstitution of membrane proteins into liposomes: application to energy-transducing membrane proteins. Biochim Biophys Acta BBA – Bioenerg. 1231 (3), 223-246 (1995).
- Szoka, F., Papahadjopoulos, D. Procedure for preparation of liposomes with large internal aqueous space and high capture by reverse-phase evaporation. Proc Natl Acad Sci. 75 (9), 4194-4198 (1978).
- . Synthetic Organelles for Energy Conservation and Delivery of Building Blocks for Lipid Biosynthesis Available from: https://www.researchsquare.com/article/rs-3385355/v1 (2023)
- Lee, K. Y., et al. Photosynthetic artificial organelles sustain and control ATP-dependent reactions in a protocellular system. Nat Biotechnol. 36 (6), 530-535 (2018).
- Méléard, P., Bagatolli, L. A., Pott, T. Giant unilamellar vesicle electroformation. Methods in Enzymology. , 161-176 (2009).
- Garten, M., Aimon, S., Bassereau, P., Toombes, G. E. S. Reconstitution of a transmembrane protein, the voltage-gated ion channel, KvAP, into giant unilamellar vesicles for microscopy and patch clamp studies. J. Vis. Exp. (95), e52281 (2015).
- Doeven, M. K., et al. lateral mobility and function of membrane proteins incorporated into giant unilamellar vesicles. Biophys J. 88 (2), 1134-1142 (2005).
- Pols, T., et al. A synthetic metabolic network for physicochemical homeostasis. Nat Commun. 10 (1), 4239 (2019).
- Bailoni, E., Poolman, B. ATP recycling fuels sustainable glycerol 3-phosphate formation in synthetic cells fed by dynamic dialysis. ACS Synth Biol. 11 (7), 2348-2360 (2022).
- Van Der Heide, T. On the osmotic signal and osmosensing mechanism of an ABC transport system for glycine betaine. EMBO J. 20 (24), 7022-7032 (2001).
- Van’T Klooster, J. S., et al. Membrane lipid requirements of the lysine transporter Lyp1 from Saccharomyces cerevisiae. J Mol Biol. 432 (14), 4023-4031 (2020).
- Lou, G., Anderluzzi, G., Woods, S., Roberts, C. W., Perrie, Y. A novel microfluidic-based approach to formulate size-tuneable large unilamellar cationic liposomes: Formulation, cellular uptake and biodistribution investigations. Eur J Pharm Biopharm. 143, 51-60 (2019).
- Weiss, M., et al. Sequential bottom-up assembly of mechanically stabilized synthetic cells by microfluidics. Nat Mater. 17 (1), 89-96 (2018).
- Pols, T., Singh, S., Deelman-Driessen, C., Gaastra, B. F., Poolman, B. Enzymology of the pathway for ATP production by arginine breakdown. FEBS J. 288 (1), 293-309 (2021).
- Yandrapalli, N., Robinson, T. Ultra-high capacity microfluidic trapping of giant vesicles for high-throughput membrane studies. Lab Chip. 19 (4), 626-633 (2019).
- Elias, M., et al. Microfluidic characterization of biomimetic membrane mechanics with an on-chip micropipette. Micro Nano Eng. 8, 100064 (2020).
- Robinson, T., Kuhn, P., Eyer, K., Dittrich, P. S. Microfluidic trapping of giant unilamellar vesicles to study transport through a membrane pore. Biomicrofluidics. 7 (4), 044105 (2013).
- Cooper, A., Girish, V., Subramaniam, A. B. Osmotic Pressure Enables High-Yield Assembly of Giant Vesicles in Solutions of Physiological Ionic Strengths. Langmuir. 39 (15), 5579-5590 (2023).
- Tantama, M., Martínez-François, J. R., Mongeon, R., Yellen, G. Imaging energy status in live cells with a fluorescent biosensor of the intracellular ATP-to-ADP ratio. Nat Commun. 4 (1), 2550 (2013).
- Setyawati, I., et al. In vitro reconstitution of dynamically interacting integral membrane subunits of energy-coupling factor transporters. eLife. 9, e64389 (2020).
- Oropeza-Guzman, E., Ríos-Ramírez, M., Ruiz-Suárez, J. C. Leveraging the coffee ring effect for a defect-free electroformation of giant unilamellar vesicles. Langmuir. 35 (50), 16528-16535 (2019).
- Estes, D. J., Mayer, M. Electroformation of giant liposomes from spin-coated films of lipids. Colloids Surf B Biointerfaces. 42 (2), 115-123 (2005).

.
