나노미터 및 마이크로미터 크기의 소포에서 평형을 벗어난 대사 네트워크 구축
Summary
우리는 막 단백질을 재구성하고 효소 및 기타 수용성 구성 요소를 마이크로미터 미만 및 마이크로미터 크기의 지질 소포에 캡슐화하기 위한 프로토콜을 제시합니다.
Abstract
정제된 구성 요소를 사용하여 통합 막 단백질, 효소 및 형광 기반 센서를 포함하는 복잡한 단백질 네트워크를 소포에 통합하는 방법을 제시합니다. 이 방법은 생물 반응기의 설계 및 건설과 복잡한 평형 외 대사 반응 네트워크 연구와 관련이 있습니다. 우리는 이전에 개발된 프로토콜에 따라 (다중) 막 단백질을 큰 단층 소포(LUV)로 재구성하는 것으로 시작합니다. 그런 다음 동결-해동-압출을 통해 정제된 효소, 대사 산물 및 형광 기반 센서(형광 단백질 또는 염료)의 혼합물을 캡슐화하고 원심분리 및/또는 크기 배제 크로마토그래피를 통해 통합되지 않은 구성 요소를 제거합니다. 대사 네트워크의 성능은 형광 판독을 통해 ATP/ADP 비율, 대사 산물 농도, 내부 pH 또는 기타 매개변수를 모니터링하여 실시간으로 측정됩니다. 100-400 nm 직경의 막단백질 함유 소포는 기존의 최적화된 절차를 사용하여 거대 단층 소포(GUV)로 전환할 수 있습니다. 이 접근법을 사용하면 용해성 구성 요소(효소, 대사 산물, 센서)를 마이크로미터 크기의 소포에 포함할 수 있으므로 생물반응기의 부피를 수십 배로 늘릴 수 있습니다. GUV를 포함하는 대사 네트워크는 광학 현미경으로 분석하기 위해 미세유체 장치에 갇혀 있습니다.
Introduction
상향식 합성생물학 분야는생명공학적 목적3,4 또는 생물의학적 목적5,6,7,8을 위한 (최소) 세포 1,2 및 대사 생물반응기를 구성하는 데 중점을 둡니다. 합성 세포의 구축은 연구자들이 토착 환경을 모방한 잘 정의된 조건에서 (막) 단백질을 연구할 수 있는 고유한 플랫폼을 제공하여 단백질 및 반응 네트워크의 창발적 특성과 숨겨진 생화학적 기능을 발견할 수 있도록 합니다9. 자율적으로 기능하는 합성 세포를 향한 중간 단계로, 대사 에너지 보존, 단백질 및 지질 합성, 항상성과 같은 살아있는 세포의 필수 기능을 포착하는 모듈이 개발되었습니다. 이러한 모듈은 삶에 대한 우리의 이해를 향상시킬 뿐만 아니라 의학8 및 생명공학10 분야에도 응용될 수 있는 잠재력을 가지고 있다.
막관통 단백질은 세포 안팎으로 분자를 운반하고, 신호를 보내고, 환경의 질에 반응하며, 수많은 생합성 역할을 하기 때문에 거의 모든 대사 네트워크의 중심에 있습니다. 따라서, 합성 세포에서 대사 모듈의 엔지니어링은 대부분의 경우 통합 및/또는 말초 막 단백질을 특정 지질과 높은 무결성(낮은 투과성)으로 구성된 막 이중층으로 재구성해야 합니다. 이러한 막 단백질을 다루는 것은 까다로우며 특정 지식과 실험 기술이 필요합니다.
인지질 소포 내에서 막 단백질을 재구성하기 위해 여러 가지 방법이 개발되었으며, 대부분 특정 단백질의 기능11,12, 조절13, 운동 특성14,15, 지질 의존성15,16 및/또는 안정성17을 연구하기 위한 목적으로 개발되었습니다. 이러한 방법에는 지질(18)이 있는 상태에서 세제-가용화 단백질을 수성 매체로 빠르게 희석하는 방법, 세제-가용화 단백질을 세제-불안정화된 지질 소포체와 함께 배양하여 세제를 제거하는 방법, 세제(들)를 폴리스티렌 비드(19)에 흡수하는 방법, 또는 투석 또는 크기 배제 크로마토그래피(20)에 의한 세제의 제거가 포함된다. 유기 용매는 예를 들어 유수계 간기(21)의 형성을 통해 지질 소포를 형성하는 데 사용되었지만, 대부분의 일체형 막 단백질은 이러한 용매에 노출될 때 비활성화됩니다.
우리 실험실에서는 주로 세제 흡수 방법으로 막 단백질을 재구성하여 대형 단층 소포(LUV)를 형성합니다19. 이 방법은 여러 막 단백질의 공동 재구성과 효소, 대사 산물 및 프로브22,23의 소포 내강에 캡슐화를 허용합니다. 막 단백질-함유 LUV는 막 단백질(26)의 완전성을 보존하기 위해 전기형성(24) 또는 겔 보조 팽창(gel-assisted swelling)25 및 특정 조건을 사용하여 수용성 성분의 캡슐화를 유무에 관계없이 거대-단층 소포(GUVs)로 전환될 수 있다.
이 논문은 L-아르기닌을 L-오르니틴으로 분해하여 ATP를 재생하는 평형외 대사 네트워크의 LUV에서 재구성을 위한 프로토콜을 제시합니다27. ATP의 형성은 인지질 합성을 위한 중요한 구성 요소인 글리세롤-3-포스페이트(G3P)의 생성과 관련이 있습니다22,28. 대사 경로는 아르기닌/오르니틴(ArcD)과 G3P/Pi 안티포터(GlpT)라는 두 개의 필수 막 단백질로 구성됩니다. 또한 ATP의 재활용을 위해 3가지 용해성 효소(ArcA, ArcB, ArcC)가 필요하며, GlpK는 L-아르기닌 분해에서 얻은 ATP를 사용하여 글리세롤을 글리세롤 3-인산으로 변환하는 데 사용됩니다(경로의 개략적인 개요는 그림 1 참조). 이 프로토콜은 지질 또는 단백질의 합성 또는 세포 분열을 위한 훨씬 더 복잡한 반응 네트워크의 미래 구축을 위한 좋은 출발점을 나타냅니다. 소포의 지질 조성은 다양한 일체형 막 단백질의 활성을 지원하며, 소포 27,29,30 안팎으로 다양한 분자를 수송하는 데 최적화되어 있다.
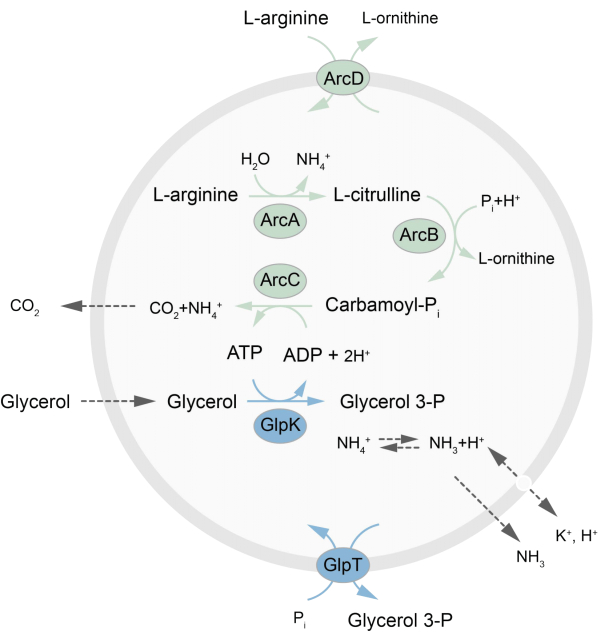
그림 1: ATP 생성과 글리세롤 3-포스페이트 합성 및 배설을 위한 경로 개요. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
요컨대, 정제된 막 단백질(도데실-β-D-말토사이드, DDM에 용해됨)을 Triton X-100으로 불안정화된 미리 형성된 지질 소포에 첨가하여 단백질을 막에 삽입할 수 있습니다. 세제 분자는 이후에 활성 폴리스티렌 비드를 첨가하여 (천천히) 제거되어 잘 밀봉된 프로테올리포좀이 형성됩니다. 그런 다음 용해성 성분을 소포에 추가하고 동결-해동 주기를 통해 캡슐화할 수 있으며, 이는 막 융합 과정에서 분자를 가둡니다. 얻어진 소포는 매우 이질적이며 많은 것이 다층입니다. 그런 다음 공극 크기가 400, 200 또는 100nm인 폴리카보네이트 필터를 통해 압출되어 보다 균일한 크기의 소포가 생성됩니다. 공극 크기가 작을수록 소포가 더 균질하고 단층류이지만 내부 부피가 더 작습니다. 통합되지 않은 단백질과 작은 분자는 크기 배제 크로마토그래피를 통해 외부 용액에서 제거됩니다. proteoLUV는 겔 보조 팽창을 통해 마이크로미터 크기의 소포로 전환될 수 있으며, 이러한 proteoGUV는 현미경 특성 분석 및 조작을 위해 미세유체 칩에 수집되고 포집됩니다. 그림 2 는 전체 프로토콜의 개략적인 개요를 보여줍니다.
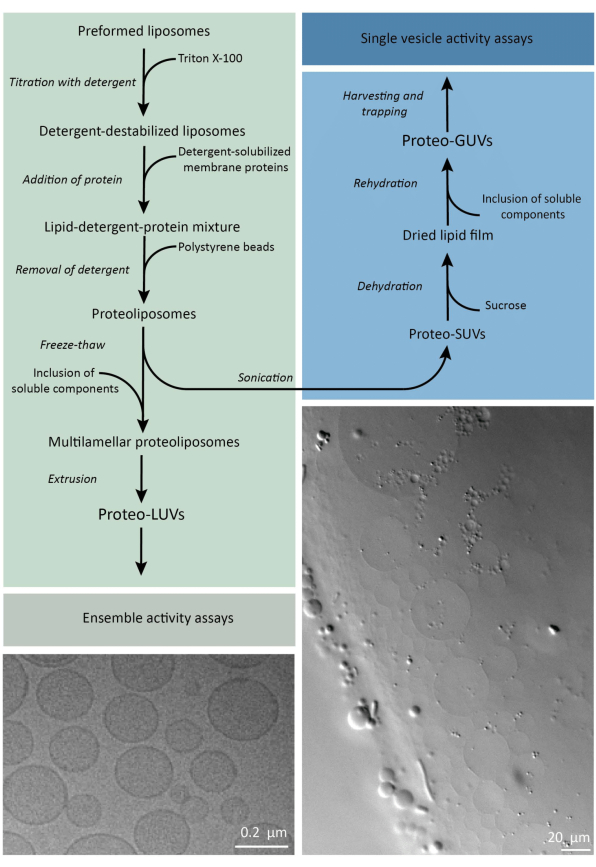
그림 2: 마이크로미터 미만(LUV) 및 마이크로미터 크기(GUV)의 지질 소포에서 막 단백질을 재구성하고 효소 및 수용성 성분을 캡슐화하기 위한 프로토콜 개요. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
재구성 및 캡슐화 프로토콜은 잘 작동하고 단백질의 기능은 유지되지만 proteoLUV와 proteoGUV는 크기가 이질적입니다. 미세유체역학(Microfluidic) 접근법31,32는 크기가 더 균질한 마이크로미터 크기의 소포를 형성할 수 있지만, 이중층의 잔류 용매가 단백질을 비활성화하기 때문에 일반적으로 막 단백질의 기능적 재구성이 불가능합니다. proteoLUV의 크기는 100nm에서 400nm까지 다양하며, 낮은 농도의 효소에서는 캡슐화로 인해 불완전한 대사 경로(확률적 효과, 그림 3 참조)를 가진 소포가 생성될 수 있습니다. LUV는 ATP 및 G3P와 같은 빌딩 블록의 생산을 위해 여기에서 볼 수 있듯이 특정 대사 모듈을 구성하는 데 이상적입니다. 이러한 proteoLUV는 잠재적으로 GUV에 캡슐화될 수 있으며 숙주 소포를 위한 소기관과 같은 구획 역할을 할 수 있습니다.
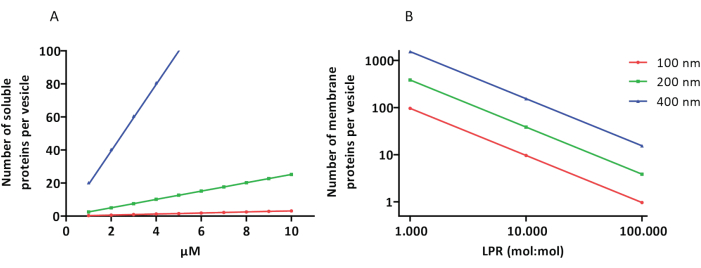
그림 3: 직경이 100, 200 또는 400nm인 소포당 분자 수. (A) 캡슐화된 단백질(효소, 프로브)이 1-10μM 범위에 있을 때. (B) 재구성은 지질당 1 – 1,000개, 1 – 10,000개, 1 – 100,000개의 막 단백질(mol/mol)에서 수행됩니다. 우리는 분자가 표시된 농도로 캡슐화되고 이러한 단백질 대 지질 비율로 막에 통합된다고 가정합니다. 일부 효소의 경우, 우리는 그들이 막에 결합하여 소포의 겉보기 농도를 증가시킬 수 있음을 보았습니다. 약어: LPR = Lipid-Protein-Ratio 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
Protocol
Representative Results
Discussion
마이크로미터 미만의 지질 소포(proteoLUV)를 포함하는 (막) 단백질의 합성과 proteoLUV를 giant-unilamellar vesicles(proteoGUV)로 전환하기 위한 프로토콜을 제시합니다. 이 프로토콜은 다른 막 단백질 13,19,30,40의 재구성 및 여기에 제시된 L-아르기닌 분해 및 글리세롤 3-인산염 합성 경로 이외의 대사 네트워크의 캡슐화에 적용할 수 있어야 합니다.<sup…
Disclosures
The authors have nothing to disclose.
Acknowledgements
저자들은 pBAD-PercevalHR 유전자의 클로닝에 대해 Aditya Iyer에게 감사를 표하고, 단백질 생산 및 정제를 도와준 Gea Schuurman-Wolters에게 감사를 표합니다. 이 연구는 NWO 중력 프로그램인 “Building a Synthetic Cell”(BaSyC)의 자금 지원을 받았습니다.
Materials
| Agarose | Sigma Aldrich | A9414-25g | |
| Amicon cut-off filter | Sigma Aldrich | Milipore centrifugal filter units Amicon Ultra | |
| BioBeads | BioRad | 152-3920 | |
| CHCl3 | Macron Fine Chemicals | MFCD00000826 | |
| D(+)-Glucose | Formedium | – | |
| D(+)-Sucrose | Formedium | – | |
| DDM | Glycon | D97002 -C | |
| Diethyl Ether | Biosolve | 52805 | |
| DMSO | Sigma-Aldrich | 276855-100ml | |
| DOPC | Avanti | 850375P-1g | |
| DOPE | Avanti | 850725P-1g | |
| DOPG | Avanti | 840475P-1g | |
| DTT | Formedium | DTT005 | |
| EtOH | J.T.Baker Avantor | MFCD00003568 | |
| Extruder | Avestin Inc | LF-1 | |
| Fluorimeter | Jasco | Spectrofluorometer FP-8300 | |
| Glycerol | BOOM | 51171608 | |
| Gravity flow column | Bio-Rad | 732-1010 | |
| Hamilton syringe 100 µL | Hamilton | 7656-01 | |
| Hamilton syringe 1000 µL | Hamilton | 81320 | |
| Handheld LCP dispenser | Art Robbins Instruments | 620-411-00 | |
| Handheld Sonicator | Hielscher Ultrasound Technology | UP50H | |
| HCl | BOOM | x76021889.1000 | |
| Imidazole | Roth | X998.4-250g | |
| K2HPO4 | Supelco | 1.05099.1000 | |
| KCl | BOOM | 76028270.1 | |
| KH2PO4 | Supelco | 1.04873.1000 | |
| Kimwipe | Kimtech Science | 7552 | |
| Large Falcon tube centrifuge | Eppendorf | Centrifuge 5810 R | |
| L-Arginine | Sigma-Aldrich | A5006-100G | |
| Light microscope | Leica | DM LS2 | |
| L-Ornithine | Roth | T204.1 | |
| LSM Laser Scanning Confocal Microscope | Zeiss | LSM 710 ConfoCor 3 | |
| MgCl2 | Sigma-Aldrich | M2670-1KG | |
| Microfluidic chip | Homemade | PDMS based | DOI: https://doi.org/10.1039/C8LC01275J |
| Na-ADP | Sigma-Aldrich | A2754-1G | |
| NaCl | Supelco | 1.06404.1000 | |
| Nanodrop Spectrometer | Isogen Life Science | ND-1000 spectrophotometer NanoDrop | |
| NaOH | Supelco | 1.06498.1000 | |
| Needles for GUVs | Henke-Ject | 14-14575 | 27 G x 3/4'' 0.4 x 20 mm |
| Needles for microfluidics | Henke-Ject | 14-15538 | 18 G x 1 1/2'' 1.2 x 40 mm |
| Ni2+ Sepharose | Cytiva | 17526802 | |
| Nigericin | Sigma-Aldrich | N7143-5MG | |
| Nutator | VWR | 83007-210 | |
| Osmolality meter | Gonotec Salmenkipp | Osmomat 3000 basic freezing point osmometer | |
| Plasmacleaner | Plasma Etch | PE-Avenger | |
| Polycarbonate filter | Cytiva Whatman | Nuclepor Track-Etch Membrane Product: 10417104 | 0.4 µm |
| Polycarbonate ultracentrifuge tube | Beckman Coulter | 355647 | |
| Pyranine | Acros Organics | H1529-1G | |
| Quartz cuvette (black) | Hellma Analytics | 108B-10-40 | |
| Sephadex G-75 resin | GE Healthcare | 17-0050-01 | |
| Sonicator | Sonics Sonics & Materials INC | Sonics vibra cell | |
| Syringe filter | Sarstedt | Filtropur S plus 0.2 | 0.2 µm |
| Syringe pump | Harvard Apparatus | A-42467 | |
| Tabletop centrifuge | Eppendorf | centrifuge 5418 | |
| Teflon spacer | Homemade | Teflon based | 45 x 26 x 1.5 or 45 x 26 x 3 or 20 x 20 x 3 mm |
| Tris | PanReac AppliChem | A1086.1000 | |
| Triton X-100 | Sigma Aldrich | T8787-100 ml | |
| Ultracentrifuge | Beckman Coulter | Optima Max-E | |
| UV lamp | Spectroline | ENB-280C/FE | |
| UV/VIS Spectrometer | Jasco | V730 spectrophotometer | |
| Valinomycin | Sigma-Aldrich | V0627-10MG | |
| Widefield fluorescence microscope | Zeiss | AxioObserver | |
| β-Casein | Sigma Aldrich | C5890-500g |
References
- Hirschi, S., Ward, T. R., Meier, W. P., Müller, D. J., Fotiadis, D. Synthetic biology: bottom-up assembly of molecular systems. Chem Rev. 122 (21), 16294-16328 (2022).
- Ivanov, I., et al. Bottom-up synthesis of artificial cells: recent highlights and future challenges. Annu Rev Chem Biomol. Eng. 12 (1), 287-308 (2021).
- Clomburg, J. M., Crumbley, A. M., Gonzalez, R. Industrial biomanufacturing: The future of chemical production. Science. 355 (6320), (2017).
- Shi, T., Han, P., You, C., Zhang, Y. -. H. P. J. An in vitro synthetic biology platform for emerging industrial biomanufacturing: Bottom-up pathway design. Synth Syst Biotechnol. 3 (3), 186-195 (2018).
- Wang, A., et al. Liver-target and glucose-responsive polymersomes toward mimicking endogenous insulin secretion with improved hepatic glucose utilization. Adv Funct Mater. 30 (13), 1910168 (2020).
- Kanter, G., et al. Cell-free production of scFv fusion proteins: an efficient approach for personalized lymphoma vaccines. Blood. 109 (8), 3393-3399 (2007).
- Zeltins, A. Construction and characterization of virus-like particles: a review. Mol Biotechnol. 53 (1), 92-107 (2013).
- Jain, K. K. Synthetic biology and personalized medicine. Med Princ Pract. 22 (3), 209-219 (2013).
- Schwille, P., Frohn, B. P. Hidden protein functions and what they may teach us. Trends Cell Biol. 32 (2), 102-109 (2022).
- Sachsenmeier, P. Industry 5.0-The relevance and implications of bionics and synthetic biology. Engineering. 2 (2), 225-229 (2016).
- Schmidt, D., Jiang, Q. -. X., MacKinnon, R. Phospholipids and the origin of cationic gating charges in voltage sensors. Nature. 444 (7120), 775-779 (2006).
- Godoy-Hernandez, A., et al. Rapid and highly stable membrane reconstitution by LAiR enables the study of physiological integral membrane protein functions. ACS Cent Sci. 9 (3), 494-507 (2023).
- Sikkema, H. R., et al. Gating by ionic strength and safety check by cyclic-di-AMP in the ABC transporter OpuA. Sci Adv. 6 (47), 7697 (2020).
- Foucaud, C., Poolman, B. Lactose transport system of Streptococcus thermophilus. Functional reconstitution of the protein and characterization of the kinetic mechanism of transport. J Biol Chem. 267 (31), 22087-22094 (1992).
- Yoneda, J. S., Sebinelli, H. G., Itri, R., Ciancaglini, P. Overview on solubilization and lipid reconstitution of Na,K-ATPase: enzyme kinetic and biophysical characterization. Biophys Rev. 12 (1), 49-64 (2020).
- Simidjiev, I., et al. Self-assembly of large, ordered lamellae from non-bilayer lipids and integral membrane proteins in vitro. Proc Natl Acad Sci. 97 (4), 1473-1476 (2000).
- Harris, N. J., Booth, P. J. Folding and stability of membrane transport proteins in vitro. Biochim Biophys Acta BBA – Biomembr. 1818 (4), 1055-1066 (2012).
- Jackson, M. L., Litman, B. J. Rhodopsin-egg phosphatidylcholine reconstitution by an octyl glucoside dilution procedure. Biochim Biophys Acta BBA – Biomembr. 812 (2), 369-376 (1985).
- Geertsma, E. R., Nik Mahmood, N. A. B., Schuurman-Wolters, G. K., Poolman, B. Membrane reconstitution of ABC transporters and assays of translocator function. Nat Protoc. 3 (2), 256-266 (2008).
- Rigaud, J. -. L., Pitard, B., Levy, D. Reconstitution of membrane proteins into liposomes: application to energy-transducing membrane proteins. Biochim Biophys Acta BBA – Bioenerg. 1231 (3), 223-246 (1995).
- Szoka, F., Papahadjopoulos, D. Procedure for preparation of liposomes with large internal aqueous space and high capture by reverse-phase evaporation. Proc Natl Acad Sci. 75 (9), 4194-4198 (1978).
- . Synthetic Organelles for Energy Conservation and Delivery of Building Blocks for Lipid Biosynthesis Available from: https://www.researchsquare.com/article/rs-3385355/v1 (2023)
- Lee, K. Y., et al. Photosynthetic artificial organelles sustain and control ATP-dependent reactions in a protocellular system. Nat Biotechnol. 36 (6), 530-535 (2018).
- Méléard, P., Bagatolli, L. A., Pott, T. Giant unilamellar vesicle electroformation. Methods in Enzymology. , 161-176 (2009).
- Garten, M., Aimon, S., Bassereau, P., Toombes, G. E. S. Reconstitution of a transmembrane protein, the voltage-gated ion channel, KvAP, into giant unilamellar vesicles for microscopy and patch clamp studies. J. Vis. Exp. (95), e52281 (2015).
- Doeven, M. K., et al. lateral mobility and function of membrane proteins incorporated into giant unilamellar vesicles. Biophys J. 88 (2), 1134-1142 (2005).
- Pols, T., et al. A synthetic metabolic network for physicochemical homeostasis. Nat Commun. 10 (1), 4239 (2019).
- Bailoni, E., Poolman, B. ATP recycling fuels sustainable glycerol 3-phosphate formation in synthetic cells fed by dynamic dialysis. ACS Synth Biol. 11 (7), 2348-2360 (2022).
- Van Der Heide, T. On the osmotic signal and osmosensing mechanism of an ABC transport system for glycine betaine. EMBO J. 20 (24), 7022-7032 (2001).
- Van’T Klooster, J. S., et al. Membrane lipid requirements of the lysine transporter Lyp1 from Saccharomyces cerevisiae. J Mol Biol. 432 (14), 4023-4031 (2020).
- Lou, G., Anderluzzi, G., Woods, S., Roberts, C. W., Perrie, Y. A novel microfluidic-based approach to formulate size-tuneable large unilamellar cationic liposomes: Formulation, cellular uptake and biodistribution investigations. Eur J Pharm Biopharm. 143, 51-60 (2019).
- Weiss, M., et al. Sequential bottom-up assembly of mechanically stabilized synthetic cells by microfluidics. Nat Mater. 17 (1), 89-96 (2018).
- Pols, T., Singh, S., Deelman-Driessen, C., Gaastra, B. F., Poolman, B. Enzymology of the pathway for ATP production by arginine breakdown. FEBS J. 288 (1), 293-309 (2021).
- Yandrapalli, N., Robinson, T. Ultra-high capacity microfluidic trapping of giant vesicles for high-throughput membrane studies. Lab Chip. 19 (4), 626-633 (2019).
- Elias, M., et al. Microfluidic characterization of biomimetic membrane mechanics with an on-chip micropipette. Micro Nano Eng. 8, 100064 (2020).
- Robinson, T., Kuhn, P., Eyer, K., Dittrich, P. S. Microfluidic trapping of giant unilamellar vesicles to study transport through a membrane pore. Biomicrofluidics. 7 (4), 044105 (2013).
- Cooper, A., Girish, V., Subramaniam, A. B. Osmotic Pressure Enables High-Yield Assembly of Giant Vesicles in Solutions of Physiological Ionic Strengths. Langmuir. 39 (15), 5579-5590 (2023).
- Tantama, M., Martínez-François, J. R., Mongeon, R., Yellen, G. Imaging energy status in live cells with a fluorescent biosensor of the intracellular ATP-to-ADP ratio. Nat Commun. 4 (1), 2550 (2013).
- Setyawati, I., et al. In vitro reconstitution of dynamically interacting integral membrane subunits of energy-coupling factor transporters. eLife. 9, e64389 (2020).
- Oropeza-Guzman, E., Ríos-Ramírez, M., Ruiz-Suárez, J. C. Leveraging the coffee ring effect for a defect-free electroformation of giant unilamellar vesicles. Langmuir. 35 (50), 16528-16535 (2019).
- Estes, D. J., Mayer, M. Electroformation of giant liposomes from spin-coated films of lipids. Colloids Surf B Biointerfaces. 42 (2), 115-123 (2005).

