シリアル希釈とめっき:微生物列挙
English
Share
Overview
ソース: ジョナサン・F・ブレイズ1,エリザベス・スーター1,クリストファー・P・コルボ1
1ワーグナーカレッジ生物科学科、1キャンパスロード、スタテンアイランドニューヨーク、10301
原核生物の定量的評価は、その豊富さ、指数関数的増殖の傾向、集団内の種の多様性、および特定の生理学的ニーズを考えると、厄介なことができます。この課題を複雑にすることは、細菌が複製する4相の性質(ラグ、ログ、静止および死)です。微生物の濃度を正確に推定する能力は、同定、単離、栽培、および特性評価を成功させるために必要である(6)。そのため、微生物学者は、臨床、産業、製薬、学術実験室環境(2,4,6)において、細菌およびウイルス負荷を確実に定量化するために、1世紀以上にわたってシリアル希釈および様々なめっき技術を採用してきました。この方法論の説明は、ドイツの科学者で医師のロバート・コッホが感染症を引き起こす薬剤に関する研究を発表した1883年に最初に現れました(2)。現代の細菌学の父と呼ばれることが多いコッホの前述の技術は、世界中の微生物の列挙のゴールドスタンダードとなっています。
シリアル希釈は、初期溶液(溶液0)を液体希釈剤(ブランク)の固定体積に連続的に再懸濁して、既知または未知の実体(溶質、生物など)の系統的な減少である。これらのブランクは通常0.45%の生理生理で構成されるが、組成物は変化させることができる(7)。実験者は、各希釈剤に対して任意の体積を選択できますが、ほとんどの場合、10の倍数であり、サンプルの対数減少を容易にします。例えば、溶液0は、10mLの栄養スープに懸濁した合計100個の大腸菌細胞を含む。溶液0の1mLを除去し、9 mLの生理生理物(希釈剤1)に添加した場合、新しい溶液(溶液1)は大腸菌の初期濃度の1/10分の1を含むであろう。この例では、新しい溶液(解1)には10の大腸菌細胞が含まれる。このプロセスを1mLの溶液1を除去し、別の9mLの生理塩分(希釈剤2)に添加することで、1つの大腸菌細胞のみを含む溶液2が得られます。各新しい溶液(希釈液9mL+溶液1mL)は合計10mLを含むので、この還元の希釈係数は10個であるか、またはこれが10倍の連続希釈であったと結論付けることができます(図1)。この例では 100 個のセルから始まり、10 倍の希釈を行っているため、1 セルの絶対最小濃度に達するには 2 つのステップのみが必要です。
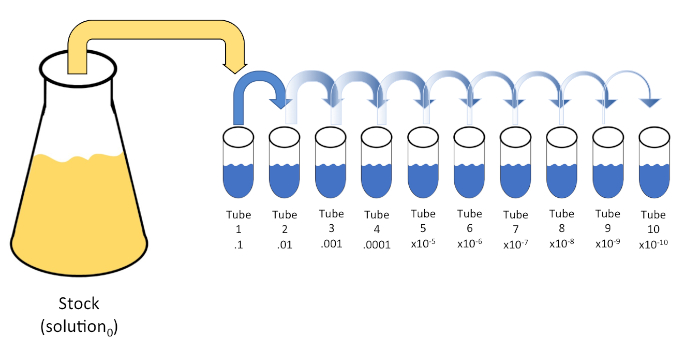
図 1: ストック ソリューションのシリアル希釈。ストック溶液(溶液0)の1 mLアリコートを、0.45%生理塩水の9 mLを含むチューブ1に添加する(希釈1)。この混合物の積は溶液1である。新しく作成した溶液1の1 mLをアリコートし、チューブ2に加えて繰り返します。アリクォートとリサスペンションは、最終的なチューブに達するまでこの方法で継続し、各ステップでそれぞれ10倍の10倍のストック濃度を希釈します。この図のより大きなバージョンを表示するには、ここをクリックしてください。
シリアル希釈は、所望の生物の管理可能な濃度を得るための最も簡単な技術であり、それは、微生物学者によって使用される多くのめっき技術のちょうど2つ、ペトリ皿のストリークと広がりによって補完されます。このアプローチのこの利点は、実験者が単一種の純粋な株を収穫したり、混合集団から別株を分離する(7)。ストリーキングは、適切な栄養素が利用可能な場合に成長する固体培地(一般的にアガロースからなる)に生物を導入することによって達成されます。硬い正弦波パターンで培地を通して無菌接種ループを静かに掃引し、実験者の波形の周波数に比例して生物を分配する。ペトリ皿を3分の1または4分の1(象限ストリーク)に分割し、皿の新しい領域が入力されるにつれて各ストリークの頻度を減らすことは、徐々にその領域を占有できる微生物の数を減らし、単一のコロニーを生成します。定量化できない細菌の芝生。スプレッドメッキは、サンプルを追加的に希釈しません。滅菌ガラス拡散機は、ペトリ皿全体に懸濁液媒体のアリコートを配布するために使用されます(図2)。スプレッドプレート上で成長するコロニーは、単一のセルから生じ、皿上の各コロニーは、CFU/mL(6)(図3)ソフト寒天およびレプリカとして表される所定の懸濁液中の1ミリリットル当たりのコロニー形成単位数を推定するためにカウントすることができる。めっきは、前述の技術のバリエーションであり、バクテリオファージおよび変異スクリーニングの分離を可能にする(1,7)。
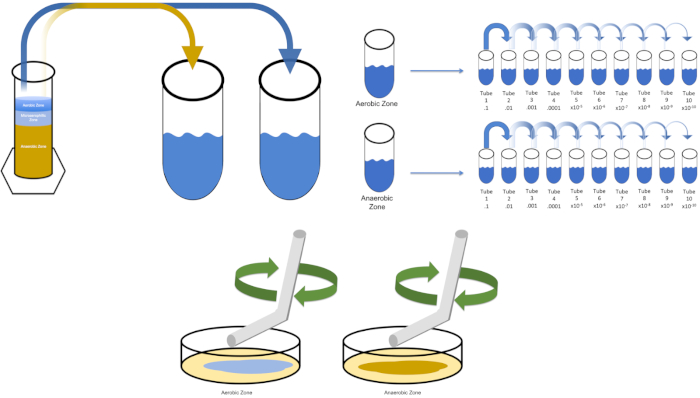
図2:細菌列挙および歪み分離のためのプレートストリーキング。ペトリ皿の底に識別情報(細菌名、日付、メディア)をラベル付けし、3分の1に分けます。ストックサンプルの適切な希釈を選択した後、無菌(使い捨てまたは炎)接種ループを取り、それを試験管(ここでは、T3)に沈めます。片側のペトリ皿カバーを少し上げて、接種ループだけが寒天にアクセスできるようにします。寒天を危険にさらさないために注意しているジグザグの方法でメディアの上部を横切って接種ループを滑空します。プレートを約1/3rd(〜118°)回転させ、ジグザグ運動の周波数を下げます。最後の時間を回転させ、ジグザグの周波数をもう一度下します。この図のより大きなバージョンを表示するには、ここをクリックしてください。
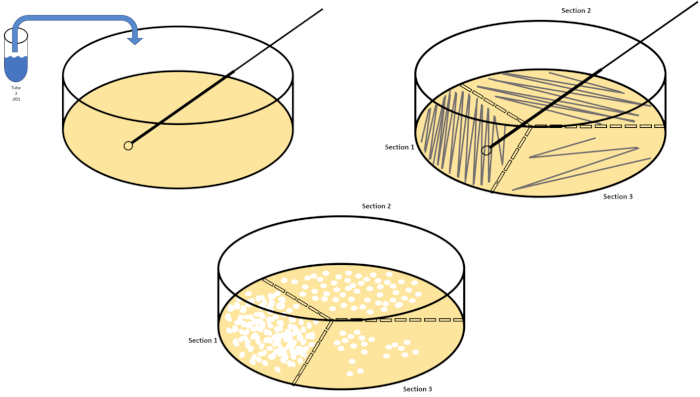
図3:スプレッドメッキ。好気性ゾーンの1gをT1で再懸濁し、次いで連続的に希釈した。滅菌ガラスまたはプラスチックの使い捨て可能な広がる棒は各皿に接種を配るために使用される。これを嫌気性ゾーンの1gで繰り返した。この図のより大きなバージョンを表示するには、ここをクリックしてください。
連続希釈と同様に、対数スケールは、組織の濃度を表すために使用されます。100mm x15mmを測定する標準的なペトリ皿で成長したコロニーの数は、成長の孤立したクラスターを識別することによって手動で(または計算処理の助けを借りて自動化される)列挙することができる。合計が 30 未満または 300 未満のカウントは、それぞれカウントする数が少なすぎる (TFTC) またはカウントが多すぎる (TNTC) と定義する必要があります。後者の場合、新しいペトリ皿を再ストリークする前に濃度を下げるシリアル希釈を行う必要があります。3つの別々のペトリ皿から識別された自己完結型コロニーの数を平均し、希釈係数によって平均を乗算すると、CFU/mLが得られます。CFU/mLのログ10を時間に対してプロットすると、生物の平均生成時間が明らかになる(7)。
Procedure
Applications and Summary
Bacterial enumeration and strain isolation by plating requires manageable concentrations of target organisms. Successful plating is therefore contingent upon serial dilution. As such, the aforementioned techniques remain the cornerstone of microbiological examination and experimentation. Though simple by design, dilution factors and plating technique can be modified to by the experimenter to bolster outcomes without compromising the integrity of each method. Plotting the four phases of bacterial growth can be helpful when characterizing desired microbes. These phases, lag, log, stationary, and death, are marked by changes in bacterial replication. The lag phase features slow growth due to physiological adaptation, the log phase is the period of maximum proliferation featuring an exponential rise in viable cells, stationary phase is then reached due to environmental limitations and accumulations of toxins, before the death phase where cell counts begin to fall. This can be accomplished by serially diluting (or 1-step diluting to avoid confusion) Solution0 every hour for a total of 8 hours, beginning at Time0 (Solution0 should be returned to a shaking incubator after each dilution). Calculate the log10 of CFU/ml for a single diluent of Time0 and plot on the Y-axis. Repeat this calculation for the sample Time1 (make sure calculate CFU/mL using the same dilution factor as Time0). Repeat until each time (Time1-Time8) are plotted on the X-axis.
References
- Allen, M.E., Gyure, R.A. (2013) An Undergraduate Laboratory Activity Demonstrating Bacteriophage Specificity. Journal of Microbial Biological Education 14: 84-92.
- Ben-David, A., Davidson, C.E. (2014) Estimation Method for Serial Dilution Experiments. Journal of Microbiological Methods 107:214-221.
- Goldman, E., Green, L.H. (2008) Practical Handbook of Microbiology.
- Koch, R. (1883) New Research Methods for Detection of Microcosms in Soil, Air and Water.
- Lederberg, J., Lederberg, E.M. (1952) Replica Plating and Indirect Selection of Bacterial Mutants. Journal of Bacteriology 63:399-406
- Pepper, I., Gerba, C., Ikner, L. (2019) Bacterial Growth Curve Analysis and its Environmental Changes. JoVE Science Education Database. Environmental Microbiology.
- Sanders., E.R. (2012) Aseptic Laboratory Technique: Plating Methods. JoVE 63:e3063.
Transcript
Sometimes, in order to identify and study bacteria we first need to isolate and enrich them from a sample. For example, samples obtained from a Winogradsky Column are mixed, meaning they contain multiple species or strains of bacteria, so studying an individual bacterium or enumerating the different kinds present can be challenging. To this end, serial dilution and plating techniques are typically employed to reliably quantify bacterial load and isolate individual colonies.
Serial dilution is a process through which the concentration of an organism, bacteria in this example, is systematically reduced through successive resuspension in fixed volumes of liquid diluent. Usually the volume of the diluent is a multiple of 10 to facilitate logarithmic reduction of the sample organism. For example, one gram of sediment is first removed from the Winogradsky zone of interest and added to 10 milliliters of an appropriate liquid medium. Then, one milliliter of this first dilution is added to another tube containing nine milliliters of medium. The process can be repeated until several different concentrations of bacteria have been prepared. Serial dilution is the key to enumeration of bacteria in this example, since mixed samples from a Winogradsky Column contain an unknown, often large, number of bacteria.
Next, streak plating and spread plating enable the isolation and enumeration of bacteria within a sample, respectively. Streaking is accomplished by introducing a diluted sample to one section of the solid medium supplemented with nutrient, which is divided into thirds. This inoculum is then spread over each third of the plate in a zig-zag pattern. As different sections of the plate are streaked, crossing from the previous sample only once, the sample is spread more thinly. This means that you may only need to streak from one dilution to achieve individual colonies in the later sections. After incubation, the streaked plates allow for observations of colony morphology, information that can help differentiate between different bacterial species.
Alternatively, if the main goal is the enumeration of the bacteria in the sample spread plating may be used. In spread plating, an aliquot of a single sample is spread evenly over the entire surface of solid medium. Typically, because we don’t know the bacterial numbers in the mixed sample, a spread plate is made for each of the dilutions or a representative sample of them. After incubation, enumeration can be performed using these spread plates. Any plates with colony counts fewer than 30 should be discarded, since small counts are subject to greater error. Similarly, any counts over 300 should be discarded because colony crowding and overlapping can lead to underestimation of colony count. If the colony counts of each of these remaining dishes is recorded and multiplied by the dilution factor, and then divided by the volume plated, this yields the colony forming units, or CFUs, per milliliter of suspension. In this video, you will learn how to qualitatively and quantitatively evaluate a sample containing a known bacterium, and the microbial communities contained in various regions of a Winogradsky Column via serial dilution, spread plating, and streak plating.
First, put on any appropriate personal protective equipment including a lab coat, gloves, and goggles. Next, sterilize the workspace with 70% ethanol and wipe down the surface. Next, gather two 500 milliliter Erlenmeyer flasks and label one broth and the other agar. To prepare LB agar solution, mix approximately 6.25 grams of LB agar, three grams of technical agar, and 250 milliliters of distilled water in the flask labeled agar.
Then, prepare LB broth by combining 2. 5 grams of LB media and 100 milliliters of distilled water in the flask labeled broth. After autoclaving the flasks, use a heat resistant glove to remove the flasks from the autoclave and place them in a 40 to 50 degree Celsius water bath. Once the flasks are 50 degrees Celsius, carefully prepare three 100 milliliter aliquots of the broth solution and label each aliquot solution zero. Next, gather 10 sterile petri dishes and label them with the date, name, type of media used, and the Winogradsky Column zone that the organisms will be harvested from. Pipette 15 milliliters of agar from the agar flask into each petri dish. Then, use the pipette tip to remove any bubbles, replace the plate lids, and allow them to solidify on the bench top overnight.
The next day, wipe down the bench top with 70% ethanol. Next, label 10 20 milliliter test tubes T1 through T10 and place them in a rack. Pipette nine milliliters of .45% saline into each tube. Now, cover each of the 10 test tubes loosely with their caps and transfer them to an autoclave-compatible test tube rack. After the cycle is complete, remove the saline blanks using heat resistant gloves and allow them to cool. Store the tubes at room temperature until they have reached approximately 22 degrees Celsius.
To cultivate a known target organism, E. coli in this example, inoculate 100 milliliters of solution zero with a single colony from a previously streaked plate. Then, cover the tube and incubate it over night at 37 degrees Celsius. To evaluate the regions of a Winogradsky Column, add approximately one gram of material from the aerobic zone to T1 and resuspend by vortexing. Then, repeat this process with one gram of material from the anaerobic zone.
Remove the tube containing solution zero inoculated with E. coli from the incubator and shake it. Then, pipette one milliliter of the solution into a T1 test tube and vortex to mix. Remove one milliliter of solution from T1 and transfer it to T2, vortexing to mix. Repeat this process through tube T10. To evaluate the aerobic and anaerobic zones of the Winogradsky Column, remove one milliliter of solution from each of the previously prepared T1 tubes and transfer it to the appropriate T2 tubes. Then, continue the serial dilutions through the T10 tubes as previously demonstrated.
To spread plate, pipette 100 microliters of the diluted sample from each T3 tube on to the corresponding petri dish. Then, use a sterile spreading rod to gently distribute the sample on to the petri dish and replace the plate lid. Repeat this process for the T6 and T9 dilutions, as previously demonstrated. Incubate the plates containing aerobic organisms in a 37 degree Celsius incubator for 24 hours. Incubate the plates containing anaerobic organisms in an anaerobic chamber set to 37 degrees Celsius for 24 hours. The next day remove the T3, T6, and T9 dilution plates from the incubator and the anaerobic chamber and transfer them to the bench top. Working with one plate at a time, glide a sterile inoculating loop across the top of the media in a zig-zag pattern. Then, replace the petri dish lid. Next, rotate the plate by 1/3 and sterilize the loop to reduce the frequency of the previously made zig-zag pattern. Again, after sterilizing the loop, rotate the plate by 1/3, reduce the frequency of the zig-zag pattern one last time, and replace the lid. Repeat this streaking method for the remaining plates, as previously shown. Then, place the streaked plates containing aerobic organisms in a 37 degree Celsius incubator overnight and the streaked plates containing anaerobic organisms in an anaerobic chamber set to 37 degrees Celsius overnight.
Cultures were harvested from the aerobic and anaerobic zones of a seven day Winogradsky Column. Then, the cultures were serially diluted prior to streaking and spreading on LB agar plates. Streaking revealed a mixed population from each of the evaluated Winogradsky zones, and the spread plates produced similar results. A plate streaked from a mixed population will result in bacterial colonies of different shapes, sizes, textures, and colors. In contrast, the streaked and spread plates containing the known organism, E. coli, demonstrated a homologous population. Generally, it is best to calculate CFUs per milliliter using the average colony count of three plates spread with the same sample and dilution factor. Multiply the average number of colonies by the dilution factor and divide by the amount aliquoted. Finally, isolated colonies chosen from each plate can be used in further enrichment assays to determine species identity.
