연속 희석 및 플레이팅: 미생물 나열
English
Share
Overview
출처: 조나단 F. 블레이즈1,엘리자베스 수터1,크리스토퍼 P. 코르보1
1 생물과학부, 바그너 칼리지, 1 캠퍼스 로드, 스태튼 아일랜드 뉴욕, 10301
원생동물의 정량적 평가는 그들의 풍부함, 기하급수적 증식을 위한 성향, 인구 내의 종 다양성 및 특정 생리적 필요를 감안할 때 번거로울 수 있습니다. 이 도전을 복합하는 것은 박테리아가 복제하는 4 상 성질입니다 (지연, 로그, 고정 및 죽음). 미생물 농도를 정확하게 추정하는 능력은 성공적인 식별, 격리, 재배 및 특성화(6)에 필요합니다. 이와 같이, 미생물학자는 임상, 산업, 제약 및 학술 실험실 환경 (2,4,6)에서 세균및 바이러스 부하를 안정적으로 정량화하기 위해 100 년 이상 직렬 희석 및 다양한 도금 기술을 사용했습니다. 이 방법론의 설명은 독일 과학자이자 의사인 로버트 코흐(Robert Koch)가 전염병 유발 요원(2)에 대한 그의 작품을 발표했을 때 1883년에 처음 나타났습니다. 종종 현대 세균학의 아버지로 불리는 코흐의 외명 기술은 전 세계적으로 미생물을 열거하는 금본위제가 되었습니다.
직렬 희석은 초기 용액(솔루션0)을액체 희석제(blank)의 고정 부수로 연속적으로 재정지하여 공지또는 알려지지 않은 실체(solute, 유기체 등)를 체계적으로 감소시다. 이러한 공백은 일반적으로 0.45% 식염수로 구성되지만 조성물은 다를 수 있습니다(7). 실험자는 각 희석제에 대해 임의의 부피를 선택할 수 있지만, 가장 자주 는 10의 배수이며, 샘플의 로그산염 감소를 용이하게 한다. 예를 들어, 용액0은 영양국물의 10mL에 매달린 총 100개의 대장균 세포를 함유하고 있다. 용액0의 1mL이 제거되고 식염수(희석1)의9mL에 추가되면, 새로운 용액(1)은 대장균의초기 농도의 1/10th를 포함하게 된다. 이 예에서, 새로운 용액(솔루션1)에는10개의 대장균 세포가 포함됩니다. 이 과정을 반복하여 1mL의 용액1을 제거하고 또 다른 9mL의 식염수(희석2)에 첨가함으로써 단일 대장균 세포만 을 포함하는 용액2를산출한다. 각각의 새로운 솔루션(희석제 + 1mL의 용액)은 총 10mL를 포함하고 있기 때문에, 이러한 감소에 대한 희석계수가 10배이거나 10배 의 일련 희석(그림1)이라고결론을 내릴 수 있다. 우리는 이 예에서 100개의 세포로 만 시작하고 10의 요인에 의해 희석하고 있기 때문에, 단지 2단계는 1 세포의 절대 최소 농도에 도달하기 위하여 요구됩니다.
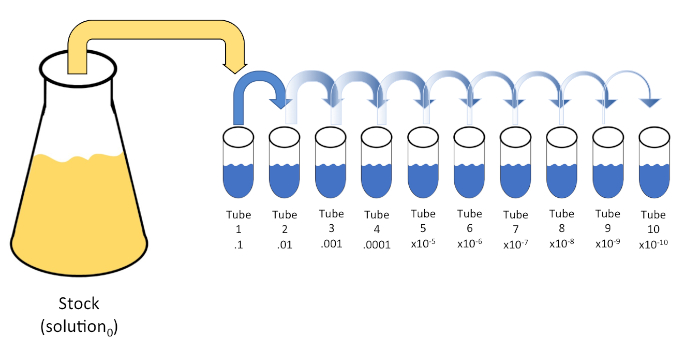
그림 1: 주식 솔루션의 직렬 희석. 스톡 용액의 1mL 알리쿼트(솔루션0)는0.45% 식염수(dilent1)의9mL를 포함하는 튜브 1에 첨가된다. 이 혼합물의 제품은 용액1입니다. 새로 만든 솔루션1의 1mL를 알리쿼팅하고 튜브 2에 추가하여 반복합니다. 알리쿼팅 및 재서스펜션은 최종 튜브에 도달할 때까지 이러한 방식으로 계속되며, 각 단계마다 10배씩 재고 농도를 희석시합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
직렬 희석은 원하는 유기체의 관리 가능한 농도를 얻기위한 가장 간단한 기술이며, 미생물학자가 사용하는 많은 도금 기술의 두 개, 페트리 접시 줄무늬 및 확산에 의해 보완된다. 이 접근법의 이점은 실험자가 단일 종의 순수한 균주를 수확하거나 혼합 된 인구 (7)로부터 균주를 분리 할 수 있다는 것입니다. 줄무늬는 적절한 영양소를 사용할 수 있는 경우 성장하게 될 고체 배지(일반적으로 아가로즈로 구성된)에 유기체를 도입함으로써 달성된다. 단단한 부비동성 패턴에서 멸균 접종 루프를 매체를 가로질러 부드럽게 쓸어 넘기면 실험자의 파형의 주파수에 비례하여 유기체를 분배합니다. 페트리 접시를 3/4(사분면 줄무늬)로 나누고, 접시의 새로운 영역이 입력됨에 따라 각 줄무늬의 빈도를 줄이면 점차 해당 지역을 점유할 수 있는 미생물의 수가 줄어들어 정량화할 수 없는 세균 잔디 대신 단일 식민지를 생성합니다. 스프레드 도금은 시료를 추가로 희석시키지 않습니다. 멸균 유리 스프레더는 전체 페트리접시(그림 2)에서스펜션 미디어의 알리쿼트를 분배하는 데 사용됩니다. 배기판상에서 자라는 콜로니는 단일 세포에서 발생하며 각 식민지는 주어진 현탁액에서 밀리리터당 콜로니 형성 유닛(CFU)의 수를 추정할 수 있으며, CFU/mL(6)(그림3) (그림 3)소프트 한천 및 복제도금은 전술한 기술의 변형이며 박터피오파지 및 돌연변이제(7)의 분리를 허용한다.
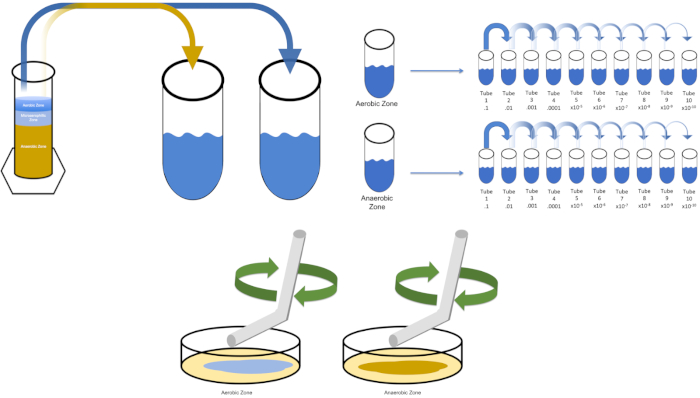
그림 2: 세균 열거 및 균주 격리를 위한 판 줄무늬. 페트리 접시의 바닥에 식별 정보(박테리아 이름, 날짜, 미디어)로 레이블을 지정하고 3분의 1로 나눕니다. 스톡 샘플의 적절한 희석을 선택한 후 멸균(일회용 또는 화염) 접종 루프를 취하고 테스트 튜브(here, T3)를 침수합니다. 한쪽에 페트리 접시 커버를 약간 올려 서 접종 루프만 한천에 접근할 수 있습니다. 지그재그 방식으로 미디어 상단을 가로질러 접종루프를 미끄러지듯 미끄러지며 한천을 손상시키지 않도록 주의하세요. 플레이트를 약 1/3rd(~118°)로 회전시키고 지그재그 동작의 빈도를 줄입니다. 마지막 시간을 회전하고 지그재그 주파수를 한 번 더 줄입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
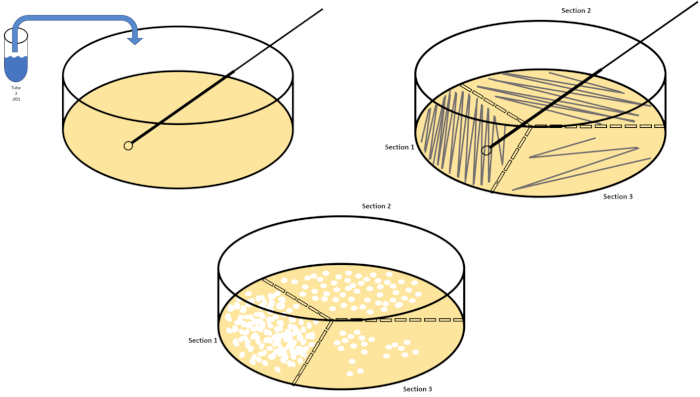
그림 3: 확산 도금. 에어로빅 영역의 1 g는 T1에서 다시 중단된 다음 직렬로 희석되었습니다. 멸균 유리 또는 플라스틱 일회용 확산 막대는 각 접시에 걸쳐 접종을 분배하는 데 사용됩니다. 이것은 혐기성 영역의 1 g로 반복되었다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
직렬 희석과 마찬가지로, 로그와리믹 스케일은 유기체 농도를 표현하기 위해 사용된다. 100mm x15mm를 측정하는 표준 페트리 접시에서 재배되는 콜로니의 수는 분리된 성장 클러스터를 식별하여 수동으로 열거(또는 전산 처리의 도움으로 자동화)할 수 있습니다. 30개 미만이거나 300보다 작은 합계는 계산하기에는 너무 적거나 계산할 수 없는 수(TNTC)로 정의되어야 합니다. 후자의 경우, 새로운 페트리 접시를 다시 휘감기 전에 농도를 줄이기 위해 직렬 희석을 수행해야합니다. 세 개의 분리된 페트리 접시에서 확인된 자급자족 식민지의 수를 평균화하고 희석 계수에 의해 평균을 곱하면 CFU/mL이 생성됩니다. 시간에 대한 CFU/mL의 로그10을 플로팅하면 유기체의 평균 생성 시간이 드러나게 된다(7).
Procedure
Applications and Summary
Bacterial enumeration and strain isolation by plating requires manageable concentrations of target organisms. Successful plating is therefore contingent upon serial dilution. As such, the aforementioned techniques remain the cornerstone of microbiological examination and experimentation. Though simple by design, dilution factors and plating technique can be modified to by the experimenter to bolster outcomes without compromising the integrity of each method. Plotting the four phases of bacterial growth can be helpful when characterizing desired microbes. These phases, lag, log, stationary, and death, are marked by changes in bacterial replication. The lag phase features slow growth due to physiological adaptation, the log phase is the period of maximum proliferation featuring an exponential rise in viable cells, stationary phase is then reached due to environmental limitations and accumulations of toxins, before the death phase where cell counts begin to fall. This can be accomplished by serially diluting (or 1-step diluting to avoid confusion) Solution0 every hour for a total of 8 hours, beginning at Time0 (Solution0 should be returned to a shaking incubator after each dilution). Calculate the log10 of CFU/ml for a single diluent of Time0 and plot on the Y-axis. Repeat this calculation for the sample Time1 (make sure calculate CFU/mL using the same dilution factor as Time0). Repeat until each time (Time1-Time8) are plotted on the X-axis.
References
- Allen, M.E., Gyure, R.A. (2013) An Undergraduate Laboratory Activity Demonstrating Bacteriophage Specificity. Journal of Microbial Biological Education 14: 84-92.
- Ben-David, A., Davidson, C.E. (2014) Estimation Method for Serial Dilution Experiments. Journal of Microbiological Methods 107:214-221.
- Goldman, E., Green, L.H. (2008) Practical Handbook of Microbiology.
- Koch, R. (1883) New Research Methods for Detection of Microcosms in Soil, Air and Water.
- Lederberg, J., Lederberg, E.M. (1952) Replica Plating and Indirect Selection of Bacterial Mutants. Journal of Bacteriology 63:399-406
- Pepper, I., Gerba, C., Ikner, L. (2019) Bacterial Growth Curve Analysis and its Environmental Changes. JoVE Science Education Database. Environmental Microbiology.
- Sanders., E.R. (2012) Aseptic Laboratory Technique: Plating Methods. JoVE 63:e3063.
Transcript
Sometimes, in order to identify and study bacteria we first need to isolate and enrich them from a sample. For example, samples obtained from a Winogradsky Column are mixed, meaning they contain multiple species or strains of bacteria, so studying an individual bacterium or enumerating the different kinds present can be challenging. To this end, serial dilution and plating techniques are typically employed to reliably quantify bacterial load and isolate individual colonies.
Serial dilution is a process through which the concentration of an organism, bacteria in this example, is systematically reduced through successive resuspension in fixed volumes of liquid diluent. Usually the volume of the diluent is a multiple of 10 to facilitate logarithmic reduction of the sample organism. For example, one gram of sediment is first removed from the Winogradsky zone of interest and added to 10 milliliters of an appropriate liquid medium. Then, one milliliter of this first dilution is added to another tube containing nine milliliters of medium. The process can be repeated until several different concentrations of bacteria have been prepared. Serial dilution is the key to enumeration of bacteria in this example, since mixed samples from a Winogradsky Column contain an unknown, often large, number of bacteria.
Next, streak plating and spread plating enable the isolation and enumeration of bacteria within a sample, respectively. Streaking is accomplished by introducing a diluted sample to one section of the solid medium supplemented with nutrient, which is divided into thirds. This inoculum is then spread over each third of the plate in a zig-zag pattern. As different sections of the plate are streaked, crossing from the previous sample only once, the sample is spread more thinly. This means that you may only need to streak from one dilution to achieve individual colonies in the later sections. After incubation, the streaked plates allow for observations of colony morphology, information that can help differentiate between different bacterial species.
Alternatively, if the main goal is the enumeration of the bacteria in the sample spread plating may be used. In spread plating, an aliquot of a single sample is spread evenly over the entire surface of solid medium. Typically, because we don’t know the bacterial numbers in the mixed sample, a spread plate is made for each of the dilutions or a representative sample of them. After incubation, enumeration can be performed using these spread plates. Any plates with colony counts fewer than 30 should be discarded, since small counts are subject to greater error. Similarly, any counts over 300 should be discarded because colony crowding and overlapping can lead to underestimation of colony count. If the colony counts of each of these remaining dishes is recorded and multiplied by the dilution factor, and then divided by the volume plated, this yields the colony forming units, or CFUs, per milliliter of suspension. In this video, you will learn how to qualitatively and quantitatively evaluate a sample containing a known bacterium, and the microbial communities contained in various regions of a Winogradsky Column via serial dilution, spread plating, and streak plating.
First, put on any appropriate personal protective equipment including a lab coat, gloves, and goggles. Next, sterilize the workspace with 70% ethanol and wipe down the surface. Next, gather two 500 milliliter Erlenmeyer flasks and label one broth and the other agar. To prepare LB agar solution, mix approximately 6.25 grams of LB agar, three grams of technical agar, and 250 milliliters of distilled water in the flask labeled agar.
Then, prepare LB broth by combining 2. 5 grams of LB media and 100 milliliters of distilled water in the flask labeled broth. After autoclaving the flasks, use a heat resistant glove to remove the flasks from the autoclave and place them in a 40 to 50 degree Celsius water bath. Once the flasks are 50 degrees Celsius, carefully prepare three 100 milliliter aliquots of the broth solution and label each aliquot solution zero. Next, gather 10 sterile petri dishes and label them with the date, name, type of media used, and the Winogradsky Column zone that the organisms will be harvested from. Pipette 15 milliliters of agar from the agar flask into each petri dish. Then, use the pipette tip to remove any bubbles, replace the plate lids, and allow them to solidify on the bench top overnight.
The next day, wipe down the bench top with 70% ethanol. Next, label 10 20 milliliter test tubes T1 through T10 and place them in a rack. Pipette nine milliliters of .45% saline into each tube. Now, cover each of the 10 test tubes loosely with their caps and transfer them to an autoclave-compatible test tube rack. After the cycle is complete, remove the saline blanks using heat resistant gloves and allow them to cool. Store the tubes at room temperature until they have reached approximately 22 degrees Celsius.
To cultivate a known target organism, E. coli in this example, inoculate 100 milliliters of solution zero with a single colony from a previously streaked plate. Then, cover the tube and incubate it over night at 37 degrees Celsius. To evaluate the regions of a Winogradsky Column, add approximately one gram of material from the aerobic zone to T1 and resuspend by vortexing. Then, repeat this process with one gram of material from the anaerobic zone.
Remove the tube containing solution zero inoculated with E. coli from the incubator and shake it. Then, pipette one milliliter of the solution into a T1 test tube and vortex to mix. Remove one milliliter of solution from T1 and transfer it to T2, vortexing to mix. Repeat this process through tube T10. To evaluate the aerobic and anaerobic zones of the Winogradsky Column, remove one milliliter of solution from each of the previously prepared T1 tubes and transfer it to the appropriate T2 tubes. Then, continue the serial dilutions through the T10 tubes as previously demonstrated.
To spread plate, pipette 100 microliters of the diluted sample from each T3 tube on to the corresponding petri dish. Then, use a sterile spreading rod to gently distribute the sample on to the petri dish and replace the plate lid. Repeat this process for the T6 and T9 dilutions, as previously demonstrated. Incubate the plates containing aerobic organisms in a 37 degree Celsius incubator for 24 hours. Incubate the plates containing anaerobic organisms in an anaerobic chamber set to 37 degrees Celsius for 24 hours. The next day remove the T3, T6, and T9 dilution plates from the incubator and the anaerobic chamber and transfer them to the bench top. Working with one plate at a time, glide a sterile inoculating loop across the top of the media in a zig-zag pattern. Then, replace the petri dish lid. Next, rotate the plate by 1/3 and sterilize the loop to reduce the frequency of the previously made zig-zag pattern. Again, after sterilizing the loop, rotate the plate by 1/3, reduce the frequency of the zig-zag pattern one last time, and replace the lid. Repeat this streaking method for the remaining plates, as previously shown. Then, place the streaked plates containing aerobic organisms in a 37 degree Celsius incubator overnight and the streaked plates containing anaerobic organisms in an anaerobic chamber set to 37 degrees Celsius overnight.
Cultures were harvested from the aerobic and anaerobic zones of a seven day Winogradsky Column. Then, the cultures were serially diluted prior to streaking and spreading on LB agar plates. Streaking revealed a mixed population from each of the evaluated Winogradsky zones, and the spread plates produced similar results. A plate streaked from a mixed population will result in bacterial colonies of different shapes, sizes, textures, and colors. In contrast, the streaked and spread plates containing the known organism, E. coli, demonstrated a homologous population. Generally, it is best to calculate CFUs per milliliter using the average colony count of three plates spread with the same sample and dilution factor. Multiply the average number of colonies by the dilution factor and divide by the amount aliquoted. Finally, isolated colonies chosen from each plate can be used in further enrichment assays to determine species identity.
