ديناميات القلة من مستقبلات سطح الخلية في الخلايا الحية من قبل المجهر الداخلي الكلي انعكاس الشامل مع عدد وتحليل السطوع
Summary
ونحن وصف نهج التصوير لتحديد الدولة المعتدلة القلة من الأقليات megfp-الموسومة-مستقبلات التي يسببها يجند ملزمه في غشاء البلازما من الخلايا الحية. يعتمد البروتوكول علي المجهر الداخلي للانعكاس الكلي (TIRF) المدمج مع تحليل العدد والسطوع (N & B).
Abstract
علي الرغم من اهميه وانتشار القلة المستقبلة ، فان بعض الطرق قابله للتطبيق للكشف عن احداث التجميع وقياس درجه التجميع. هنا ، ونحن وصف نهج التصوير لتحديد الدولة المعتدلة القلة من mEGFP-الموسومة-مستقبلات المتجانسات في غشاء الخلايا الحية. يعتمد البروتوكول علي المجهر الداخلي للانعكاس الكلي (TIRF) المدمج مع تحليل العدد والسطوع (N & B). N & B هو أسلوب مشابه للتحليل الطيفي للعلاقة الفلورية (FCS) والرسم البياني للعد الفوتون (PCH) ، والتي تستند إلى التحليل الإحصائي للتقلبات في كثافة الفلورية من الفلورية التي تنشر داخل وخارج الاضاءه حجم خلال فتره المراقبة. علي وجه الخصوص ، N & B هو تبسيط PCH للحصول علي معلومات عن متوسط عدد البروتينات في مخاليط القلة. وتوصف شده تذبذب الكثافة بالسطوع الجزيئي للفلوكوفيري ومتوسط عدد الفلوبوهورس داخل حجم الاضاءه. التالي ، لا يعتبر N & B سوي اللحظتين الاولي والثانية من توزيع السعه ، وهما متوسط الكثافة والتباين. هذا هو ، في الوقت نفسه ، وقوه وضعف الأسلوب. لأنه يتم النظر في لحظات فقط ، N & B لا يمكن تحديد الجزء المولي من القلة غير المعروفة في خليط ، ولكنه فقط يقدر متوسط حاله قله العدد من الخليط. ومع ذلك ، فانه يمكن تطبيقها علي سلسله زمنيه صغيره نسبيا (بالمقارنة مع أساليب لحظه أخرى) من الصور من الخلايا الحية علي أساس بكسل بكسل ، وذلك ببساطه عن طريق رصد تقلبات الوقت من كثافة مضان. فانه يقلل من الوقت الفعلي لكل بكسل إلى بضعة ميكروثانية ، مما يسمح الاستحواذ في النطاق الزمني من ثوان إلى مللي ثانيه ، وهو أمر ضروري لحركيه القلة السريعة. وأخيرا ، يمكن استكشاف مناطق الخلايا الكبيرة وكذلك المقصورات شبه الخلوية.
Introduction
ونحن وصف إجمالي انعكاس الداخلية الفلورية-عدد والسطوع (TIRF-N & B) نهج التصوير لتحديد متوسط الدولة القلة من جزيئات مستقبلات في غشاء البلازما من الخلايا الحية ، وتهدف إلى ربط التجمع مستقبلات ديناميات الوظيفة البيولوجية للبروتينات (الشكل 1).
عند الربط خارج الخلية وملزمه ، والمستقبلات بدء محول الاشاره داخل الخلايا اعتمادا علي التشكيل الخاصة بهم ، والقلة ، والمستقبلات المشتركة المحتملة وتكوين الغشاء. علي الرغم من اهميه وانتشار القلة المستقبلة ، المعترف بها كحدث رئيسي في الإشارات الخلوية1،2،3،4،5،6، 7, طرق قليله يمكن الكشف عن تجمع الاحداث وقياس درجه التجميع تجريبيا8,9. وحده التخزين البؤري (x ، y ≈ 300 nm ، z ≈ 900 nm) غير محلول بشكل كاف لإثبات التفاعل الجزيئي والقياس ، حتى بعد التحسين بواسطة خوارزميات استعاده الصورة10. لا يمكن حل تكوين الوحدة الفرعية من قله البروتين علي أساس المكانية بحته حتى من خلال أساليب فائقه الدقة في x, y القرار من 20-70 nm مثل النخيل11, العاصفة12, وتيد13. وعلاوة علي ذلك ، فان الدقة الزمنيه (بترتيب الدقائق لكل صوره) لا يمكن ان تتبع الحركية في نطاق الثواني. جزيء واحد خطوه التبييض علي حل القياس من قله البروتين فقط إذا كانت غير المتنقلة14.
واحده من الطرق الأكثر تنوعا لقياس الكثافة وقله الفلوريسسينتلي من البروتينات الموسومة داخل صور واحده هو تحليل توزيع كثافة المكانية (SpIDA) ، والتي تعتمد علي أخذ العينات المكانية. وهو ينطبق علي كل من الخلايا الثابتة كيميائيا والحية ، ويسمح تحليل العديد من المناطق التي تهم الخلية في وقت واحد باستخدام المجهر القياسية الفلورية15. بدلا من ذلك ، وأساليب لحظه ، مثل الطيفية الارتباط فلوري (FCS)16، المدرج التكراري العد الفوتون (pch)17، وعدد والسطوع (N & B) 18،19، هي مناسبه لقلهالسنالكمية القياسات. وتحلل هذه الطرق تقلبات الكثافة الفلورية التي يمكن ملاحظتها في الوقت الذي ينتشر فيه الفلوريد ويخرج من حجم الاضاءه. الاتساع من التقلبات شده يستطيع كنت بشكل فريد وصفت بالسطوع جزيئيه من ال [فلوفيبور] (ε) والرقم معدله [فلوبفرس] (ن) ضمن الاناره حجم17 (شكل 2). عاده ، يمكن الحصول علي معامل انتشار الفلوروبيورس ومتوسط عدد الجزيئات (المرتبطة عكسيا بقيمه G (0)) داخل حجم الاضاءه بواسطة FCS20. ومع ذلك ، منذ نشر الوقت فقط الجداول مع جذر مكعب من كتله ، FCS ليست حساسة بما فيه الكفاية للكشف عن التغيرات في الكتلة الجزيئية21. في الممارسة العملية ، لا يمكن الكشف عن اللون واحد FCS ديميريشن من مستقبلات الغشاء. PCH يحل مخاليط من قله المواليد المختلفة بدقه. باستخدام أكثر من لحظتين من توزيع السعه ، يكتشف جزيئات السطوع المختلفة التي تشغل نفس حجم الاضاءه. المسح الضوئي FCS22 والتطورات ، مثل الزوج مثيره للاهتمام-ارتباط السطوع الجزيئي (pcomb) النهج23، وعرضت لتوسيع نطاق تطبيق أساليب الارتباط فلوري في النظم البيولوجية24 ، تبقي الطرق نقطه واحده تفتقر إلى القدرة علي القياسات السريعة في مساحة كبيره من الخلية ، والتي تتطلب العديد من الملاحظات المتتابعة في كل بكسل والحصول علي البيانات في ترتيب الثواني.
N & B هو نسخه مبسطه من pch التي تعتبر فقط اللحظات الاولي والثانية من سعة التوزيع الفلوري ، وهي متوسط الكثافة ، ، والفرق ، σ2 (الشكل 2)18،19 وبسبب ذلك ، فانه لا يمكن تحديد الجزء المولي من القلة غير المعروفة في خليط ، ولكن فقط تقديرات الدولة المعتدلة القلة من الخليط. ومع ذلك ، N & B لديه ميزه العمل مع سلسله زمنيه أصغر نسبيا من الصور من الخلايا الحية من PCH علي أساس بكسل بكسل ، وذلك ببساطه عن طريق رصد التقلبات في الوقت المناسب من كثافة مضان. لان N & B يقلل من الوقت لكل بكسل إلى بضع ميكروثانية ، فانه يمكن ان تتبع حركيه القلة السريعة علي مناطق الخلايا الكبيرة ، مما يسمح اكتساب الصورة علي مقياس الوقت من الثواني في المسح الضوئي المجهر (علي سبيل المثال ، التنسيق ، 2-الفوتون) ومللي ثانيه في المجهر القائم علي الكاميرا (علي سبيل المثال ، TIRFM).
وقد أظهرت عده تقارير قدره N & B علي قياس عدد الوحدات الفرعية في مجموعات البروتين عن طريق تصوير مناطق الخلايا الموسعة. تم الكشف عن مجموعات باكيلين-EGFP في مواقع التصاق في الخلايا تشو-K125، وتم وصف التجميع داخل الخلايا من الببتيد Httex1p المسببة للامراض في الخلايا كوس-726. وقد تم تطبيق N & B لاتباع القلة التي يحركها الأمر من مستقبلات erbb27، وتاثير يجند علي كلوثوب (klb) و FGFR1c في الخلايا هيلا28. تم استخدام الجمع بين التصوير TIRF وتحليل N & B لإظهار ان دينامين-2 هو رباعيه في المقام الأول في جميع انحاء الغشاء الخلوي بأكمله29. قمنا بتطبيق N & B لكل من المسح النقطي والصور tirf لإثبات ديميريشن الموجهة من upar ومستقبلات غشاء الخلية FGFR130,31.
طرق الارتباط الفلورية ، مثل N & B ، FCS و PCH ، وتستند إلى فكره انه في حجم مفتوح الاحتلال عدد من الجزيئات يتبع توزيع Poisson. لأنه فقط الفوتونات التي تنبعث منها الفلوروبيورس يمكن الكشف عنها ، والقيمة المتوسطة لكثافة مضان قياس  مقابل الوقت في بكسل من الصورة ، ، هو نتاج متوسط عدد الفلوبورس في حجم الاضاءه ، ن ، وبهم السطوع الجزيئي ، ε17:
مقابل الوقت في بكسل من الصورة ، ، هو نتاج متوسط عدد الفلوبورس في حجم الاضاءه ، ن ، وبهم السطوع الجزيئي ، ε17:

حيث يتم التعبير عن ε كعدد من الفوتونات المنبعثة لكل وحده من الوقت (تقليديا في الثانية) لكل جزيء عندما يكون الجزيء في مركز حجم الاضاءه.
السطوع هو خاصيه لكل فلوكوفيري في عمليه استحواذ معينه ، في حين ان الكثافة هي مجموع جميع المساهمات من جميع الفلوبورس. في المسابقات البيولوجية ، سيزداد السطوع مع زيادة عدد الفلوريد الذي يتقلب معا ، مما يعطي معلومات عن حاله قله الفلوريسسينتلي من البروتين الموسوم. يتم قياس سعة التذبذب في بكسل معينه من التباين في اشاره مضان ، σ2:

حيث المتوسطة من المربع الشدة  ,, والمربع من المدى الشدة
,, والمربع من المدى الشدة  ,, يحسب من الفردية كثافة قيم في كل [بكسل] من كل اطار:
,, يحسب من الفردية كثافة قيم في كل [بكسل] من كل اطار:
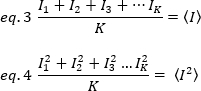
حيث K هو عدد الإطارات الاجماليه في سلسله الوقت. من الناحية التجريبية ، فمن الضروري حساب لسلسله الصورة بأكملها الفرق الذي يصف مبعثر قيم كثافة الفردية في كل بكسل من صوره واحده حول قيمه كثافة متوسط. ويشمل الفرق جميع التقلبات من أصول مختلفه. في تقريب اولي, التباين واجبه ال ينتشر جسيمات في الاناره حجم, σ20, يستطيع كنت فصلت من التباين واجبه إلى الكاشف طلقه خردق ضوضاء, σ2[د.]. والفرق بينهما مستقل ؛ التالي ، يتم إعطاء الفرق الإجمالي من خلال المبلغ الخاص بهم:

الفرق ، بسبب التقلبات الجزيئية داخل وخارج حجم الكشف ، يعتمد خطيا علي السطوع الجزيئي وكثافة:

أعاده ترتيب المكافئ 6 وفقا لمكافئ 1:

وفقا للمفهوم النموذجي في الطيفي الترابط الفلوري ، المعادلة 7 تنص علي ان الفرق بسبب عدد من التقلبات يعتمد علي مربع من سطوع الجسيمات.
ثم, الفرق بسبب تقلبات كاشف هو وظيفة خطيه من كثافة الكشف, تحت افتراض ان يتم تشغيل كاشف تحت الحد التشبع19:

في حاله الفوتون العد كاشفات a= 1 و c= 0 ، التالي فان الفرق كاشف يساوي متوسط كثافة:

لتطبيق هذه المفاهيم علي القياسات الحقيقية في الخلايا الحية ، Gratton وزملاءه18 تحديد سطوع واضح ، B ، لكل بكسل كنسبه الفرق علي مدي كثافة متوسط:

B هي المعلمة التي تقاس بالتجربة. في هذا العمل ، يتم التقاط صور السلسلة الزمنيه لمستقبلات FGFR1 في غشاء البلازما من الخلايا هيلا من قبل المجهر TIRF ومتوسط سطوع واضح ، B ، يتم تحديده من قبل تحليل N & B. بعد ذلك ، بعد أضافه FGF2 ، يتم التقاط سلسله زمنيه متتالية لمتابعه التغييرات في التجميع الذاتي لجزيئات المستقبلات في سطح الغشاء بعد تحفيز المستقبلات مع ligand المتعارف عليه.
ومع ذلك ، منذ كاشف المجهر TIRF هو كاميرا EMCCD ، والتعبير عن سطوع واضح يحتاج إلى تعديل كما19:

حيث أزاحه هو أزاحه كثافة الكترونيات الكشف التي هي سمه من سمات إعدادات كاشف. يتم إعطاء الفرق ومتوسط كثافة للكشف التناظرية علي التوالي من قبل:
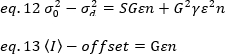
حيث G هو كسب التناظرية في المستويات الرقمية (DL/الفوتونات) ، S، والمستويات الرقمية لكل فوتون19، وتعطي من قبل منحدر كثافة مقابل مؤامرة الفرق لمصدر الضوء مع كثافة ثابته (اي تقلبات الزمنيه). يرتبط عامل γ بشكل وحده تخزين الكشف عن البكسل. وفقا ل Hassler وآخرون32، عامل γ يساوي 0.3 للتصوير tirf العمل في الكسب الأقصى من الكاميرا الكشف19. الازاحه ، S و G المعلمات هي خصائص الكاميرا والمجهر. السطوع الظاهر ، B ، يتم الحصول عليه عن طريق أعاده ترتيب المكافئ 11 وفقا لمكافئ 12 و 13:

تجريبيا ، ε هو وظيفة معقده من كثافة الليزر وكفاءه الكشف عن النظام. ومع ذلك ، بما ان B/S يعتمد خطيا علي ε ، فمن المهم تحديد القيمة النسبية لε لوضع الكشف المعطي:

حيث ε ‘ يتناسب مع الε. ومع ذلك ، يتم اجراء المعايرة باستخدام مرجع داخلي.
Protocol
Representative Results
Discussion
N & B يتطلب العديد من الاحتياطات في اختيار نموذج الخلية واستراتيجية وضع العلامات. ويمكن تطبيقه فقط علي الخلايا الحية التي لا تزال ملتزمه بثبات خلال وقت التقاط الصورة. ويمكن التعامل مع التقلبات الاضافيه الناجمة عن النزوح الجامد للخلية بأكملها مع نهج استعاده الصورة المناسبة38. و?…
Offenlegungen
The authors have nothing to disclose.
Acknowledgements
ويدعم هذا المركز وزاره الاستخبارات والابتكار واليقع والمؤسسة المناصرة للمؤسسة الخيرية ، وهو أحد مراكز الامتياز في سيفيرو اوتشوا (سيف-2015-0505). ويدعمنا أيضا الصندوق الأوروبي للتنمية الاقليميه “Una manera de هاجر Europa”. وتنوه جامعه كاليفورنيا بالدعم المقدم من الرابطة الايطاليه للبحث عن السرطان ، وجمعيه البحوث الدولية للسرطان (المعروفة الآن باسم بحوث السرطان في جميع انحاء العالم) ، ووزارة الصحة الايطاليه. وتقر تحكيم بان “مؤسسه بيانكا ديل مؤنتي دي لومباردي” لدعمها الجزئي لعمله مع الزمالة الكهروضوئية “Progetetetalita ايفانو Becchi” 2011-2012.
Materials
| 3-Colour Fast TIRF Leica AM TIRF MC inverted microscope, with smi-automatic TIRF alignment. The microscope is equipped with a diode 488 nm laser, a 100×1.46 oil TIRF objective, Ex/Em Bandpass filters at 490/20 and 525/50, temperature/CO2 incubator and Andor DU 8285 VP EMCCD camera. The microscope is operated by Leica LIF software. | Leica Microsystems, Wetzlar, Germany | ||
| Albumin from Bovine Serum 98% minimun | Sigma-Aldrich, St. Louis, MI, USA | A7906-100G | |
| DMEM without Phenol Red with 25 mM HEPES | GIBCO Thermo Fisher Scientific,Waltham, MA, USA | 21063029 | Used serum free for microscopy |
| DMEM high-glucose GlutaMAX I | GIBCO Thermo Fisher Scientific,Waltham, MA, USA | 10566-016 | Used for complete medium |
| Dulbecco's Phosphate Buffered Saline 10x (PBS) | Biowest, Nuaillé, France | X0515-500 | |
| Emission splitting system Photometrics DV2 | TeledynePhotometrics, Tucson, AZ, USA | ||
| Fetal Bovine Serum, qualified, Brazil | GIBCO Thermo Fisher Scientific,Waltham, MA, USA | 10270106 | 10% inactivated supplement for complete medium |
| Glass bottom 35-mm sterile 1.5 dishes | MatTek, Ashland, MA, USA | P35G-0.170-14-C | uncoated, glass thickness 0.17 microns |
| GraphPad Prism | GraphPad Software Inc., San Diego, CA, USA | ||
| Human cervical carcinoma (HeLa), serum-free animal component (AC) cells | Millipore-Sigma ECACC, Darmstadt, Germany | CB_08011102 | |
| iXonEM+ 897 EMCCD (back-illuminated) ANDOR camera controlled by ANDOR Solis software | Oxford Instruments, Andor TM Technology, Abingdon-on-Thames, UK | This camera, installed in an additional port of the microscope, is used for acquiring the N&B time series | |
| Matlab Executable N&B routine | Unit of Microscopy and Dynamic Imaging, CNIC, Madrid, Spain | download at https://www.cnic.es/en/investigacion/2/1187/tecnologia | |
| MatLab v.2018b | The MathWorks, Inc. Natick, MA, USA | download at https://www.mathworks.com/products/matlab.html | |
| Penicillin:Streptomycin for tissue culture 100x | Biowhittaker Inc. Walkersville, MD, USA | LONZA 17-602E | supplement for medium at Penicillin/Streptomycin 100U/100µg. |
| pN1-mEGFP-FGFR1 expression vector | Unit of Gynecological Oncology Research, European Institute of Oncology IRCCS, Milan, Italy | Zamai et al., 2019 | |
| pN1-N-Gly-mEGFP-GPI expression vector | Unit of Microscopy and Dynamic Imaging, CNIC, Madrid, Spain | Hellriegel et al., 2011 | |
| pN1-N-Gly-mEGFP-mEGFP-GPI expression vector | Unit of Microscopy and Dynamic Imaging, CNIC, Madrid, Spain | Hellriegel et al., 2011 | |
| Recombinant FGF2 | PeproTech EC, Ltd., London, UK | Ligand solution: 20ng/mL of FGF2 in PBS supplemented with 0.01%BSA. | |
| Sodium pyruvate GIBCO | ThermoFisher Scientific | 11360070 | 1mM supplement for medium |
| TransIt-LT1 Transfection Reagent | MirusBio LLC, Madison, WI, USA | MIR 2300 | |
| Trypsin-EDTA (0.25%), phenol red | GIBCO Thermo Fisher Scientific,Waltham, MA, USA | 25200056 | |
| Type F Immersion liquid 10 mL | Leica Microsystems, Wetzlar, Germany | 11513 859 | |
| UltraPure BSA (50 mg/mL) | ThermoFisher Scientific | AM2618 | 0.1% supplement for medium without phenol red used for transfections |
Referenzen
- Agwuegbo, U. C., Jonas, K. C. Molecular and functional insights into gonadotropin hormone receptor dimerization and oligomerization. Minerva Ginecologica. 70 (5), 539-548 (2018).
- Ferre, S., et al. G protein-coupled receptor oligomerization revisited: functional and pharmacological perspectives. Pharmacological Reviews. 66 (2), 413-434 (2014).
- Marsango, S., Ward, R. J., Alvarez-Curto, E., Milligan, G. Muscarinic receptor oligomerization. Neuropharmacology. 136 (Pt C), 401-410 (2018).
- Oishi, A., Cecon, E., Jockers, R. Melatonin Receptor Signaling: Impact of Receptor Oligomerization on Receptor Function. International Review of Cell and Molecular Biology. 338, 59-77 (2018).
- Thelen, M., Munoz, L. M., Rodriguez-Frade, J. M., Mellado, M. Chemokine receptor oligomerization: functional considerations. Current Opinion in Pharmacology. 10 (1), 38-43 (2010).
- Van Craenenbroeck, K. GPCR oligomerization: contribution to receptor biogenesis. Subcellular Biochemistry. 63, 43-65 (2012).
- Wnorowski, A., Jozwiak, K. Homo- and hetero-oligomerization of beta2-adrenergic receptor in receptor trafficking, signaling pathways and receptor pharmacology. Cell Signaling Technology. 26 (10), 2259-2265 (2014).
- Fricke, F., Dietz, M. S., Heilemann, M. Single-molecule methods to study membrane receptor oligomerization. Chemphyschem. 16 (4), 713-721 (2015).
- Vidi, P. A., Ejendal, K. F., Przybyla, J. A., Watts, V. J. Fluorescent protein complementation assays: new tools to study G protein-coupled receptor oligomerization and GPCR-mediated signaling. Molecular and Cellular Endocrinology. 331 (2), 185-193 (2011).
- Trussell, H. J., et al., Trussell, J., et al. . Academic Press Library in Signal Processing. 4, 3-9 (2014).
- Betzig, E., et al. Imaging intracellular fluorescent proteins at nanometer resolution. Science. 313 (5793), 1642-1645 (2006).
- Rust, M. J., Bates, M., Zhuang, X. Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy (STORM). Nature Methods. 3 (10), 793-795 (2006).
- Nagerl, U. V., Willig, K. I., Hein, B., Hell, S. W., Bonhoeffer, T. Live-cell imaging of dendritic spines by STED microscopy. Proceedings of the National Academy of Sciences of the United States of America. 105 (48), 18982-18987 (2008).
- Tsekouras, K., Custer, T. C., Jashnsaz, H., Walter, N. G., Presse, S. A novel method to accurately locate and count large numbers of steps by photobleaching. Molecular Biology of the Cell. 27 (22), 3601-3615 (2016).
- Godin, A. G., et al. Revealing protein oligomerization and densities in situ using spatial intensity distribution analysis. Proceedings of the National Academy of Sciences of the United States of America. 108 (17), 7010-7015 (2011).
- Qian, H., Elson, E. L. Distribution of molecular aggregation by analysis of fluctuation moments. Proceedings of the National Academy of Sciences of the United States of America. 87 (14), 5479-5483 (1990).
- Chen, Y., Muller, J. D., So, P. T., Gratton, E. The photon counting histogram in fluorescence fluctuation spectroscopy. Biophysical Journal. 77 (1), 553-567 (1999).
- Dalal, R. B., Digman, M. A., Horwitz, A. F., Vetri, V., Gratton, E. Determination of particle number and brightness using a laser scanning confocal microscope operating in the analog mode. Microscopy Research and Technique. 71 (1), 69-81 (2008).
- Unruh, J. R., Gratton, E. Analysis of molecular concentration and brightness from fluorescence fluctuation data with an electron multiplied CCD camera. Biophysical Journal. 95 (11), 5385-5398 (2008).
- Hess, S. T., Huang, S., Heikal, A. A., Webb, W. W. Biological and chemical applications of fluorescence correlation spectroscopy: a review. Biochemie. 41 (3), 697-705 (2002).
- Muller, J. D., Chen, Y., Gratton, E. Fluorescence correlation spectroscopy. Methods in Enzymology. 361, 69-92 (2003).
- Levi, V., Ruan, Q., Kis-Petikova, K., Gratton, E. Scanning FCS, a novel method for three-dimensional particle tracking. Biochemical Society Transactions. 31 (Pt 5), 997-1000 (2003).
- Hinde, E., et al. Quantifying the dynamics of the oligomeric transcription factor STAT3 by pair correlation of molecular brightness. Nature Communications. 7, 11047 (2016).
- Waithe, D., et al. Optimized processing and analysis of conventional confocal microscopy generated scanning FCS data. Methods. 140-141, 62-73 (2018).
- Digman, M. A., Dalal, R., Horwitz, A. F., Gratton, E. Mapping the number of molecules and brightness in the laser scanning microscope. Biophysical Journal. 94 (6), 2320-2332 (2008).
- Ossato, G., et al. A two-step path to inclusion formation of huntingtin peptides revealed by number and brightness analysis. Biophysical Journal. 98 (12), 3078-3085 (2010).
- Nagy, P., Claus, J., Jovin, T. M., Arndt-Jovin, D. J. Distribution of resting and ligand-bound ErbB1 and ErbB2 receptor tyrosine kinases in living cells using number and brightness analysis. Proceedings of the National Academy of Sciences of the United States of America. 107 (38), 16524-16529 (2010).
- Ming, A. Y., et al. Dynamics and Distribution of Klothobeta (KLB) and fibroblast growth factor receptor-1 (FGFR1) in living cells reveal the fibroblast growth factor-21 (FGF21)-induced receptor complex. Journal of Biological Chemistry. 287 (24), 19997-20006 (2012).
- Ross, J. A., et al. Oligomerization state of dynamin 2 in cell membranes using TIRF and number and brightness analysis. Biophysical Journal. 100 (3), L15-L17 (2011).
- Hellriegel, C., Caiolfa, V. R., Corti, V., Sidenius, N., Zamai, M. Number and brightness image analysis reveals ATF-induced dimerization kinetics of uPAR in the cell membrane. FASEB J. 25 (9), 2883-2897 (2011).
- Zamai, M., et al. Number and brightness analysis reveals that NCAM and FGF2 elicit different assembly and dynamics of FGFR1 in live cells. Journal of Cell Science. 132 (1), (2019).
- Hassler, K., et al. Total internal reflection fluorescence correlation spectroscopy (TIR-FCS) with low background and high count-rate per molecule. Optics Express. 13 (19), 7415-7423 (2005).
- Di Rienzo, C., Gratton, E., Beltram, F., Cardarelli, F. From fast fluorescence imaging to molecular diffusion law on live cell membranes in a commercial microscope. Journal of Visualized Experiments. (92), e51994 (2014).
- Beenken, A., Mohammadi, M. The FGF family: biology, pathophysiology and therapy. Nature Reviews Drug Discovery. 8 (3), 235-253 (2009).
- Joubert, J., Sharma, D. Light microscopy digital imaging. Current Protocols in Cytometry. , (2011).
- Gell, C., Berndt, M., Enderlein, J., Diez, S. TIRF microscopy evanescent field calibration using tilted fluorescent microtubules. Journal of Microscopy. 234 (1), 38-46 (2009).
- Burghardt, T. P. Measuring incidence angle for through-the-objective total internal reflection fluorescence microscopy. Journal of Biomedical Optics. 17 (12), 126007 (2012).
- Trullo, A., Corti, V., Arza, E., Caiolfa, V. R., Zamai, M. Application limits and data correction in number of molecules and brightness analysis. Microscopy Research and Technique. 76 (11), 1135-1146 (2013).
- Caiolfa, V. R., et al. Monomer-dimer dynamics and distribution of GPI-anchored uPAR are determined by cell surface protein assemblies. Journal of Cell Biology. 179 (5), 1067-1082 (2007).
- Campbell, R. E., et al. A monomeric red fluorescent protein. Proceedings of the National Academy of Sciences of the United States of America. 99 (12), 7877-7882 (2002).
- Cutrale, F., et al. Using enhanced number and brightness to measure protein oligomerization dynamics in live cells. Nature Protocols. 14 (2), 616-638 (2019).
- Dunsing, V., Chiantia, S. A Fluorescence Fluctuation Spectroscopy Assay of Protein-Protein Interactions at Cell-Cell Contacts. Journal of Visualized Experiments. (142), (2018).

