Динамика олигомеризации рецепторов поверхности клеток в живых клетках с помощью общей внутренней флуоресценции в сочетании с анализом числа и яркости
Summary
Мы описываем подход к визуализации для определения среднего олигомерного состояния mEGFP-тегами-рецепторами олигомеров, индуцированных связыванием лиганд в плазменной мембране живых клеток. Протокол основан на микроскопии Total Internal Reflection Fluorescence (TIRF) в сочетании с анализом числа и яркости (N и B).
Abstract
Несмотря на важность и повсеместность олигомеризации рецепторов, для обнаружения событий кластеризации и измерения степени кластеризации применяется несколько методов. Здесь мы описываем подход к визуализации для определения среднего олигомерного состояния mEGFP-тегами-рецепторами гомокомплексов в мембране живых клеток. Протокол основан на микроскопии Total Internal Reflection Fluorescence (TIRF) в сочетании с анализом числа и яркости (N и B). Н З/Б – это метод, похожий на флуоресценцию-корреляционную спектроскопию (FCS) и фотон, считающий гистограмму (PCH), которые основаны на статистическом анализе колебаний интенсивности флюорофорора, распространяющихся в и из освещения объема во время наблюдения. В частности, Н И Б является упрощением ПХГ для получения информации о среднем количестве белков в олигомерных смесях. Амплитуды интенсивности описываются молекулярной яркостью фторофора и средним количеством флюорофоров в объеме освещения. Таким образом, Н З/Б рассматривает только первый и второй моменты распределения амплитуды, а именно, средняя интенсивность и дисперсия. Это, в то же время, сила и слабость метода. Поскольку учитываются только два момента, Н И Б не может определить моляровую фракцию неизвестных олигомеров в смеси, но она оценивает только среднее состояние олигомеризации смеси. Тем не менее, он может быть применен к относительно небольшим сериям времени (по сравнению с другими методами) изображений живых клеток на пиксельной основе, просто отслеживая временные колебания интенсивности флуоресценции. Это уменьшает эффективное время на пиксель до нескольких микросекунд, что позволяет приобретения в диапазоне секунд до миллисекунд, что необходимо для быстрой кинетики олигомеризации. Наконец, можно исследовать большие клеточные области, а также субклеточные отсеки.
Introduction
Мы описываем подход к визуализации Total Internal Reflection Fluorescence-Number и Brightness (TIRF-N’amp;B) для определения среднего олигомерного состояния молекул рецепторов в плазменной мембране живых клеток, направленных на увязку рецепторной сборки динамика к биологической функции белков(Рисунок 1).
При внеклеточном связывании лиганд рецепторы инициируют трансдукцию внутриклеточного сигнала в зависимости от их конформации, олигомеризации, потенциальных корецепторов и мембранного состава. Несмотря на важность и повсеместность олигомеризации рецепторов, признанных ключевым событием в клеточной сигнализации1,2,3,4,5,6, 7, несколько методов могут обнаружить события кластеризации и измерить степень кластеризации экспериментально8,9. Конфокальный объем (x,y’300 nm, z z 900 nm) недостаточно решен для доказательства молекулярного взаимодействия и стоихиометрии, даже после оптимизации алгоритмами восстановления изображения10. Подедины состав белковых олигомеров не может быть решен на чисто пространственной основе даже с помощью супер-разрешения методов на х, разрешение 20-70 нм, таких как PALM11, STORM12, и STED13. Более того, их временное разрешение (в порядке минут на изображение) не может следовать за кинетиками в диапазоне секунд. Одномолекулярная ступенчатое отбеливание разрешает стойихиометрию белковых олигомеров только в том случае, если они неподвижны14.
Одним из наиболее универсальных методов измерения плотности и олигомеризации флуоресцентно отмеченных белков в пределах одного изображения является анализ распределения пространственной интенсивности (SpIDA), который опирается на пространственную выборку. Он применим как к химически фиксированным, так и к живым клеткам, и позволяет анализировать несколько областей, представляющих интерес для клетки, одновременно с помощью стандартной флуоресценции15. Кроме того, методы момента, такие как флуоресценция-корреляция спектроскопии (FCS)16, фотон подсчета гистограммы (PCH)17, и количество и яркость (N и B)18,19, подходят для количественного олигомера Измерения. Эти методы анализируют колебания интенсивности флуоресценции, которые могут наблюдаться во времени, когда фторфоры рассеивается в и из объема освещения. Амплитуда колебаний интенсивности можно однозначно описать молекулярной яркостью флюорофора (к) и средним количеством флюорофоров (n) в пределах объема освещения17 (Рисунок 2). Как правило, коэффициент диффузии фторфоров и среднее количество молекул (обратно связанных с значением G(0) в пределах объема освещения могут быть получены FCS20. Однако, так как время диффузии только масштабируется с кубической корнем массы, FCS не является достаточно чувствительным для обнаружения изменений в молекулярной массе21. На практике, один цвет FCS не может обнаружить димеризацию мембранных рецепторов. PCH разрешает смеси различных олигомеров точно. Используя более двух мгновений распределения амплитуды, он обнаруживает молекулы разной яркости, которые занимают один и тот же объем освещения. Сканирование FCS22 и разработки, такие как интересная парно-корреляция молекулярной яркости (pCOMB) подход23, введены для расширения диапазона применимости методов корреляции флуоресценции в биологических системах24 , остаются одноточечными методами, не имеющими возможности быстрых измерений на большой площади ячейки, требующих много последовательных наблюдений на каждом пикселе и получения данных в порядке секунд.
Н З/Б является упрощенной версией PCH, которая учитывает только первый и второй моменты амплитуды распределения флуоресценции, а именно, средняя интенсивность, lt; I’gt;, и дисперсия,No 2 (Рисунок 2)18,19 и, из-за этого, он не может определить моляровую фракцию неизвестных олигомеров в смеси, а лишь оценивает среднее состояние олигомеризации смеси. Тем не менее, Н И Б имеет то преимущество, что работает с относительно меньшими временные ряды изображений живых клеток, чем PCH на пиксель за пикселем основе, просто путем мониторинга колебаний во времени флуоресценции интенсивности. Поскольку Н И Б сокращает время за пикселем до нескольких микросекунд, он может следить за быстрой кинетикой олигомеризации на больших клеточных площадях, позволяя присугнять изображение в временной шкале секунд в сканирующей микроскопии (например, confocal, 2-фотон) и миллисекундах микроскопии на основе камеры (например, TIRFM).
В нескольких докладах была продемонстрирована способность Н И Б количественно очислять количество субъединиц в белковых кластерах с помощью визуализации расширенных клеточных регионов. Кластеры Paxillin-EGFP были обнаружены на участках адгезии в клетках CHO-K125,а внутриклеточная агрегация патогенного пептида Httex1p была описана в клетках COS-726. Н З/Б был применен для следующих лиганд-управляемых олигомеризации рецептора ErbB27, и влияние лиганд FGF21 на Klothob (KLB) и FGFR1c в клетках HeLa28. Сочетание tIRF изображений и N и B анализ был использован, чтобы показать, что динамин-2 в первую очередь тетрамерические всей клеточной мембраны29. Мы применили N и B как raster сканирования и TIRF изображения доказать лиганд-управляемый димеризации uPAR и FGFR1 клеточных мембранных рецепторов30,31.
Методы корреляции флуоресценции, такие как Н И Б, FCS и PCH, основаны на том, что в открытом томе число частиц следует за распределением Пуассона. Потому что только фотоны, что фторофоры излучают могут быть обнаружены, среднее значение для измеренной интенсивности флуоресценции по сравнению со временем в пикселе изображения,  является продуктом среднего количества флюорофоров в объеме освещения,, и их молекулярная яркость, No17:
является продуктом среднего количества флюорофоров в объеме освещения,, и их молекулярная яркость, No17:

где выражено как количество фотонов, испускаемых на единицу времени (обычно в секунду) на молекулу, когда молекула находится в центре объема освещения.
Яркость является свойством каждого фторофора в данном приобретении создана, в то время как интенсивность сумма всех взносов от всех фторофоров. В биологических конкурсах, яркость будет увеличиваться с увеличением числа флюорофоров, которые колеблются вместе, давая информацию о состоянии олигомеризации флуоресцентно помечены белка. Амплитуда колебаний на данном пикселе измеряется из дисперсии сигнала флуоресценции,No 2:

Где среднее значение квадрата  интенсивности, и квадрат среднего
интенсивности, и квадрат среднего  интенсивности, , вычисляются из индивидуальных значений интенсивности в каждом пикселе каждого кадра:
интенсивности, , вычисляются из индивидуальных значений интенсивности в каждом пикселе каждого кадра:
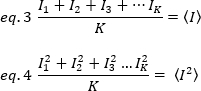
где K — это количество общих кадров в временных рядах. Экспериментально необходимо вычислить для всей серии изображений дисперсию, описывающая рассеяние значений индивидуальной интенсивности на каждом пикселе одного изображения вокруг значения средней интенсивности. Разница включает в себя все колебания разного происхождения. В первом приближении, дисперсия из-за рассеяния частиц в объеме освещения,20, могут быть отделены от дисперсии из-за шума выстрела детектора,2d. Эти два отклонения являются независимыми; таким образом, общая дисперсия дается их сумма:

Разница, из-за молекулярных колебаний в и из объема обнаружения, линейно зависит от молекулярной яркости и интенсивности:

Перестановка eq. 6 в соответствии с eq. 1:

Согласно типичной концепции флуоресценции корреляции спектроскопии, уравнение 7 гласит, что дисперсия из-за количества колебаний зависит от квадрата яркости частицы.
Затем, дисперсия из-за колебаний детектора является линейной функцией обнаруженной интенсивности, при предположении, что детектор работает ниже предела насыщения19:

В случае фотонововых детекторов 1и с0, таким образом, дисперсия детектора равна средней интенсивности:

Чтобы применить эти понятия к реальным измерениям в живых клетках, Gratton и его коллеги18 определяют кажущуюся яркость, B, для каждого пикселя как соотношение дисперсии над средней интенсивностью:

B является параметр, который измеряется экспериментально. В этой работе, временные ряды изображения рецепторов FGFR1 на плазменной мембране клеток HeLa захвачены tIRF микроскопии и средняя кажущаяся яркость, B, определяется анализом N и B. Затем, после добавления FGF2, последовательные временные ряды фиксируются, чтобы следить за изменениями в самосборке молекул рецепторов на поверхности мембраны после стимуляции рецептора каноническим лигандой.
Однако, поскольку детектором микроскопа TIRF является камера EMCCD, выражение видимой яркости должно быть изменено как19:

где смещение является смещение интенсивности обнаружения электроники, что является характерной для настроек детектора. Дисперсия и средняя интенсивность для аналогового детектора, соответственно, даются:
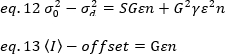
где G является аналоговым увеличением в цифровых уровнях (DL/фотоны), S, цифровые уровни на фотон19, дается наклон интенсивности против дисперсии участка для источника света с постоянной интенсивностью (без временных колебаний). Коэффициент q связан с формой объема обнаружения пикселей. По данным Hassler et al.32, коэффициент q равен 0,3 для tIRF изображения, работающего при максимальном выигрыше камеры обнаружения19. Параметры смещения, S и G являются характеристиками камеры и микроскопа. Очевидная яркость, B, получена путем перестановки eq. 11 в соответствии с eq. 12 и 13:

Экспериментально, является сложной функцией интенсивности лазера и эффективности обнаружения системы. Тем не менее, поскольку B/S линейно зависит от К, важно определить только относительную стоимость к для данного режима обнаружения:

где « пропорционально К. Тем не менее, калибровка выполняется с помощью внутренней ссылки.
Protocol
Representative Results
Discussion
Н ЗБи требует нескольких мер предосторожности при выборе модели ячейки и стратегии маркировки. Он может быть применен только к живым клеткам, которые остаются пристежными во время захвата изображения. Дополнительные колебания из-за всего жесткого смещения ячейки могут быть обработан?…
Offenlegungen
The authors have nothing to disclose.
Acknowledgements
CNIC поддерживается Министерством Cencia, Innovacion y Universidades и Фондом Pro CNIC, а также является Центром передового опыта Северо-Очоа (SEV-2015-0505). Нас также поддерживает Европейский фонд регионального развития (ФЕДЕР) «Una manera de hacer Europa». UC признает поддержку со стороны Associazione Italiana Ricerca sul Cancro, Ассоциации международных исследований рака (теперь известный как Всемирное исследование рака), и итальянского министерства здравоохранения. A.T. признать Fondazione Banca дель Монте ди Ломбардия для частичной поддержки его работы с. стипендий Progetto Professionalit Ивано Бекки 2011-2012.
Materials
| 3-Colour Fast TIRF Leica AM TIRF MC inverted microscope, with smi-automatic TIRF alignment. The microscope is equipped with a diode 488 nm laser, a 100×1.46 oil TIRF objective, Ex/Em Bandpass filters at 490/20 and 525/50, temperature/CO2 incubator and Andor DU 8285 VP EMCCD camera. The microscope is operated by Leica LIF software. | Leica Microsystems, Wetzlar, Germany | ||
| Albumin from Bovine Serum 98% minimun | Sigma-Aldrich, St. Louis, MI, USA | A7906-100G | |
| DMEM without Phenol Red with 25 mM HEPES | GIBCO Thermo Fisher Scientific,Waltham, MA, USA | 21063029 | Used serum free for microscopy |
| DMEM high-glucose GlutaMAX I | GIBCO Thermo Fisher Scientific,Waltham, MA, USA | 10566-016 | Used for complete medium |
| Dulbecco's Phosphate Buffered Saline 10x (PBS) | Biowest, Nuaillé, France | X0515-500 | |
| Emission splitting system Photometrics DV2 | TeledynePhotometrics, Tucson, AZ, USA | ||
| Fetal Bovine Serum, qualified, Brazil | GIBCO Thermo Fisher Scientific,Waltham, MA, USA | 10270106 | 10% inactivated supplement for complete medium |
| Glass bottom 35-mm sterile 1.5 dishes | MatTek, Ashland, MA, USA | P35G-0.170-14-C | uncoated, glass thickness 0.17 microns |
| GraphPad Prism | GraphPad Software Inc., San Diego, CA, USA | ||
| Human cervical carcinoma (HeLa), serum-free animal component (AC) cells | Millipore-Sigma ECACC, Darmstadt, Germany | CB_08011102 | |
| iXonEM+ 897 EMCCD (back-illuminated) ANDOR camera controlled by ANDOR Solis software | Oxford Instruments, Andor TM Technology, Abingdon-on-Thames, UK | This camera, installed in an additional port of the microscope, is used for acquiring the N&B time series | |
| Matlab Executable N&B routine | Unit of Microscopy and Dynamic Imaging, CNIC, Madrid, Spain | download at https://www.cnic.es/en/investigacion/2/1187/tecnologia | |
| MatLab v.2018b | The MathWorks, Inc. Natick, MA, USA | download at https://www.mathworks.com/products/matlab.html | |
| Penicillin:Streptomycin for tissue culture 100x | Biowhittaker Inc. Walkersville, MD, USA | LONZA 17-602E | supplement for medium at Penicillin/Streptomycin 100U/100µg. |
| pN1-mEGFP-FGFR1 expression vector | Unit of Gynecological Oncology Research, European Institute of Oncology IRCCS, Milan, Italy | Zamai et al., 2019 | |
| pN1-N-Gly-mEGFP-GPI expression vector | Unit of Microscopy and Dynamic Imaging, CNIC, Madrid, Spain | Hellriegel et al., 2011 | |
| pN1-N-Gly-mEGFP-mEGFP-GPI expression vector | Unit of Microscopy and Dynamic Imaging, CNIC, Madrid, Spain | Hellriegel et al., 2011 | |
| Recombinant FGF2 | PeproTech EC, Ltd., London, UK | Ligand solution: 20ng/mL of FGF2 in PBS supplemented with 0.01%BSA. | |
| Sodium pyruvate GIBCO | ThermoFisher Scientific | 11360070 | 1mM supplement for medium |
| TransIt-LT1 Transfection Reagent | MirusBio LLC, Madison, WI, USA | MIR 2300 | |
| Trypsin-EDTA (0.25%), phenol red | GIBCO Thermo Fisher Scientific,Waltham, MA, USA | 25200056 | |
| Type F Immersion liquid 10 mL | Leica Microsystems, Wetzlar, Germany | 11513 859 | |
| UltraPure BSA (50 mg/mL) | ThermoFisher Scientific | AM2618 | 0.1% supplement for medium without phenol red used for transfections |
Referenzen
- Agwuegbo, U. C., Jonas, K. C. Molecular and functional insights into gonadotropin hormone receptor dimerization and oligomerization. Minerva Ginecologica. 70 (5), 539-548 (2018).
- Ferre, S., et al. G protein-coupled receptor oligomerization revisited: functional and pharmacological perspectives. Pharmacological Reviews. 66 (2), 413-434 (2014).
- Marsango, S., Ward, R. J., Alvarez-Curto, E., Milligan, G. Muscarinic receptor oligomerization. Neuropharmacology. 136 (Pt C), 401-410 (2018).
- Oishi, A., Cecon, E., Jockers, R. Melatonin Receptor Signaling: Impact of Receptor Oligomerization on Receptor Function. International Review of Cell and Molecular Biology. 338, 59-77 (2018).
- Thelen, M., Munoz, L. M., Rodriguez-Frade, J. M., Mellado, M. Chemokine receptor oligomerization: functional considerations. Current Opinion in Pharmacology. 10 (1), 38-43 (2010).
- Van Craenenbroeck, K. GPCR oligomerization: contribution to receptor biogenesis. Subcellular Biochemistry. 63, 43-65 (2012).
- Wnorowski, A., Jozwiak, K. Homo- and hetero-oligomerization of beta2-adrenergic receptor in receptor trafficking, signaling pathways and receptor pharmacology. Cell Signaling Technology. 26 (10), 2259-2265 (2014).
- Fricke, F., Dietz, M. S., Heilemann, M. Single-molecule methods to study membrane receptor oligomerization. Chemphyschem. 16 (4), 713-721 (2015).
- Vidi, P. A., Ejendal, K. F., Przybyla, J. A., Watts, V. J. Fluorescent protein complementation assays: new tools to study G protein-coupled receptor oligomerization and GPCR-mediated signaling. Molecular and Cellular Endocrinology. 331 (2), 185-193 (2011).
- Trussell, H. J., et al., Trussell, J., et al. . Academic Press Library in Signal Processing. 4, 3-9 (2014).
- Betzig, E., et al. Imaging intracellular fluorescent proteins at nanometer resolution. Science. 313 (5793), 1642-1645 (2006).
- Rust, M. J., Bates, M., Zhuang, X. Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy (STORM). Nature Methods. 3 (10), 793-795 (2006).
- Nagerl, U. V., Willig, K. I., Hein, B., Hell, S. W., Bonhoeffer, T. Live-cell imaging of dendritic spines by STED microscopy. Proceedings of the National Academy of Sciences of the United States of America. 105 (48), 18982-18987 (2008).
- Tsekouras, K., Custer, T. C., Jashnsaz, H., Walter, N. G., Presse, S. A novel method to accurately locate and count large numbers of steps by photobleaching. Molecular Biology of the Cell. 27 (22), 3601-3615 (2016).
- Godin, A. G., et al. Revealing protein oligomerization and densities in situ using spatial intensity distribution analysis. Proceedings of the National Academy of Sciences of the United States of America. 108 (17), 7010-7015 (2011).
- Qian, H., Elson, E. L. Distribution of molecular aggregation by analysis of fluctuation moments. Proceedings of the National Academy of Sciences of the United States of America. 87 (14), 5479-5483 (1990).
- Chen, Y., Muller, J. D., So, P. T., Gratton, E. The photon counting histogram in fluorescence fluctuation spectroscopy. Biophysical Journal. 77 (1), 553-567 (1999).
- Dalal, R. B., Digman, M. A., Horwitz, A. F., Vetri, V., Gratton, E. Determination of particle number and brightness using a laser scanning confocal microscope operating in the analog mode. Microscopy Research and Technique. 71 (1), 69-81 (2008).
- Unruh, J. R., Gratton, E. Analysis of molecular concentration and brightness from fluorescence fluctuation data with an electron multiplied CCD camera. Biophysical Journal. 95 (11), 5385-5398 (2008).
- Hess, S. T., Huang, S., Heikal, A. A., Webb, W. W. Biological and chemical applications of fluorescence correlation spectroscopy: a review. Biochemie. 41 (3), 697-705 (2002).
- Muller, J. D., Chen, Y., Gratton, E. Fluorescence correlation spectroscopy. Methods in Enzymology. 361, 69-92 (2003).
- Levi, V., Ruan, Q., Kis-Petikova, K., Gratton, E. Scanning FCS, a novel method for three-dimensional particle tracking. Biochemical Society Transactions. 31 (Pt 5), 997-1000 (2003).
- Hinde, E., et al. Quantifying the dynamics of the oligomeric transcription factor STAT3 by pair correlation of molecular brightness. Nature Communications. 7, 11047 (2016).
- Waithe, D., et al. Optimized processing and analysis of conventional confocal microscopy generated scanning FCS data. Methods. 140-141, 62-73 (2018).
- Digman, M. A., Dalal, R., Horwitz, A. F., Gratton, E. Mapping the number of molecules and brightness in the laser scanning microscope. Biophysical Journal. 94 (6), 2320-2332 (2008).
- Ossato, G., et al. A two-step path to inclusion formation of huntingtin peptides revealed by number and brightness analysis. Biophysical Journal. 98 (12), 3078-3085 (2010).
- Nagy, P., Claus, J., Jovin, T. M., Arndt-Jovin, D. J. Distribution of resting and ligand-bound ErbB1 and ErbB2 receptor tyrosine kinases in living cells using number and brightness analysis. Proceedings of the National Academy of Sciences of the United States of America. 107 (38), 16524-16529 (2010).
- Ming, A. Y., et al. Dynamics and Distribution of Klothobeta (KLB) and fibroblast growth factor receptor-1 (FGFR1) in living cells reveal the fibroblast growth factor-21 (FGF21)-induced receptor complex. Journal of Biological Chemistry. 287 (24), 19997-20006 (2012).
- Ross, J. A., et al. Oligomerization state of dynamin 2 in cell membranes using TIRF and number and brightness analysis. Biophysical Journal. 100 (3), L15-L17 (2011).
- Hellriegel, C., Caiolfa, V. R., Corti, V., Sidenius, N., Zamai, M. Number and brightness image analysis reveals ATF-induced dimerization kinetics of uPAR in the cell membrane. FASEB J. 25 (9), 2883-2897 (2011).
- Zamai, M., et al. Number and brightness analysis reveals that NCAM and FGF2 elicit different assembly and dynamics of FGFR1 in live cells. Journal of Cell Science. 132 (1), (2019).
- Hassler, K., et al. Total internal reflection fluorescence correlation spectroscopy (TIR-FCS) with low background and high count-rate per molecule. Optics Express. 13 (19), 7415-7423 (2005).
- Di Rienzo, C., Gratton, E., Beltram, F., Cardarelli, F. From fast fluorescence imaging to molecular diffusion law on live cell membranes in a commercial microscope. Journal of Visualized Experiments. (92), e51994 (2014).
- Beenken, A., Mohammadi, M. The FGF family: biology, pathophysiology and therapy. Nature Reviews Drug Discovery. 8 (3), 235-253 (2009).
- Joubert, J., Sharma, D. Light microscopy digital imaging. Current Protocols in Cytometry. , (2011).
- Gell, C., Berndt, M., Enderlein, J., Diez, S. TIRF microscopy evanescent field calibration using tilted fluorescent microtubules. Journal of Microscopy. 234 (1), 38-46 (2009).
- Burghardt, T. P. Measuring incidence angle for through-the-objective total internal reflection fluorescence microscopy. Journal of Biomedical Optics. 17 (12), 126007 (2012).
- Trullo, A., Corti, V., Arza, E., Caiolfa, V. R., Zamai, M. Application limits and data correction in number of molecules and brightness analysis. Microscopy Research and Technique. 76 (11), 1135-1146 (2013).
- Caiolfa, V. R., et al. Monomer-dimer dynamics and distribution of GPI-anchored uPAR are determined by cell surface protein assemblies. Journal of Cell Biology. 179 (5), 1067-1082 (2007).
- Campbell, R. E., et al. A monomeric red fluorescent protein. Proceedings of the National Academy of Sciences of the United States of America. 99 (12), 7877-7882 (2002).
- Cutrale, F., et al. Using enhanced number and brightness to measure protein oligomerization dynamics in live cells. Nature Protocols. 14 (2), 616-638 (2019).
- Dunsing, V., Chiantia, S. A Fluorescence Fluctuation Spectroscopy Assay of Protein-Protein Interactions at Cell-Cell Contacts. Journal of Visualized Experiments. (142), (2018).

