Dinâmica de oligomerização dos receptores de superfície celular em células vivas pela microscopia de fluorescência de reflexão interna total combinada com análise de número e brilho
Summary
Descrevemos uma abordagem de imagem para a determinação do estado oligomérico médio de oligomeros de receptores marcados por mEGFP induzidos pela ligação de ligand na membrana plasmática das células vivas. O protocolo é baseado na microscopia de Fluorescência Total de Reflexão Interna (TIRF) combinada com análise de Número e Brilho (N&B).
Abstract
Apesar da importância e ubiquidade da oligomerização do receptor, poucos métodos são aplicáveis para detectar eventos de agrupamento e medir o grau de agrupamento. Aqui, descrevemos uma abordagem de imagem para determinar o estado oligomérico médio de homocomplexos do receptor marcado pelo mEGFP na membrana das células vivas. O protocolo é baseado na microscopia de Fluorescência Total de Reflexão Interna (TIRF) combinada com análise de Número e Brilho (N&B). A N&B é um método semelhante à espectroscopia fluorescência-correlação (FCS) e fótons contando histograma (PCH), que são baseados na análise estatística das flutuações da intensidade da fluorescência da fluorofia difundindo dentro e fora de uma iluminação volume durante um tempo de observação. Em particular, a N&B é uma simplificação da PCH para obter informações sobre o número médio de proteínas em misturas oligoméricas. As amplitudes da flutuação da intensidade são descritas pelo brilho molecular do fluorophore e pelo número médio de fluorophores dentro do volume da iluminação. Assim, a N&B considera apenas o primeiro e segundo momentos da distribuição de amplitude, ou seja, a intensidade média e a variação. Esta é, ao mesmo tempo, a força e a fraqueza do método. Como apenas dois momentos são considerados, a N&B não pode determinar a fração molar de oligomeros desconhecidos em uma mistura, mas estima apenas o estado médio de oligomerização da mistura. No entanto, ele pode ser aplicado a séries de tempo relativamente pequenas (em comparação com outros métodos de momento) de imagens de células vivas em uma base pixel-a-pixel, simplesmente monitorando as flutuações de tempo da intensidade da fluorescência. Ele reduz o tempo eficaz por pixel para alguns microssegundos, permitindo a aquisição no intervalo de tempo de segundos a milissegundos, o que é necessário para cinética de oligomerização rápida. Finalmente, grandes áreas celulares, bem como compartimentos subcelulares podem ser explorados.
Introduction
Descrevemos uma abordagem de imagem total de reflexão interna fluorescência-número e brilho (TIRF-N&B) para determinar o estado oligomérico médio das moléculas receptoras na membrana plasmática das células vivas, com o objetivo de vincular a montagem do receptor dinâmica para a função biológica das proteínas (Figura 1).
Após a ligação extracelular do ligand, os receptores iniciam a transdução de sinal intracelular dependendo de sua conformação, oligomerização, potenciais co-receptores e composição da membrana. Apesar da importância e ubiquidade da oligomerização do receptor, reconhecida como um evento-chave na sinalização celular1,2,3,4,5,6, 7,poucos métodos podem detectar eventos de agrupamento e medir o grau de agrupamento experimentalmente8,9. O volume confocal (x, y – 300 nm, z 900 nm) é insuficientemente resolvido para provar a interação molecular e stoichiometria, mesmo após a otimização por algoritmos de restauração de imagem10. A composição sub-unitária de oligomeros proteicos não pode ser resolvida de forma puramente espacial, mesmo por métodos de super-resolução em x,y resolução de 20-70 nm, como PALM11, STORM12,e STED13. Além disso, sua resolução temporal (na ordem dos minutos por imagem) não pode seguir a cinética na faixa de segundos. Único molécula passo-branqueamento resolve a stoichiometria de oligomeros de proteína apenas se eles são imóvel14.
Um dos métodos mais versáteis para medir a densidade e o oligomerização de proteínas fluorescentes marcadas dentro de imagens individuais é a análise de distribuição de intensidade espacial (SpIDA), que depende da amostragem espacial. É aplicável a células quimicamente fixas e vivas, e permite a análise de várias regiões de interesse da célula simultaneamente usando microscopia fluorescência padrão15. Alternativamente, métodos momentâneos, como espectroscopia fluorescência-correlação (FCS)16,fótons contando histograma (PCH)17, e Número e Brilho (N&B)18,19, são adequados para oligomer quantitativo Medidas. Esses métodos analisam as flutuações de intensidade da fluorescência que podem ser observadas no tempo em que os fluorofforas se difundem dentro e fora de um volume de iluminação. As amplitudes das flutuações de intensidade podem ser descritas exclusivamente pelo brilho molecular do fluorofforre (ε) e pelo número médio de fluorofônicos (n) dentro do volume de iluminação17 (Figura 2). Normalmente, o coeficiente de difusão dos fluorofforos e o número médio de moléculas (inversamente relacionadas ao valor G(0) dentro do volume de iluminação podem ser obtidos pela FCS20. No entanto, uma vez que o tempo de difusão apenas escalas com a raiz cúbica da massa, FCS não é suficientemente sensível para detectar alterações na massa molecular21. Na prática, a FCS de cor única não consegue detectar a dimerização dos receptores de membrana. PCH resolve misturas de diferentes oligomeros com precisão. Usando mais de dois momentos da distribuição da amplitude, detecta moléculas do brilho diferente que ocupam o mesmo volume da iluminação. Digitalização FCS22 e desenvolvimentos, como a interessante par-correlação de brilho molecular (pCOMB) abordagem23, introduzido para estender a gama de aplicabilidade dos métodos de correlação fluorescência em sistemas biológicos24 , permanecem métodos de ponto único sem a capacidade de medições rápidas em uma grande área de uma célula, exigindo muitas observações consecutivas em cada pixel e aquisição de dados na ordem de segundos.
A N&B é uma versão simplificada do PCH que considera apenas o primeiro e segundo momentos da amplitude da distribuição de fluorescência, ou seja, a intensidade média, e a variação, σ2 (Figura 2)18,19 e, por causa disso, não pode determinar a fração molar de oligomeros desconhecidos em uma mistura, mas estima somente o estado médio do oligomerization da mistura. No entanto, a N&B tem a vantagem de trabalhar com uma série de imagens de imagens de células vivas relativamente menores do que a PCH em uma base pixel-a-pixel, simplesmente monitorando as flutuações no tempo da intensidade da fluorescência. Como a N&B reduz o tempo por pixel para alguns microssegundos, ela pode seguir cinética de oligomerização rápida sobre grandes áreas celulares, permitindo a aquisição de imagem em uma escala de tempo de segundos em microscopia de varredura de raster (por exemplo, confocal, 2 fótons) e milissegundos em microscopia baseada em câmera (por exemplo, TIRFM).
Vários relatos demonstraram a capacidade da N&B de quantificar o número de subunidades em aglomerados de proteínas por meio de imagens de regiões celulares estendidas. Os clusters Paxillin-EGFP foram detectados nos locais de adesão nas células CHO-K125,e a agregação intracelular do peptídeo httex1p patogênico foi descrita nas células COS-726. A N&B foi aplicada para seguir a oligomerização ativa da ligand do receptorErbB 27,e o efeito do ligand FGF21 em Klothob (KLB) e fgfr1c em célulasHeLa 28. A combinação de imagens tirf e análise de N&B foi usada para mostrar que a dinamina-2 é principalmente tetramérica em toda a membrana celular29. Aplicamos a N&B a imagens de varredura de raster e TIRF para provar a dimerização conduzida por ligand dos receptores de membrana celular uPAR e FGFR130,31.
Métodos de correlação de fluorescência, como N&B, FCS e PCH, são baseados na noção de que, em um volume aberto, o número de bolhas de partículas segue uma distribuição de Poisson. Como somente os fótons que os fluorofônicos emitem podem ser detectados, o valor médio para uma intensidade  de fluorescência medida versus o tempo em um pixel da imagem, é o produto do número médio de fluorofosforas no volume de iluminação, n, e sua brilho molecular, ε17:
de fluorescência medida versus o tempo em um pixel da imagem, é o produto do número médio de fluorofosforas no volume de iluminação, n, e sua brilho molecular, ε17:

onde o ε é expresso como o número de fótons emitidos por unidade de tempo (convencionalmente por segundo) por molécula quando a molécula está no centro do volume de iluminação.
O brilho é propriedade de cada fluorofofóbico em uma determinada aquisição criada, enquanto a intensidade é a soma de todas as contribuições de todos os fluorofos. Em competições biológicas, o brilho aumentará com o aumento do número de fluorofos que flutuam junto, dando a informação no estado do oligomerization da proteína fluorescente-etiquetada. As amplitudes de flutuação em um determinado pixel é medido a partir da variação do sinal de fluorescência, σ2:

Onde a média do quadrado  de intensidade, e o quadrado
de intensidade, e o quadrado  da média de intensidade, são computados a partir dos valores de intensidade individual em cada pixel de cada quadro:
da média de intensidade, são computados a partir dos valores de intensidade individual em cada pixel de cada quadro:
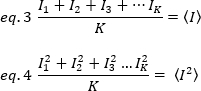
onde K é o número de quadros totais na série de tempo. Experimentalmente, é necessário calcular para toda a série de imagens a variação que descreve a dispersão dos valores de intensidade individual em cada pixel de uma única imagem em torno do valor de intensidade média. A variação inclui todas as flutuações de diferentes origens. Em uma primeira aproximação, a variação devido às partículas de difusão no volume de iluminação, σ20,pode ser separada da variância devido ao ruído do tiro do detector, σ2d. As duas variações são independentes; assim, a variação total é dada por sua soma:

A variação, devido a flutuações moleculares dentro e fora do volume de detecção, é linearmente dependente do brilho e intensidade moleculares:

Reorganizando eq. 6 de acordo com o eq. 1:

De acordo com o conceito típico de espectroscopia de correlação de fluorescência, a equação 7 afirma que a variação devido ao número de flutuações depende do quadrado do brilho das partículas.
Em seguida, a variação devido às flutuações do detector é uma função linear da intensidade detectada, a suposição de que o detector é operado abaixo de seu limite de saturação19:

No caso dos detectores de contagem de fótons =1e c=0, assim a variação do detector é igual à intensidade média:

Para aplicar esses conceitos a medições reais em células vivas, Gratton e colegas18 definem o brilho aparente, B, para cada pixel como a proporção da variação sobre a intensidade média:

B é o parâmetro que é medido experimentalmente. Neste trabalho, imagens de séries de tempo de receptores FGFR1 na membrana plasmática das células HeLa são capturadas pela microscopia TIRF e o brilho aparente médio, B, é determinado pela análise de N&B. Então, após a adição de FGF2, séries de tempo consecutivos são capturadas para acompanhar as mudanças na auto-montagem das moléculas receptoras na superfície da membrana após a estimulação do receptor com o ligand canônico.
No entanto, uma vez que o detector do microscópio TIRF é uma câmera EMCCD, a expressão para o brilho aparente precisa ser modificada como19:

onde o deslocamento é o deslocamento da intensidade da eletrônica da deteção que é uma característica das ajustes do detetor. A variação e a intensidade média de um detector analógico são, respectivamente, dadas por:
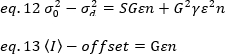
onde G é o ganho analógico em níveis digitais (DL/fótons), S, os níveis digitais por fóton19, é dado pela inclinação de uma intensidade versus variação parcela para uma fonte de luz com intensidade constante (sem flutuações temporais). O fator γ está relacionado à forma do volume de detecção de pixels. De acordo com Hassler et al.32,o fator γ é igual a 0,3 para imagens TIRF trabalhando no ganho máximo da câmera de detecção19. Os parâmetros de deslocamento, S e G são características da câmera e do microscópio. O brilho aparente, B, é obtido reorganizando eq. 11 de acordo com eq. 12 e 13:

Experimentalmente, o ε é uma função complexa da intensidade do laser e da eficiência de detecção do sistema. No entanto, uma vez que B / S é linearmente dependente de ε, só é importante determinar o valor relativo do ε para um determinado modo de detecção:

onde o ε’ é proporcional ao ε. Ainda assim, uma calibração é realizada usando uma referência interna.
Protocol
Representative Results
Discussion
A N&B requer várias precauções na escolha do modelo celular e da estratégia de rotulagem. Ele pode ser aplicado apenas para células vivas que permanecem aderidas de forma enova durante o tempo de captura de imagem. Flutuações extras devido ao deslocamento rígido de toda a célula podem ser tratadas com abordagens adequadas de restauração de imagem38. No entanto, geralmente quando uma célula se move, a membrana celular também se deforma, e a deformação da estrutura, produzindo grande …
Offenlegungen
The authors have nothing to disclose.
Acknowledgements
O CNIC é apoiado pelo Ministério de Ciencia, Innovación y Universidades e pela Fundação Pro CNIC, e é um Centro de Excelência Severo Ochoa (SEV-2015-0505). Também somos apoiados pelo Fundo Europeu de Desenvolvimento Regional (FEDER) “Una manera de hacer Europa”. A UC reconhece o apoio da Associazione Italiana Ricerca sul Cancro, da Association for International Cancer Research (agora conhecida como Worldwide Cancer Research) e do Ministério da Saúde italiano. A.T. reconhece o “Fondazione Banca del Monte di Lombardia” por apoiar parcialmente seu trabalho com a Bolsa PV “Progetto Professionalità Ivano Becchi” 2011-2012.
Materials
| 3-Colour Fast TIRF Leica AM TIRF MC inverted microscope, with smi-automatic TIRF alignment. The microscope is equipped with a diode 488 nm laser, a 100×1.46 oil TIRF objective, Ex/Em Bandpass filters at 490/20 and 525/50, temperature/CO2 incubator and Andor DU 8285 VP EMCCD camera. The microscope is operated by Leica LIF software. | Leica Microsystems, Wetzlar, Germany | ||
| Albumin from Bovine Serum 98% minimun | Sigma-Aldrich, St. Louis, MI, USA | A7906-100G | |
| DMEM without Phenol Red with 25 mM HEPES | GIBCO Thermo Fisher Scientific,Waltham, MA, USA | 21063029 | Used serum free for microscopy |
| DMEM high-glucose GlutaMAX I | GIBCO Thermo Fisher Scientific,Waltham, MA, USA | 10566-016 | Used for complete medium |
| Dulbecco's Phosphate Buffered Saline 10x (PBS) | Biowest, Nuaillé, France | X0515-500 | |
| Emission splitting system Photometrics DV2 | TeledynePhotometrics, Tucson, AZ, USA | ||
| Fetal Bovine Serum, qualified, Brazil | GIBCO Thermo Fisher Scientific,Waltham, MA, USA | 10270106 | 10% inactivated supplement for complete medium |
| Glass bottom 35-mm sterile 1.5 dishes | MatTek, Ashland, MA, USA | P35G-0.170-14-C | uncoated, glass thickness 0.17 microns |
| GraphPad Prism | GraphPad Software Inc., San Diego, CA, USA | ||
| Human cervical carcinoma (HeLa), serum-free animal component (AC) cells | Millipore-Sigma ECACC, Darmstadt, Germany | CB_08011102 | |
| iXonEM+ 897 EMCCD (back-illuminated) ANDOR camera controlled by ANDOR Solis software | Oxford Instruments, Andor TM Technology, Abingdon-on-Thames, UK | This camera, installed in an additional port of the microscope, is used for acquiring the N&B time series | |
| Matlab Executable N&B routine | Unit of Microscopy and Dynamic Imaging, CNIC, Madrid, Spain | download at https://www.cnic.es/en/investigacion/2/1187/tecnologia | |
| MatLab v.2018b | The MathWorks, Inc. Natick, MA, USA | download at https://www.mathworks.com/products/matlab.html | |
| Penicillin:Streptomycin for tissue culture 100x | Biowhittaker Inc. Walkersville, MD, USA | LONZA 17-602E | supplement for medium at Penicillin/Streptomycin 100U/100µg. |
| pN1-mEGFP-FGFR1 expression vector | Unit of Gynecological Oncology Research, European Institute of Oncology IRCCS, Milan, Italy | Zamai et al., 2019 | |
| pN1-N-Gly-mEGFP-GPI expression vector | Unit of Microscopy and Dynamic Imaging, CNIC, Madrid, Spain | Hellriegel et al., 2011 | |
| pN1-N-Gly-mEGFP-mEGFP-GPI expression vector | Unit of Microscopy and Dynamic Imaging, CNIC, Madrid, Spain | Hellriegel et al., 2011 | |
| Recombinant FGF2 | PeproTech EC, Ltd., London, UK | Ligand solution: 20ng/mL of FGF2 in PBS supplemented with 0.01%BSA. | |
| Sodium pyruvate GIBCO | ThermoFisher Scientific | 11360070 | 1mM supplement for medium |
| TransIt-LT1 Transfection Reagent | MirusBio LLC, Madison, WI, USA | MIR 2300 | |
| Trypsin-EDTA (0.25%), phenol red | GIBCO Thermo Fisher Scientific,Waltham, MA, USA | 25200056 | |
| Type F Immersion liquid 10 mL | Leica Microsystems, Wetzlar, Germany | 11513 859 | |
| UltraPure BSA (50 mg/mL) | ThermoFisher Scientific | AM2618 | 0.1% supplement for medium without phenol red used for transfections |
Referenzen
- Agwuegbo, U. C., Jonas, K. C. Molecular and functional insights into gonadotropin hormone receptor dimerization and oligomerization. Minerva Ginecologica. 70 (5), 539-548 (2018).
- Ferre, S., et al. G protein-coupled receptor oligomerization revisited: functional and pharmacological perspectives. Pharmacological Reviews. 66 (2), 413-434 (2014).
- Marsango, S., Ward, R. J., Alvarez-Curto, E., Milligan, G. Muscarinic receptor oligomerization. Neuropharmacology. 136 (Pt C), 401-410 (2018).
- Oishi, A., Cecon, E., Jockers, R. Melatonin Receptor Signaling: Impact of Receptor Oligomerization on Receptor Function. International Review of Cell and Molecular Biology. 338, 59-77 (2018).
- Thelen, M., Munoz, L. M., Rodriguez-Frade, J. M., Mellado, M. Chemokine receptor oligomerization: functional considerations. Current Opinion in Pharmacology. 10 (1), 38-43 (2010).
- Van Craenenbroeck, K. GPCR oligomerization: contribution to receptor biogenesis. Subcellular Biochemistry. 63, 43-65 (2012).
- Wnorowski, A., Jozwiak, K. Homo- and hetero-oligomerization of beta2-adrenergic receptor in receptor trafficking, signaling pathways and receptor pharmacology. Cell Signaling Technology. 26 (10), 2259-2265 (2014).
- Fricke, F., Dietz, M. S., Heilemann, M. Single-molecule methods to study membrane receptor oligomerization. Chemphyschem. 16 (4), 713-721 (2015).
- Vidi, P. A., Ejendal, K. F., Przybyla, J. A., Watts, V. J. Fluorescent protein complementation assays: new tools to study G protein-coupled receptor oligomerization and GPCR-mediated signaling. Molecular and Cellular Endocrinology. 331 (2), 185-193 (2011).
- Trussell, H. J., et al., Trussell, J., et al. . Academic Press Library in Signal Processing. 4, 3-9 (2014).
- Betzig, E., et al. Imaging intracellular fluorescent proteins at nanometer resolution. Science. 313 (5793), 1642-1645 (2006).
- Rust, M. J., Bates, M., Zhuang, X. Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy (STORM). Nature Methods. 3 (10), 793-795 (2006).
- Nagerl, U. V., Willig, K. I., Hein, B., Hell, S. W., Bonhoeffer, T. Live-cell imaging of dendritic spines by STED microscopy. Proceedings of the National Academy of Sciences of the United States of America. 105 (48), 18982-18987 (2008).
- Tsekouras, K., Custer, T. C., Jashnsaz, H., Walter, N. G., Presse, S. A novel method to accurately locate and count large numbers of steps by photobleaching. Molecular Biology of the Cell. 27 (22), 3601-3615 (2016).
- Godin, A. G., et al. Revealing protein oligomerization and densities in situ using spatial intensity distribution analysis. Proceedings of the National Academy of Sciences of the United States of America. 108 (17), 7010-7015 (2011).
- Qian, H., Elson, E. L. Distribution of molecular aggregation by analysis of fluctuation moments. Proceedings of the National Academy of Sciences of the United States of America. 87 (14), 5479-5483 (1990).
- Chen, Y., Muller, J. D., So, P. T., Gratton, E. The photon counting histogram in fluorescence fluctuation spectroscopy. Biophysical Journal. 77 (1), 553-567 (1999).
- Dalal, R. B., Digman, M. A., Horwitz, A. F., Vetri, V., Gratton, E. Determination of particle number and brightness using a laser scanning confocal microscope operating in the analog mode. Microscopy Research and Technique. 71 (1), 69-81 (2008).
- Unruh, J. R., Gratton, E. Analysis of molecular concentration and brightness from fluorescence fluctuation data with an electron multiplied CCD camera. Biophysical Journal. 95 (11), 5385-5398 (2008).
- Hess, S. T., Huang, S., Heikal, A. A., Webb, W. W. Biological and chemical applications of fluorescence correlation spectroscopy: a review. Biochemie. 41 (3), 697-705 (2002).
- Muller, J. D., Chen, Y., Gratton, E. Fluorescence correlation spectroscopy. Methods in Enzymology. 361, 69-92 (2003).
- Levi, V., Ruan, Q., Kis-Petikova, K., Gratton, E. Scanning FCS, a novel method for three-dimensional particle tracking. Biochemical Society Transactions. 31 (Pt 5), 997-1000 (2003).
- Hinde, E., et al. Quantifying the dynamics of the oligomeric transcription factor STAT3 by pair correlation of molecular brightness. Nature Communications. 7, 11047 (2016).
- Waithe, D., et al. Optimized processing and analysis of conventional confocal microscopy generated scanning FCS data. Methods. 140-141, 62-73 (2018).
- Digman, M. A., Dalal, R., Horwitz, A. F., Gratton, E. Mapping the number of molecules and brightness in the laser scanning microscope. Biophysical Journal. 94 (6), 2320-2332 (2008).
- Ossato, G., et al. A two-step path to inclusion formation of huntingtin peptides revealed by number and brightness analysis. Biophysical Journal. 98 (12), 3078-3085 (2010).
- Nagy, P., Claus, J., Jovin, T. M., Arndt-Jovin, D. J. Distribution of resting and ligand-bound ErbB1 and ErbB2 receptor tyrosine kinases in living cells using number and brightness analysis. Proceedings of the National Academy of Sciences of the United States of America. 107 (38), 16524-16529 (2010).
- Ming, A. Y., et al. Dynamics and Distribution of Klothobeta (KLB) and fibroblast growth factor receptor-1 (FGFR1) in living cells reveal the fibroblast growth factor-21 (FGF21)-induced receptor complex. Journal of Biological Chemistry. 287 (24), 19997-20006 (2012).
- Ross, J. A., et al. Oligomerization state of dynamin 2 in cell membranes using TIRF and number and brightness analysis. Biophysical Journal. 100 (3), L15-L17 (2011).
- Hellriegel, C., Caiolfa, V. R., Corti, V., Sidenius, N., Zamai, M. Number and brightness image analysis reveals ATF-induced dimerization kinetics of uPAR in the cell membrane. FASEB J. 25 (9), 2883-2897 (2011).
- Zamai, M., et al. Number and brightness analysis reveals that NCAM and FGF2 elicit different assembly and dynamics of FGFR1 in live cells. Journal of Cell Science. 132 (1), (2019).
- Hassler, K., et al. Total internal reflection fluorescence correlation spectroscopy (TIR-FCS) with low background and high count-rate per molecule. Optics Express. 13 (19), 7415-7423 (2005).
- Di Rienzo, C., Gratton, E., Beltram, F., Cardarelli, F. From fast fluorescence imaging to molecular diffusion law on live cell membranes in a commercial microscope. Journal of Visualized Experiments. (92), e51994 (2014).
- Beenken, A., Mohammadi, M. The FGF family: biology, pathophysiology and therapy. Nature Reviews Drug Discovery. 8 (3), 235-253 (2009).
- Joubert, J., Sharma, D. Light microscopy digital imaging. Current Protocols in Cytometry. , (2011).
- Gell, C., Berndt, M., Enderlein, J., Diez, S. TIRF microscopy evanescent field calibration using tilted fluorescent microtubules. Journal of Microscopy. 234 (1), 38-46 (2009).
- Burghardt, T. P. Measuring incidence angle for through-the-objective total internal reflection fluorescence microscopy. Journal of Biomedical Optics. 17 (12), 126007 (2012).
- Trullo, A., Corti, V., Arza, E., Caiolfa, V. R., Zamai, M. Application limits and data correction in number of molecules and brightness analysis. Microscopy Research and Technique. 76 (11), 1135-1146 (2013).
- Caiolfa, V. R., et al. Monomer-dimer dynamics and distribution of GPI-anchored uPAR are determined by cell surface protein assemblies. Journal of Cell Biology. 179 (5), 1067-1082 (2007).
- Campbell, R. E., et al. A monomeric red fluorescent protein. Proceedings of the National Academy of Sciences of the United States of America. 99 (12), 7877-7882 (2002).
- Cutrale, F., et al. Using enhanced number and brightness to measure protein oligomerization dynamics in live cells. Nature Protocols. 14 (2), 616-638 (2019).
- Dunsing, V., Chiantia, S. A Fluorescence Fluctuation Spectroscopy Assay of Protein-Protein Interactions at Cell-Cell Contacts. Journal of Visualized Experiments. (142), (2018).

