数と明るさの分析と組み合わせた全内部反射蛍光顕微鏡による生細胞における細胞表面受容体のオリゴマー化ダイナミクス
Summary
生細胞の形質膜におけるリガンド結合によって誘導されるmEGFPタグ付き受容体オリゴマーの平均オリゴマー状態の測定のためのイメージングアプローチについて説明する。このプロトコルは、数と明るさ(N&B)分析と組み合わせた全内部反射蛍光(TIRF)顕微鏡に基づいています。
Abstract
受容体オリゴマー化の重要性とユビキタスにもかかわらず、クラスタリングイベントを検出し、クラスタリングの程度を測定するための方法はほとんどありません。ここでは、生細胞の膜におけるMEGFPタグ付き受容体ホモ複合体の平均オリゴマー状態を決定するイメージングアプローチについて説明する。このプロトコルは、数と明るさ(N&B)分析と組み合わせた全内部反射蛍光(TIRF)顕微鏡に基づいています。N&Bは、蛍光相関分光法(FCS)および光子計数ヒストグラム(PCH)に似た方法で、照明の中で拡散する蛍光強度の変動の統計的分析に基づいています。観測時間中の体積。特に、N&BはPCHの簡素化であり、オリゴマー混合物中のタンパク質の平均数に関する情報を得る。強度変動振幅は、蛍光色素の分子輝度と照明体積内の蛍光色素の平均数によって記述される。したがって、N&Bは振幅分布の第1と第2のモーメント、すなわち平均強度と分散のみを考慮します。これは、同時に、方法の強さと弱さである。2つの瞬間しか考慮されないため、N&Bは混合物中の未知のオリゴマーのモル分率を決定することはできませんが、混合物の平均オリゴマー化状態を推定するだけです。それにもかかわらず、蛍光強度の時間変動を監視するだけで、生細胞の画像の比較的小さな時系列(他のモーメント法と比較して)にピクセル単位で適用することができます。ピクセルあたりの有効時間を数マイクロ秒に短縮し、高速オリゴマー化動態に必要な秒からミリ秒までの時間範囲での取得を可能にします。最後に、大きな細胞領域と細胞下コンパートメントを探索することができます。
Introduction
我々は、受容体アセンブリの連結を目指し、生細胞の形質膜における受容体分子の平均オリゴマー状態を決定するための全内部反射蛍光数と輝度(TIRF-N&B)イメージングアプローチを記述する。タンパク質の生物学的機能に対するダイナミクス (図 1)
細胞外リガンド結合の際、受容体は、その立体構造、オリゴマー化、潜在的な共受容体および膜組成に応じて細胞内シグナル伝達を開始する。受容体オリゴマー化の重要性とユビキタス性にもかかわらず、細胞シグナル伝達1、2、3、4、5、6の重要な事象として認識される。 7、クラスタリングイベントを検出し、実験的にクラスタリングの程度を測定できる方法はほとんどありません。共焦点体積(x,y≥ 300 nm,z≥900 nm)は、分子相互作用および動物化測定を証明するために十分に解決されていないが、画像復元アルゴリズム10による最適化後も。タンパク質オリゴマーのサブユニット組成は、PALM11、STORM12、STED13などの20〜70nmのX、y分解能法であっても、純粋に空間的に分解することはできません。さらに、時間分解能 (画像あたりの分単位) は、秒単位のキネティクスに従うことはできません。単分子ステップ漂白は、それらが不動である場合にのみタンパク質オリゴマーのムチオメトリーを解決する。
単一画像内の蛍光タグ付きタンパク質の密度とオリゴマー化を測定する最も汎用性の高い方法の1つは、空間サンプリングに依存する空間強度分布解析(SpIDA)です。これは、化学的に固定された細胞と生細胞の両方に適用可能であり、標準蛍光顕微鏡15を用いて細胞の目的のいくつかの領域の分析を同時に可能にする。あるいは、蛍光相関分光法(FCS)16、フォトン計数ヒストグラム(PCH)17、数と明るさ(N&B)18、19などのモーメント法は、定量的オリゴマーに適している測定。これらの方法は、蛍光体が照明体積の間で拡散する時間内に観察できる蛍光強度変動を分析する。強度変動の振幅は、蛍光色素の分子輝度(ε)と照明音量17内の蛍光色素(n)の平均数によって一意に記述することができる(図2)。典型的には、蛍光体の拡散係数および照明容積内の分子の平均数(G(0)値に逆作用する)は、FCS20によって得ることができる。しかし、拡散時間は質量の立方根のみでスケールするので、FCSは分子量21の変化を検出するのに十分な感受性を持たない。実際には、単色FCSは膜受容体の二量体化を検出できない。PCHは異なるオリゴマーの混合物を正確に解決する。振幅分布の2回以上の瞬間を使用して、同じ照明ボリュームを占める異なる明るさの分子を検出します。分子輝度(pCOMB)アプローチ23の興味深い対相関などの走査FCS22および開発は、生物学的システムにおける蛍光相関法の適用範囲を広げるために導入された24、セルの広い領域での高速測定の能力を欠いている単一点法のままであり、各ピクセルで多くの連続した観測値を必要とし、秒単位でデータを取得する必要があります。
N&B は、蛍光分布の振幅の最初と 2 番目のモーメント、すなわち平均強度、、および分散 σ2 (図 2)18、19 のみを考慮する PCH の簡略化されたバージョンです。そして、そのために、混合物中の未知のオリゴマーのモル分率を決定することはできないが、混合物の平均オリゴマー化状態を推定するだけである。それにもかかわらず、N&Bは、蛍光強度の時間の変動を監視するだけで、PCHよりも比較的小さな時系列の生細胞画像をピクセル単位で操作できるという利点があります。N&B はピクセルあたりの時間を数マイクロ秒に短縮するため、大きなセル領域に対する高速オリゴマー化動態に従うことができ、ラスター スキャン顕微鏡 (共焦点、2 光子など) およびミリ秒で数秒のタイム スケールで画像を取得できます。カメラベースの顕微鏡検査(例えば、TIRFM)で。
いくつかの報告は、拡張細胞領域をイメージングすることによってタンパク質クラスター内のサブユニットの数を定量化するN&Bの能力を実証している。Paxillin-EGFPクラスターはCHO-K1細胞25の接着部位で検出され、かつ病原性Httex1pペプチドの細胞内凝集はCOS-7細胞26に記載した。N&Bは、ErbB受容体27のリガンド駆動オリゴマー化に続いて適用し、HeLa細胞28におけるクロトゴブ(KLB)およびFGFR1cに対するリガンドFGF21の効果を適用した。TIRFイメージングとN&B分析の組み合わせは、ダイナミン-2が主に細胞膜29全体にわたって四量体であることを示すために使用された。我々は、uPARおよびFGFR1細胞膜受容体30、31のリガンド駆動二量体化を証明するために、ラスタースキャンとTIRF画像の両方にN&Bを適用した。
N&B、FCS、PCHなどの蛍光相関法は、オープンボリュームでは粒子の職業数がポアソン分布に従るという概念に基づいています。蛍光体が放出するフォトンのみが検出できるので、画像のピクセルにおける測定された蛍光強度と時間の平均値は、 照明体積における蛍光体の平均数の積であり、n、分子輝度, ε17:
照明体積における蛍光体の平均数の積であり、n、分子輝度, ε17:

ここで ε は、分子が照明ボリュームの中心にある場合に、分子あたりの時間単位あたりの光子の数 (従来は 1 秒あたり) として表されます。
明るさは、所定の取得設定における各蛍光色素の特性であり、強度はすべての蛍光色素からのすべての寄与の合計です。生物学的コンテストでは、一緒に変動するフルオロフォアの数の増加に伴って明るさが増加し、蛍光タグ付きタンパク質のオリゴマー化状態に関する情報が与られます。特定のピクセルの変動振幅は、蛍光シグナルσ2の分散から測定されます。

強度の二乗の平均、 および強度の平均の二乗は、
および強度の平均の二乗は、 各フレームの各ピクセルの個々の強度値から計算されます。
各フレームの各ピクセルの個々の強度値から計算されます。
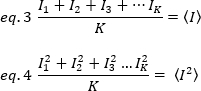
ここで、Kは時系列の合計フレーム数です。実験的には、平均強度値の周囲の単一の画像の各ピクセルにおける個々の強度値の散乱を記述する分散を画像シリーズ全体に対して計算する必要があります。分散には、異なる原点のすべての変動が含まれます。第1近似では、照明体積中の拡散粒子による分散、σ20、検出器ショットノイズによる分散から分離することができる、σ2d。2 つの分散は独立しています。したがって、合計分散は合計で与えられます。

この分散は、検出体積の分子変動に起因して、分子の明るさと強度に直線的に依存します。

eq. 1 に従って eq. 6 を並べ替える:

蛍光相関分光法における典型的な概念によれば、式7は、変動数による分散が粒子輝度の二乗に依存することを示す。
そして、検出器の変動による分散は、検出された強度の線形関数であり、検出器がその飽和限界19以下で動作することを前提とする。

フォトン計数検出器a=1 およびc=0 の場合、検出器の分散は平均強度と等しくなります。

これらの概念を生細胞の実際の測定値に適用するために、Gratton と同僚18は、各ピクセルの見かけの明るさ B を平均強度に対する分散の比率として定義します。

Bは実験的に測定されるパラメータである。本研究では、HeLa細胞の形質膜におけるFGFR1受容体の時系列画像をTIRF顕微鏡で捉え、平均見かけ明るさBをN&B分析により決定する。次いで、FGF2を添加した後、公索リガンドによる受容体の刺激後の膜表面における受容体分子の自己集合の変化に従って連続した時系列が捕捉される。
しかし、TIRF顕微鏡の検出器はEMCCDカメラであるため、見かけの明るさの発現を19として変更する必要があります。

ここで、オフセットは検出器設定の特性である検出エレクトロニクスの強度オフセットです。アナログ検出器の分散と平均強度は、それぞれ次のように求められています。
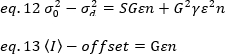
ここで、Gはデジタルレベル(DL/フォトン)のアナログゲインであり、S、フォトン19あたりのデジタルレベルは、一定の強度(時間的変動なし)を持つ光源の強度対分散プロットの傾きによって与えられます。γ因子は、画素検出ボリュームの形状に関連する。Hassler et al.32によれば、γ因子は、検出カメラ19の最大ゲインで動作するTIRFイメージングに対して0.3に等しい。オフセット、SおよびGパラメータは、カメラおよび顕微鏡の特性である。見かけの明るさBは、eq. 12および13に従ってeq. 11を並べ替えることによって得られる。

実験的には、εはレーザー強度とシステムの検出効率の複雑な関数です。それにもかかわらず、B/Sはεに直線的に依存しているため、特定の検出モードのεの相対値を決定することが重要です。

ここでε’は ε に比例します。それでも、キャリブレーションは内部参照を使用して実行されます。
Protocol
Representative Results
Discussion
N&B では、セル モデルとラベリング戦略を選択する際に、いくつかの予防措置が必要です。画像のキャプチャ時間中に安定して付着したままの生細胞にのみ適用できます。セル全体の剛性変位による余分な変動は、適切な画像復元アプローチ38で処理され得る。しかしながら、一般に細胞が移動すると、細胞膜も変形し、構造変形が起き、大きな余分な分散を生じ、膜タンパ…
Offenlegungen
The authors have nothing to disclose.
Acknowledgements
CNICは、シエンシア省、イノバシオン・イ・ウニベルシダーデス、プロCNIC財団の支援を受けており、セベロ・オチョア・センター・オブ・エクセレンス(SEV-2015-0505)です。また、欧州地域開発基金(FEDER)「ウナ・マネラ・デ・ハーサー・エウロパ」の支援を受けています。UCは、アソシアツィオーネ・イタリアン・リケルカ・スル・カンクロ、国際がん研究協会(現在は世界がん研究)、イタリア保健省の支援を認めています。A.T.は、PVフェローシップ「プロゲット・プロイリタ・イヴァーノ・ベッキ」2011-2012で彼の作品を部分的にサポートした「フォンダツィオーネ・バンカ・デル・モンテ・ディ・ロンバルディア」を認めます。
Materials
| 3-Colour Fast TIRF Leica AM TIRF MC inverted microscope, with smi-automatic TIRF alignment. The microscope is equipped with a diode 488 nm laser, a 100×1.46 oil TIRF objective, Ex/Em Bandpass filters at 490/20 and 525/50, temperature/CO2 incubator and Andor DU 8285 VP EMCCD camera. The microscope is operated by Leica LIF software. | Leica Microsystems, Wetzlar, Germany | ||
| Albumin from Bovine Serum 98% minimun | Sigma-Aldrich, St. Louis, MI, USA | A7906-100G | |
| DMEM without Phenol Red with 25 mM HEPES | GIBCO Thermo Fisher Scientific,Waltham, MA, USA | 21063029 | Used serum free for microscopy |
| DMEM high-glucose GlutaMAX I | GIBCO Thermo Fisher Scientific,Waltham, MA, USA | 10566-016 | Used for complete medium |
| Dulbecco's Phosphate Buffered Saline 10x (PBS) | Biowest, Nuaillé, France | X0515-500 | |
| Emission splitting system Photometrics DV2 | TeledynePhotometrics, Tucson, AZ, USA | ||
| Fetal Bovine Serum, qualified, Brazil | GIBCO Thermo Fisher Scientific,Waltham, MA, USA | 10270106 | 10% inactivated supplement for complete medium |
| Glass bottom 35-mm sterile 1.5 dishes | MatTek, Ashland, MA, USA | P35G-0.170-14-C | uncoated, glass thickness 0.17 microns |
| GraphPad Prism | GraphPad Software Inc., San Diego, CA, USA | ||
| Human cervical carcinoma (HeLa), serum-free animal component (AC) cells | Millipore-Sigma ECACC, Darmstadt, Germany | CB_08011102 | |
| iXonEM+ 897 EMCCD (back-illuminated) ANDOR camera controlled by ANDOR Solis software | Oxford Instruments, Andor TM Technology, Abingdon-on-Thames, UK | This camera, installed in an additional port of the microscope, is used for acquiring the N&B time series | |
| Matlab Executable N&B routine | Unit of Microscopy and Dynamic Imaging, CNIC, Madrid, Spain | download at https://www.cnic.es/en/investigacion/2/1187/tecnologia | |
| MatLab v.2018b | The MathWorks, Inc. Natick, MA, USA | download at https://www.mathworks.com/products/matlab.html | |
| Penicillin:Streptomycin for tissue culture 100x | Biowhittaker Inc. Walkersville, MD, USA | LONZA 17-602E | supplement for medium at Penicillin/Streptomycin 100U/100µg. |
| pN1-mEGFP-FGFR1 expression vector | Unit of Gynecological Oncology Research, European Institute of Oncology IRCCS, Milan, Italy | Zamai et al., 2019 | |
| pN1-N-Gly-mEGFP-GPI expression vector | Unit of Microscopy and Dynamic Imaging, CNIC, Madrid, Spain | Hellriegel et al., 2011 | |
| pN1-N-Gly-mEGFP-mEGFP-GPI expression vector | Unit of Microscopy and Dynamic Imaging, CNIC, Madrid, Spain | Hellriegel et al., 2011 | |
| Recombinant FGF2 | PeproTech EC, Ltd., London, UK | Ligand solution: 20ng/mL of FGF2 in PBS supplemented with 0.01%BSA. | |
| Sodium pyruvate GIBCO | ThermoFisher Scientific | 11360070 | 1mM supplement for medium |
| TransIt-LT1 Transfection Reagent | MirusBio LLC, Madison, WI, USA | MIR 2300 | |
| Trypsin-EDTA (0.25%), phenol red | GIBCO Thermo Fisher Scientific,Waltham, MA, USA | 25200056 | |
| Type F Immersion liquid 10 mL | Leica Microsystems, Wetzlar, Germany | 11513 859 | |
| UltraPure BSA (50 mg/mL) | ThermoFisher Scientific | AM2618 | 0.1% supplement for medium without phenol red used for transfections |
Referenzen
- Agwuegbo, U. C., Jonas, K. C. Molecular and functional insights into gonadotropin hormone receptor dimerization and oligomerization. Minerva Ginecologica. 70 (5), 539-548 (2018).
- Ferre, S., et al. G protein-coupled receptor oligomerization revisited: functional and pharmacological perspectives. Pharmacological Reviews. 66 (2), 413-434 (2014).
- Marsango, S., Ward, R. J., Alvarez-Curto, E., Milligan, G. Muscarinic receptor oligomerization. Neuropharmacology. 136 (Pt C), 401-410 (2018).
- Oishi, A., Cecon, E., Jockers, R. Melatonin Receptor Signaling: Impact of Receptor Oligomerization on Receptor Function. International Review of Cell and Molecular Biology. 338, 59-77 (2018).
- Thelen, M., Munoz, L. M., Rodriguez-Frade, J. M., Mellado, M. Chemokine receptor oligomerization: functional considerations. Current Opinion in Pharmacology. 10 (1), 38-43 (2010).
- Van Craenenbroeck, K. GPCR oligomerization: contribution to receptor biogenesis. Subcellular Biochemistry. 63, 43-65 (2012).
- Wnorowski, A., Jozwiak, K. Homo- and hetero-oligomerization of beta2-adrenergic receptor in receptor trafficking, signaling pathways and receptor pharmacology. Cell Signaling Technology. 26 (10), 2259-2265 (2014).
- Fricke, F., Dietz, M. S., Heilemann, M. Single-molecule methods to study membrane receptor oligomerization. Chemphyschem. 16 (4), 713-721 (2015).
- Vidi, P. A., Ejendal, K. F., Przybyla, J. A., Watts, V. J. Fluorescent protein complementation assays: new tools to study G protein-coupled receptor oligomerization and GPCR-mediated signaling. Molecular and Cellular Endocrinology. 331 (2), 185-193 (2011).
- Trussell, H. J., et al., Trussell, J., et al. . Academic Press Library in Signal Processing. 4, 3-9 (2014).
- Betzig, E., et al. Imaging intracellular fluorescent proteins at nanometer resolution. Science. 313 (5793), 1642-1645 (2006).
- Rust, M. J., Bates, M., Zhuang, X. Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy (STORM). Nature Methods. 3 (10), 793-795 (2006).
- Nagerl, U. V., Willig, K. I., Hein, B., Hell, S. W., Bonhoeffer, T. Live-cell imaging of dendritic spines by STED microscopy. Proceedings of the National Academy of Sciences of the United States of America. 105 (48), 18982-18987 (2008).
- Tsekouras, K., Custer, T. C., Jashnsaz, H., Walter, N. G., Presse, S. A novel method to accurately locate and count large numbers of steps by photobleaching. Molecular Biology of the Cell. 27 (22), 3601-3615 (2016).
- Godin, A. G., et al. Revealing protein oligomerization and densities in situ using spatial intensity distribution analysis. Proceedings of the National Academy of Sciences of the United States of America. 108 (17), 7010-7015 (2011).
- Qian, H., Elson, E. L. Distribution of molecular aggregation by analysis of fluctuation moments. Proceedings of the National Academy of Sciences of the United States of America. 87 (14), 5479-5483 (1990).
- Chen, Y., Muller, J. D., So, P. T., Gratton, E. The photon counting histogram in fluorescence fluctuation spectroscopy. Biophysical Journal. 77 (1), 553-567 (1999).
- Dalal, R. B., Digman, M. A., Horwitz, A. F., Vetri, V., Gratton, E. Determination of particle number and brightness using a laser scanning confocal microscope operating in the analog mode. Microscopy Research and Technique. 71 (1), 69-81 (2008).
- Unruh, J. R., Gratton, E. Analysis of molecular concentration and brightness from fluorescence fluctuation data with an electron multiplied CCD camera. Biophysical Journal. 95 (11), 5385-5398 (2008).
- Hess, S. T., Huang, S., Heikal, A. A., Webb, W. W. Biological and chemical applications of fluorescence correlation spectroscopy: a review. Biochemie. 41 (3), 697-705 (2002).
- Muller, J. D., Chen, Y., Gratton, E. Fluorescence correlation spectroscopy. Methods in Enzymology. 361, 69-92 (2003).
- Levi, V., Ruan, Q., Kis-Petikova, K., Gratton, E. Scanning FCS, a novel method for three-dimensional particle tracking. Biochemical Society Transactions. 31 (Pt 5), 997-1000 (2003).
- Hinde, E., et al. Quantifying the dynamics of the oligomeric transcription factor STAT3 by pair correlation of molecular brightness. Nature Communications. 7, 11047 (2016).
- Waithe, D., et al. Optimized processing and analysis of conventional confocal microscopy generated scanning FCS data. Methods. 140-141, 62-73 (2018).
- Digman, M. A., Dalal, R., Horwitz, A. F., Gratton, E. Mapping the number of molecules and brightness in the laser scanning microscope. Biophysical Journal. 94 (6), 2320-2332 (2008).
- Ossato, G., et al. A two-step path to inclusion formation of huntingtin peptides revealed by number and brightness analysis. Biophysical Journal. 98 (12), 3078-3085 (2010).
- Nagy, P., Claus, J., Jovin, T. M., Arndt-Jovin, D. J. Distribution of resting and ligand-bound ErbB1 and ErbB2 receptor tyrosine kinases in living cells using number and brightness analysis. Proceedings of the National Academy of Sciences of the United States of America. 107 (38), 16524-16529 (2010).
- Ming, A. Y., et al. Dynamics and Distribution of Klothobeta (KLB) and fibroblast growth factor receptor-1 (FGFR1) in living cells reveal the fibroblast growth factor-21 (FGF21)-induced receptor complex. Journal of Biological Chemistry. 287 (24), 19997-20006 (2012).
- Ross, J. A., et al. Oligomerization state of dynamin 2 in cell membranes using TIRF and number and brightness analysis. Biophysical Journal. 100 (3), L15-L17 (2011).
- Hellriegel, C., Caiolfa, V. R., Corti, V., Sidenius, N., Zamai, M. Number and brightness image analysis reveals ATF-induced dimerization kinetics of uPAR in the cell membrane. FASEB J. 25 (9), 2883-2897 (2011).
- Zamai, M., et al. Number and brightness analysis reveals that NCAM and FGF2 elicit different assembly and dynamics of FGFR1 in live cells. Journal of Cell Science. 132 (1), (2019).
- Hassler, K., et al. Total internal reflection fluorescence correlation spectroscopy (TIR-FCS) with low background and high count-rate per molecule. Optics Express. 13 (19), 7415-7423 (2005).
- Di Rienzo, C., Gratton, E., Beltram, F., Cardarelli, F. From fast fluorescence imaging to molecular diffusion law on live cell membranes in a commercial microscope. Journal of Visualized Experiments. (92), e51994 (2014).
- Beenken, A., Mohammadi, M. The FGF family: biology, pathophysiology and therapy. Nature Reviews Drug Discovery. 8 (3), 235-253 (2009).
- Joubert, J., Sharma, D. Light microscopy digital imaging. Current Protocols in Cytometry. , (2011).
- Gell, C., Berndt, M., Enderlein, J., Diez, S. TIRF microscopy evanescent field calibration using tilted fluorescent microtubules. Journal of Microscopy. 234 (1), 38-46 (2009).
- Burghardt, T. P. Measuring incidence angle for through-the-objective total internal reflection fluorescence microscopy. Journal of Biomedical Optics. 17 (12), 126007 (2012).
- Trullo, A., Corti, V., Arza, E., Caiolfa, V. R., Zamai, M. Application limits and data correction in number of molecules and brightness analysis. Microscopy Research and Technique. 76 (11), 1135-1146 (2013).
- Caiolfa, V. R., et al. Monomer-dimer dynamics and distribution of GPI-anchored uPAR are determined by cell surface protein assemblies. Journal of Cell Biology. 179 (5), 1067-1082 (2007).
- Campbell, R. E., et al. A monomeric red fluorescent protein. Proceedings of the National Academy of Sciences of the United States of America. 99 (12), 7877-7882 (2002).
- Cutrale, F., et al. Using enhanced number and brightness to measure protein oligomerization dynamics in live cells. Nature Protocols. 14 (2), 616-638 (2019).
- Dunsing, V., Chiantia, S. A Fluorescence Fluctuation Spectroscopy Assay of Protein-Protein Interactions at Cell-Cell Contacts. Journal of Visualized Experiments. (142), (2018).

