Visualisierung von Lignification Dynamik in Anlagen mit Klicken Sie auf Chemie: doppelte Kennzeichnung ist Glückseligkeit!
Summary
BLISS, ein dual Kennzeichnung Protokoll zur Untersuchung Lignification Dynamik, wurde entwickelt. Mit synthetischen Monolignol klicken Sie Reporter und eine sequentielle Kombination aus SPAAC und CuAAC Bioorthogonal Reaktionen, diese Methode ebnet den Weg für eingehende Analyse der Faktoren, die die Biogenese der Lignine in Plantaregulieren.
Abstract
Lignin ist eines der am weitesten verbreiteten Biopolymere auf dem Planeten und ein wichtiger Bestandteil von Lignozellulose Biomasse. Dieser phenolischen Polymeren spielt eine wichtige strukturelle und schützende Rolle in die Entwicklung und das Leben der höheren Pflanzen. Obwohl die komplizierten Mechanismen Lignification Prozesse in Vivo stark die industrielle Verwertung vieler pflanzlicher Produkte auswirken, hat die wissenschaftliche Gemeinschaft noch einen langen Weg zu gehen, um sie zu entziffern. In einem einfachen drei-Stufen-Workflow ermöglicht das doppelte Kennzeichnung Protokoll enthaltenen Bioimaging Studien aktiv verholzenden Zonen von Pflanzengewebe. Der erste Schritt besteht in der metabolischen Gründung von zwei unabhängigen chemischen Reporter, Surrogate von zwei einheimischen Monolignols, die Anlass zu Lignin H und G-Einheiten. Nach Einarbeitung in die wachsenden Lignin Polymere ist jeder Reporter dann speziell mit eigenem fluoreszierende Sonde über eine sequentielle Kombination von Bioorthogonal SPAAC/CuAAC klicken Sie auf Reaktionen gekennzeichnet. In Kombination mit Lignin Autofluoreszenz, dieser Ansatz führt zu der Generation von dreifarbigen Lokalisierung Karten von Lignin in pflanzlichen Zellwänden durch konfokale Fluoreszenzmikroskopie und präzise räumliche informiert über das Vorhandensein oder Fehlen von active Lignification Maschinen auf der Skala des pflanzlichen Geweben, Zellen und verschiedenen Zellwand Schichten.
Introduction
In den letzten zwei Jahrzehnten entstanden die Chemie Reporter-Strategie als eine leistungsstarke zweistufige Methodik, die Dynamik und die Funktionen der nicht genetisch codierte Biomoleküle zu untersuchen. 1 , 2 , 3 in dieser Strategie ein synthetisches Analogon das Biomolekül von Interesse mit einer kleinen Modulation – der Chemie Reporter – ist zunächst metabolisiert des lebenden Organismus, und dann eine chemische Sonde (zB., ein Fluorophor für Fluoreszenz konfokale Mikroskopie-Tomographie) ist der eingebaute Reporter über Bioorthogonal Click Chemie kovalent verbunden. Die Sonde muss rasch und gezielt reagieren mit der eingeführten chemische Modifikation während inert gegenüber jedem Biomoleküle im lebenden System vorhanden. Diese Methode überwindet in vielerlei Hinsicht die Grenzen der gemeinsamen Biokonjugaten Techniken durch den Einsatz von hochspezifischen Klick Chemie man damit die Möglichkeit zu verfolgen, Metaboliten oder Biomakromoleküle, die zuvor nicht zugänglich waren in lebenden Systemen4,5,6.
Trotz der schnell wachsenden Popularität dieser mächtigen Methode in bakterielle und tierischen Zellen sind Berichte über den Einsatz in Pflanzenbiologie überraschend wenige und weit zwischen7,8,9,10, 11,12. Wir interessierten uns vor allem bei der Anwendung dieser Strategie in Pflanzen, die Bildung von Lignin, eines der am weitesten verbreiteten Biopolymere auf dem Planeten und ein wichtiger Bestandteil von Lignozellulose Biomasse zu studieren. 13 , 14 Lignin ist ein phenolischen Polymer, das spielt eine wichtige strukturelle und schützende Rolle in der Entwicklung und Leben der höheren Pflanzen.
Es besteht im Allgemeinen aus drei 4-Hydroxyphenylpropanoid Moieties: H (p– Hydroxyphenyl), G (Guaiacyl) und S (Syringyl) Einheiten bzw. abgeleitet von drei “Monolignols” (p– Coumaryl, Coniferyl und Sinapyl Alkohole) sind über den Phenylpropanoid Weg im Zytoplasma der Zelle (Abbildung 1) synthetisiert. Nach dem Export in der Zellwand, Monolignols oxidiert sind radikale durch Peroxidasen oder Laccases nach dem durchlaufen sie rein chemischen radikalen Kupplungsreaktionen Lignin Polymere polymerisieren, bezeichnet ein Prozess Lignification. 15 , 16 obwohl Lignine stark beeinflussen die industrielle Verwertung vieler pflanzlicher Produkte, die wissenschaftliche Gemeinschaft hat noch einen langen Weg zu gehen, um die komplizierten Mechanismen Lignification zu entschlüsseln.
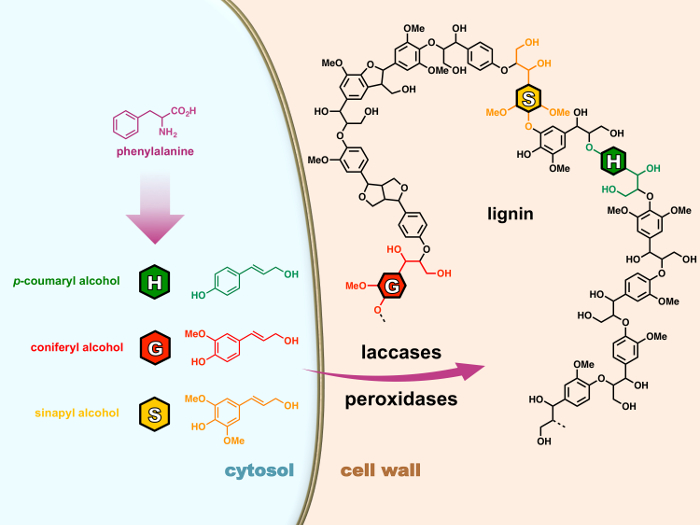
Abbildung 1: der Lignification-Prozess in Pflanzenzellen. Monolignols sind Biosynthesized aus Phenylalanin in der Zellflüssigkeit. Nach dem Export in der Zellwand, Monolignols oxidiert sind radikale durch Peroxidasen oder Laccases nach dem durchlaufen sie rein chemischen radikalen Kupplungsreaktionen Lignin Polymere polymerisieren, bezeichnet ein Prozess Lignification. Bitte klicken Sie hier für eine größere Version dieser Figur.
Obwohl Berichte über den Einsatz von Bioorthogonal Reaktionen Glycan Analyse zahlreich sind, sind2,3,17 ihre Anwendungsbeispiele auf andere Arten von Biomolekülen weniger. Die Verwendung der Bioorthogonal Chemie für Lignin Bioimaging Zwecke wurde erst vor kurzem erstmals von Tobimatsu Et al. 8 di Arabidopsis Thaliana über die Einbeziehung von Coniferyl Alkohol Surrogate in das Polymer Lignin informieren wo es bildet die G-Einheiten,8,9 so demonstriert die Machbarkeitsstudie, die Chemie Reporter Strategien gelten in diesem Zusammenhang. Die Verwendung von CuAAC wurde auch anhand eine unterschiedliche Coniferyl-Alkohol-Derivat ein paar Monate später von Bukowski Et Al. 9 jedoch Lignin enthält auch H und S Einheiten und ein tieferes Verständnis des Lignification Prozesses erfordert mehr wissen über wie alle die Monolignols in das Polymer einbezogen werden und welche Faktoren kann Einfluss auf seine Zusammensetzung. Neue Fortschritte in diesem Bereich ist heute abhängig von der Entwicklung von effizienten Methoden mehrere chemische Reporter in lebenden Systemen gleichzeitig verfolgen. Obwohl ein paar Artikel über Glykoproteinen in den letzten Jahren18,19,die Grundlagen haben bleibt20,21,22, doppelte Kennzeichnung Ansätze eine große Herausforderung dar Bioorthogonal Chemie. Wenn eine reproduzierbare Single-Beschriftung klicken Sie auf Protokoll ist schwer zu entwickeln, dann doppelte Kennzeichnung Ansätze, die die Optimierung im Tandem von zwei sich gegenseitig erfordern sind kompatibel Bioorthogonal Reaktionen auf zwei separaten chemischen Reporter noch schwieriger. Die wenigen Beispiele, die diesen Aspekt Pionier verwendet eine Kombination von Stamm-gefördert-Azid-Alkinen Cycloaddition (SPAAC) und Alken-kurz inverse elektronische Nachfrage Diels-Alder (DAInv) Reaktionen Glykoproteinen in tierischen Zellen zu studieren. Allerdings dachten wir, dass die Bioorthogonality der DAinv Reaktion nicht in dieser Anwendung aufgrund der strukturellen Merkmale des Lignins garantiert werden könnte (bestehend aus elektronenreichen substituierte Cinnamyl-Art Monomere, die mit Elektron-Armen reagieren können Dienes wie die kurz-Sonden verwendet im DAinv Reaktionen) und dass das unspezifische Kennzeichnung erzeugen kann. Darüber hinaus erfordert die DAInv Reaktion chemische Griffe, die synthetisch schwer zu Zugang, wie auch als sperrig und lipophilen erhöhen dadurch die Möglichkeit, dass die Rate der Aufnahme, Transport und/oder Lokalisierung der chemischen Industrie Reporter in Vivo können betroffen sein. Gelten wir als sei Letzteres besonders relevant bei einem Klick Chemie Ansatz für das Studium Lignification, wir entschieden eine andere Richtung und entwickelt eine Bioorthogonal Ligation Imaging sequentielle Strategie (Glückseligkeit) mit einem Kombination von Strain-Promoted Azid-Alkinen Cycloaddition (SPAAC) und Kupfer katalysierte Azid-Alkinen Cycloaddition (CuAAC) in-vivo. 23
Diese zwei Reaktionen sind in der Tat die zwei wichtigsten Bioorthogonal klicken Sie auf Reaktionen, die bisher verwendet wurden, und vor allem in der wenigen Beispiele des Lignins imaging, die wurden vor kurzem veröffentlicht. 8 , 9 unsere Doppelstrategie Kennzeichnung ermöglicht den Einsatz einer Azid glyko-auf einem Monolignol Reporter und ein terminal Alkinen andererseits zwei chemischen Griffe, die (i) Plattierung in Richtung biologisch relevante Strukturen und (Ii) sehr klein (Abbildung 2 ). Infolgedessen wird die Auswirkungen dieser synthetischen Modifikationen auf die physikalisch-chemischen Eigenschaften der das Biomolekül untersuchten minimiert, wodurch mögliche Diskrepanzen zwischen den natürlichen und unnatürlichen Monolignol Substraten in den Bereichen Verkehr und Metabolization Preise während der metabolische Aufnahme Schritt. Obwohl die Kombination von SPAAC und CuAAC auf den ersten Blick sehr intuitiv scheint, ist es nach unserer Kenntnis nur das zweite Beispiel doppelte Kennzeichnung mit dieser Strategie und die erste Anwendung auf Strukturen als Glykoproteinen. 12 , 23
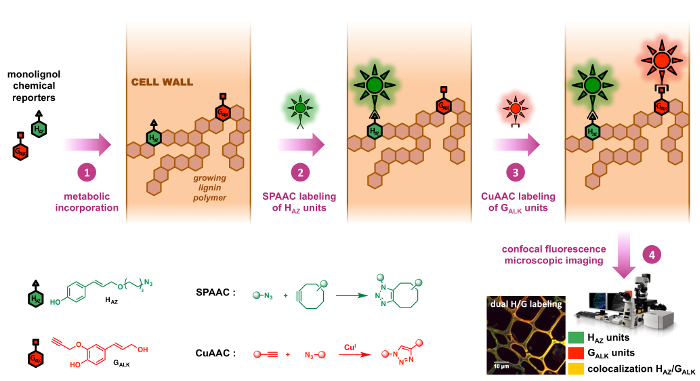
Abbildung 2: BLISS doppelte Kennzeichnung Strategie. Tagged Analoga von native H und G Monolignols sind chemische Reporter HAZ und GALK . Sie fließen zunächst in die wachsende Lignin Polymere der Zellwände durch exogene Fütterung (Schritt 1). Cyclooctyne – und Azid-funktionalisiert fluoreszierende Sonden werden dann nacheinander auf die eingebaute Reporter durch Bioorthogonal ligiert Chemie klicken: die SPAAC Reaktion (Schritt 2) ist hochspezifisch HAZ Einheiten und ist gefolgt von einer CuAAC Reaktion ( Schritt 3), das ist spezifisch GALK Einheiten (Schritt 3), so dass die konkrete Lokalisierung von beiden Reportern unabhängig voneinander in der gleichen Probe. Bitte klicken Sie hier für eine größere Version dieser Figur.
Wir erstens entwickelt und validiert das Azid-Tags Monolignol Reporter HAZ (Ersatz von p– Coumaryl Spiritus) und Vorläufer des Lignins H Einheiten und dann entwickelt der Glückseligkeit Kennzeichnung Doppelstrategie in dem dient es zusammen mit der zuvor berichtete Alkinen getaggt GALK,9 (Ersatz von Coniferyl Alkohol) und Vorläufer des Lignins G Einheiten. In diesem reproduzierbare Protokoll entwickelt und getestet in Flachs ist eine wirtschaftlich bedeutende Pflanzenarten, die dual metabolische Einbindung der HAZ und GALK in Lignin zuerst vor dem sequentiellen SPAAC/CuAAC erreicht. Kennzeichnung. Hier sind tagged HAZ Einheiten zunächst speziell über die SPAAC Ligatur der ein Cyclooctyne funktionalisiert Fluorophor, gefolgt von CuAAC-vermittelten Ligatur von zweiten fluoreszierende Sonden auf tagged GALK gekennzeichnet Einheiten. Diese Methode kann wurde verwendet, um die Dynamik von Lignification Prozessen in pflanzlichen Zellwänden zu untersuchen und angewandte in Vivo , Querschnitte, lebenden Stämme sowie Sämlinge von verschiedenen Pflanzenarten einzudämmen.
Protocol
Representative Results
Discussion
Wie bereits erwähnt ist das duale Kennzeichnung BLISS Protokoll in diesem Papier präsentiert eines der ersten Beispiele für eine SPAAC/CuAAC Kombination in Vivo12,23. Jeder Schritt wurde gründlich optimiert und validiert, und es ist sehr wichtig, dass die Reihenfolge, in der die beiden klicken Chemie Kennzeichnung Reaktionen werden sequentiell durchgeführt, eingehalten wird (d. h. SPAAC zuerst, gefolgt von CuAAC). Alle Kreuz-Kontrollen haben gezeigt, dass jede Kennzeichnung Schritt spezifisch ist, wenn das BLISS-Protokoll angewandte23 ist : Durchführung der SPAAC Schritt führt zunächst zu hoch chemische Kennzeichnung HAZ -Azid Funktionen durch die Cyclooctyne funktionalisiert Fluorophor durch Reaktion mit schnellen Kinetik [3 + 2] Cycloaddition. Nachdem HAZ Einheiten markiert sind, kann der CuAAC Schritt erfordern copper(I) katalysiert Aktivierung von GALK terminal Alkinen Generierung von Triazol Links durch Reaktion mit der Azid-Fluor-545-Sonde erfolgen. Im Gegensatz dazu der umgekehrten Reihenfolge (d. h. CuAAC zuerst, gefolgt von SPAAC) sollte nicht verwendet werden, da sie zu GALK und HAZ Einheit führt Kreuz-Kupplung, die konkurriert mit Fluorophor Ligatur und führt zu einen dramatischen Verlust des Signals . Es ist auch wichtig zu betonen die Notwendigkeit, die zwischengeschalteten Waschschritten unspezifische Färbung zu vermeiden.
Wir haben gezeigt, dass unsere Methode auf verschiedenen biologischen Experiment Designs angewendet werden kann. Die Glückseligkeit Kennzeichnung Protokoll war zunächst auf Freihand Querschnitte der Flachs Stämme (ca. 150-250 µm dick) aufgetragen, die zuvor schneiden und mit der Klick-Ready Monolignols inkubiert wurden. Obwohl diese Konstruktion den Vorteil, die benötigten Mengen an Chemie Reporter hat (wie Inkubation Volumen reduziert werden) zu minimieren und die Produktion von statistischen Wiederholungen zu erleichtern, ist es nicht, streng genommen, eine in Vivo -System und in einigen Fällen können nicht alle Aspekte der real räumlich-zeitliche Lignification Dynamik wider. In einer zweiten experimentellen Design passten wir daher das BLISS-Protokoll an eine Methode, die zuvor verwendet wurde, um die Aufnahme der radioaktiven Monolignols im Kiefer und Gingko27zu studieren. Bei diesem Ansatz die Wurzeln und Stängel der Pflanze sind räumlich getrennt und die Basis des ganzen Stammes wird in der Monolignol Lösung in was “Blumenvase” Ansatz genannt werden kann inkubiert. Nach dem Ausscheiden aus den Stielen der gewünschten (Inkubationszeit), werden Querschnitte geschnitten und das BLISS-Protokoll durchgeführt. Dies erlaubt uns zu zeigen, (i) dass die geänderten Monolignols durch den lebenden Stamm transportiert werden und fließen in den wachsenden Polymere Lignin in den Zellwänden und (Ii), dass die Lokalisierung Muster im Wesentlichen identisch mit dem des Querschnitts Ansatz. Diese Art von Experiment hat das Verdienst, in eine echte lebende Pflanze durchgeführt / live Zelle nähern, so dass mehr Experimente und mehr eingehende Studien, erfordert aber größere Mengen an Chemie Reporter. Schließlich diente das BLISS-Protokoll auch mit Flachs-Pflanze-Sämlinge, vertritt eine echte Streckenmodell, in denen der Chemie Reporter durch die Wurzeln aufgenommen werden müssen vor dem Transport, des Stamms, lebt. Während dieses Modell hat den klaren Vorteil in lebenden Pflanzen, in der Praxis durchgeführt wird es beschränkt sich auf Jungpflanzen und eignet sich nicht wirklich für die Untersuchung von Lignification Dynamik in Altanlagen aus praktischen Gründen (lange Inkubationszeit, erhöhte Menge der Chemie Reporter). Dennoch, diese drei Experiment Designs ergänzen und alle haben ihre vor- und Nachteile im Hinblick auf die praktischen Aspekte und biologische Bedeutung abhängig von der biologischen Frage beantwortet werden.
Entwickelt, um die Lignification Dynamik im Flachs zu studieren, unser Protokoll ist sehr anpassungsfähig, nicht nur in Bezug auf biologische Experiment Design, sondern auch im Hinblick auf seine Anwendung auf andere Pflanzen Arten und Organe/Gewebe. BLISS kann beispielsweise leicht übertragen werden, der Arabidopsis oder die Populus-Gattungen, die besser geeignet, um Studien mit Knock Out “oder” Knock-Down durch Mutation entstehende Variationen für verschiedene Gene sind. Im Prinzip können doppelte Kennzeichnung Studien mit unserem Ansatz auch auf andere Biomoleküle erweitert werden mithilfe von zwei verschiedene modifizierten Vorstufen der pflanzlichen Zellwand Polymere – einschließlich aller drei wichtigsten Monolignols oder ihre metabolische Vorläufer sowie diverse Monosaccharide, die die Polysaccharid-Matrix darstellen. Seit seiner Gründung wurde Bioorthogonal Chemie in der Tat hauptsächlich entwickelt, um Glykane/Polysaccharide durch metabolische Oligosaccharid engineering (MOE)4,5,17,28zu untersuchen, aber überraschenderweise gab es nur sehr wenige Anwendungen, Biologie Pflanzen bisher7,8,9,10,11,12. Im Hinblick auf die Kompatibilität der Reaktionen war die Studie von Lignin in der Tat einem komplexen Fall zu lösen, da beide chemischen Reporter in der gleichen netzförmigen Polymer eingearbeitet werden. Die Möglichkeit der unbeschrifteten HAZ– warGALK Querverbindung entsteht Bildung das Hauptproblem, aufgrund der räumlichen Nähe der GALK und HAZ Einheiten innerhalb der 3D zu überwinden Struktur von Lignin23, eine Einschränkung, die möglicherweise nicht vorhanden, wenn die beiden chemischen Reporter nicht einbezogen werden in die gleiche Art von Biopolymer oder in der gleichen räumlichen Region einer bestimmten Zelle.
In größerem Umfang konnte die BLISS-Methodik im Wesentlichen auf eine Zweifarben-Fluoreszenz imaging Studie in Bakterien oder tierischen Modellen, die mit zwei unterschiedlichen chemischen Reporter trägt ein Azid und terminal Alkinen Tag, bzw. angewendet werden.
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
Wir sind verpflichtet, die Forschung Verband FRABio und die TisBio imaging Plattform (Univ. Lille, CNRS, FR 3688, FRABio, BiochimieStructurale et Fonctionnelle des Assemblagen Biomoléculaires) für die Bereitstellung der technischen Rahmenbedingungen zur Erreichung dieses Ziels arbeiten.
Materials
| (E)-4-(3-(2-(2-(2-azidoethoxy)ethoxy)ethoxy)prop-1-en-1-yl)phenol (HAZ) | Synthesized as in Lion et al. Cell Chem. Biol. 2017, 24, 3, 326-338 | ||
| (E)-4-hydroxy-3-propargyloxycinnamyl alcohol (GALK) | Synthesized as in Lion et al. Cell Chem. Biol. 2017, 24, 3, 326-338 | ||
| 2% sodium hypochlorite | |||
| 20 cm high glass tube | |||
| 250 mL Schott glass bottle | |||
| 48-well Plate | |||
| 5/6-TAMRA-PEG3-Azide | Jena Bioscience | CLK-AZ109-1 | |
| Aluminium foil | |||
| Cheese cloth | |||
| Compost containing clay | |||
| Coniferyl alcohol (G) | Sigma Aldrich | MFCD00002922 | |
| Copper (II) sulfate pentahydrate | |||
| DBCO-PEG4-5/6-Carboxyrhodamine 110 | Jena Bioscience | CLK-A127-1 | |
| Milli-Q Ultrapure water | |||
| Eppendorf 1,5 mL | |||
| EtOH | |||
| Flax seeds (L. usitatissimum L.) | |||
| Fluoromount-G™ Slide Mounting Medium | Electron Microscopy Sciences | 17984-25 | |
| Glass coverslip | |||
| Glass microscope slide | |||
| Growth chamber | CLF-Plant Climatics | For 2-week-old plants culture | |
| Growth chamber | Angelantoni Life Sciences | For 2-month-old plants culture | |
| Magenta plant culture box | For 2-week-old seedling culture | ||
| Methanol | Toxic (SGH02, SGH06, SGH08), work with gloves under a hood | ||
| Micropipette | |||
| Nail polish | |||
| Nikon A1R confocal microscope | Nikon | ||
| Orbital shaker | |||
| Parafilm | |||
| p-Coumaryl alcohol (H) | Carbosynth | FC145653 | |
| Plastic cap | |||
| Plastic pipette | |||
| Plastic pot | For 2-month-old plants culture | ||
| Razor blade | |||
| Rubber band | |||
| Sodium Ascorbate | |||
| Sterile clamp | |||
| Vertical support | |||
| Vortex | |||
| Reagents for liquid and solid ½ MS medium | |||
| KH2PO4 | |||
| KNO3 | |||
| NH4NO3 | |||
| MgSO4.7H2O | |||
| CaCl2.2H2O | |||
| MnSO4.H2O | |||
| ZnSO4.7H2O | |||
| H3BO3 | |||
| KI | |||
| Na2MoO4.2H2O | |||
| CuSO4.5H2O | |||
| CoCl2.6H2O | |||
| Na2EDTA.2H2O | |||
| FeSO4.7H2O | |||
| Thiamine.HCl | |||
| Pyridoxine.HCl | |||
| Glycine | |||
| Nicotinic acid | |||
| Myo-inositol | |||
| Saccharose | |||
| MES hydrate | |||
| Agar |
Riferimenti
- Grammel, M., Hang, H. C. Chemical reporters for biological discovery. Nat Chem Biol. 9 (8), 475-484 (2013).
- Prescher, J. A., Bertozzi, C. R. Chemistry in living systems. Nat Chem Biol. 1 (1), 13-21 (2005).
- Sletten, E. M., Bertozzi, C. R. Bioorthogonal chemistry: fishing for selectivity in a sea of functionality. Ange Chemie (Int Ed. Engl). 48 (38), 6974-6998 (2009).
- Chang, P. V., et al. Metabolic labeling of sialic acids in living animals with alkynyl sugars. Ange Chemie (Int Ed. Engl). 48 (22), 4030-4033 (2009).
- Gilormini, P. A., et al. A sequential bioorthogonal dual strategy: ManNAl and SiaNAl as distinct tools to unravel sialic acid metabolic pathways. Chem. Commun. 52 (11), 2318-2321 (2016).
- Mbua, N. E., et al. Abnormal accumulation and recycling of glycoproteins visualized in Niemann-Pick type C cells using the chemical reporter strategy. Proc Natl Acad Sci U S A. 110 (25), 10207-10212 (2013).
- Anderson, C. T., Wallace, I. S., Somerville, C. R. Metabolic click-labeling with a fucose analog reveals pectin delivery, architecture, and dynamics in Arabidopsis cell walls. Proc Natl Acad Sci U S A. 109 (4), 1329-1334 (2012).
- Tobimatsu, Y., et al. A click chemistry strategy for visualization of plant cell wall lignification. Chem. Commun. 50 (82), 12262-12265 (2014).
- Bukowski, N., et al. Development of a clickable designer monolignol for interrogation of lignification in plant cell walls. Bioconjugate Chem. 25 (12), 2189-2196 (2014).
- Pandey, J. L., et al. A versatile click-compatible monolignol probe to study lignin deposition in plant cell walls. PLOS ONE. 10 (4), e0121334 (2015).
- Pandey, J. L., et al. Investigating biochemical and developmental dependencies of lignification with a click-compatible monolignol analog in Arabidopsis thaliana stems. Front Plant Sci. 7, (2016).
- Zhu, Y., Wu, J., Chen, X. Metabolic labeling and imaging of N-linked glycans in Arabidopsis thaliana. Ange Chemie (Int Ed. Engl). 55 (32), 9301-9305 (2016).
- Boerjan, W., Ralph, J., Baucher, M. Lignin biosynthesis. Annu Rev Plant Biol. 54, 519-546 (2003).
- Weng, J. -. K., Chapple, C. The origin and evolution of lignin biosynthesis. New Phytol. 187 (2), 273-285 (2010).
- Mottiar, Y., Vanholme, R., Boerjan, W., Ralph, J., Mansfield, S. D. Designer lignins: harnessing the plasticity of lignification. Curr Opin Biotechnol. 37, 190-200 (2016).
- Rinaldi, R., et al. Paving the way for lignin valorisation: recent advances in bioengineering, biorefining and catalysis. Ange Chemie (Int Ed). 55 (29), 8164-8215 (2016).
- Wratil, P. R., Horstkorte, R., Reutter, W. Metabolic glycoengineering with N-acyl side chain modified mannosamines. Ange Chemie (Int Ed. Engl). 55 (33), 9482-9512 (2016).
- Feng, L., et al. Bifunctional unnatural sialic acids for dual metabolic labeling of cell-surface sialylated glycans. J Am Chem Soc. 135 (25), 9244-9247 (2013).
- Dumont, M., et al. Plant cell wall imaging by metabolic click-mediated labelling of rhamnogalacturonan II using azido 3-deoxy- d – manno -oct-2-ulosonic acid. Plant J. 85 (3), 437-447 (2016).
- Niederwieser, A., et al. Two-color glycan labeling of live cells by a combination of Diels-Alder and click chemistry. Ange Chemie Int Ed. 52 (15), 4265-4268 (2013).
- Späte, A. -. K., et al. Exploring the potential of norbornene-modified mannosamine derivatives for metabolic glycoengineering. Chem Bio Chem. 17 (14), 1374-1383 (2016).
- Späte, A. -. K., et al. Rapid Labeling of metabolically engineered cell-surface glycoconjugates with a carbamate-linked cyclopropene reporter. Bioconjugate Chem. 25 (1), 147-154 (2014).
- Lion, C., et al. BLISS: a bioorthogonal dual-labeling strategy to unravel lignification dynamics in plants. Cell Chem Biol. 24 (3), 326-338 (2017).
- del Río, J. C., et al. Structural characterization of guaiacyl-rich lignins in flax (Linum usitatissimum) fibers and shives. J Agric Food Chemist. 59 (20), 11088-11099 (2011).
- Huis, R., et al. Natural hypolignification is associated with extensive oligolignol accumulation in flax stems. Plant Physiol. 158 (4), 1893-1915 (2012).
- Chantreau, M., et al. Ectopic lignification in the flax lignified bast fiber1 mutant stem is associated with tissue-specific modifications in gene expression and cell wall composition. The Plant Cell. 26 (11), 4462-4482 (2014).
- Terashima, N., Ko, C., Matsushita, Y., Westermark, U. Monolignol glucosides as intermediate compounds in lignin biosynthesis. Revisiting the cell wall lignification and new 13C-tracer experiments with Ginkgo biloba and Magnolia liliiflora. Holzforschung. 70 (9), 801-810 (2016).
- Noel, M., et al. Probing the CMP-sialic acid donor specificity of two human β-d-galactoside sialyltransferases (ST3Gal I and ST6Gal I) selectively acting on O- and N-glycosylproteins. Chembiochem: Eur J Chem Biol. , (2017).

