उपन्यास प्राकृतिक उत्पादों को उजागर करने के लिए एक उपकरण के रूप में मास स्पेक्ट्रोमेट्री-गाइडेड जीनोम माइनिंग
Summary
एक बड़े पैमाने पर स्पेक्ट्रोमेट्री निर्देशित जीनोम खनन प्रोटोकॉल की स्थापना की और यहां वर्णित है । यह जीनोम अनुक्रम सूचना और एलसी-एमएस/एमएस विश्लेषण पर आधारित है और इसका उद्देश्य जटिल माइक्रोबियल और पौधों के अर्क से अणुओं की पहचान को सुगम बनाना है ।
Abstract
प्राकृतिक उत्पादों द्वारा कवर रासायनिक अंतरिक्ष अपार और व्यापक रूप से अपरिचित है । इसलिए, प्रकृति और संभावित मानव लाभों (जैसे, दवा खोज अनुप्रयोगों के लिए) में उनके कार्यों का व्यापक मूल्यांकन करने के लिए सुविधाजनक तरीके वांछित हैं। यह प्रोटोकॉल जीनोम खनन (जीएम) और आणविक नेटवर्किंग (एमएन) के संयोजन का वर्णन करता है, दो समकालीन दृष्टिकोण जो कच्चे मेटाबोलिक अर्क से रासायनिक संरचना हस्ताक्षरों के साथ पूरे जीनोम अनुक्रमण में जीन क्लस्टर-एन्कोडेड एनोटेशन से मेल खाते हैं। नई प्राकृतिक संस्थाओं की खोज की दिशा में यह पहला कदम है। इन अवधारणाओं, जब एक साथ लागू किया, एमएस निर्देशित जीनोम खनन के रूप में यहां परिभाषित कर रहे हैं । इस विधि में, मुख्य घटकों को पहले से नामित किया जाता है (एमएन का उपयोग करना), और संरचनात्मक रूप से संबंधित नए उम्मीदवार जीनोम अनुक्रम एनोटेशन (जीएम का उपयोग करके) से जुड़े होते हैं। जीएम और एमएन का संयोजन पहले से ज्ञात यौगिकों से एनालॉग की पहचान करने के लिए नए अणु बैकबोन या फसल मेटाबोलिक प्रोफाइल को लक्षित करने के लिए एक लाभदायक रणनीति है।
Introduction
माध्यमिक चयापचय की जांच में अक्सर विशिष्ट जैविक गतिविधियों के लिए कच्चे तेल के अर्क की स्क्रीनिंग शामिल होती है जिसके बाद सक्रिय अंशों से संबंधित घटकों की शुद्धि, पहचान और लक्षण वर्णन होता है। यह प्रक्रिया कुशल साबित हुई है, जिससे कई रासायनिक संस्थाओं के अलगाव को बढ़ावा मिल रहा है । हालांकि, आजकल इसे मुख्य रूप से पुनर्खोज की उच्च दरों के कारण अव्यवहार्य के रूप में देखा जाता है। चूंकि दवा उद्योग ने विशेष चयापचय की भूमिकाओं और कार्यों के ज्ञान के बिना क्रांति ला दी, इसलिए उनकी पहचान प्रयोगशाला की स्थितियों के तहत की गई थी जो प्रकृति1का सही प्रतिनिधित्व नहीं करती थी। आज, प्राकृतिक सिग्नलिंग प्रभावों, स्राव और अज्ञेय रूप से कम सांद्रता पर अधिकांश लक्ष्यों की उपस्थिति की बेहतर समझ है। इसके अतिरिक्त, इस प्रक्रिया के नियमन से अकादमिक समुदाय और दवा उद्योग को इस ज्ञान का लाभ उठाने में मदद मिलेगी । यह साइलेंट बायोसिंथेटिक जीन क्लस्टर (बीजीएस)2से संबंधित मेटाबोलाइट्स के सीधे अलगाव से जुड़े शोध को भी लाभ ान्वित करेगा ।
इस संदर्भ में, जीनोमिक अनुक्रमण में प्रगति ने सूक्ष्मजीव मेटाबोलाइट्स की स्क्रीनिंग में नए सिरे से रुचि ली है। इसका कारण यह है कि खुला बायोसिंथेटिक समूहों की जीनोमिक जानकारी का विश्लेषण जीन एन्कोडिंग उपन्यास यौगिकों को प्रकट कर सकता है जो प्रयोगशाला की स्थितियों के तहत नहीं मनाया या उत्पादित नहीं किया जाता है। कई माइक्रोबियल पूरे जीनोम परियोजनाएं या ड्राफ्ट आज उपलब्ध हैं, और यह संख्या हर साल बढ़ रही है, जीनोम खनन3,,4के माध्यम से उपन्यास जैव सक्रिय अणुओं को उजागर करने के लिए बड़े पैमाने पर संभावनाएं प्रदान करती हैं।
बायोसिंथेटिक जीन क्लस्टर के एटलस संयुक्त जीनोम संस्थान (JGI IMG-एबीसी)2के एकीकृत माइक्रोबियल जीनोम मंच के एक घटक के रूप में स्वचालित रूप से मिकेन जीन समूहों का वर्तमान सबसे बड़ा संग्रह है । हाल ही में, बायोसिंथेटिक जीन क्लस्टर्स (एमआईबीआईजी) मानकीकरण पहल के लिए न्यूनतम जानकारी ने बीजीएस के मैनुअल पुनर्निर्धारण को बढ़ावा दिया है, जो एक अत्यधिक क्यूरेटेड संदर्भ डेटासेट5प्रदान करता है। आजकल, आनुवंशिक डेटा के कम्प्यूटेशनल खनन और ज्ञात माध्यमिक मेटाबोलाइट्स के लिए उनके कनेक्शन को सक्षम करने के लिए बहुत सारे उपकरण उपलब्ध हैं। नए बायोएक्टिव प्राकृतिक उत्पादों (यानी, विषमलोगस अभिव्यक्ति, लक्ष्य जीन विलोपन, इन विट्रो पुनर्गठन, जीनोमिक अनुक्रम, आइसोटोप-गाइडेड स्क्रीनिंग [जेनोमिसोटोिक दृष्टिकोण], स्थानीय और वैश्विक नियामकों में हेरफेर, प्रतिरोध लक्ष्य आधारित खनन, संस्कृति स्वतंत्र खनन, और हाल ही में, एमएस-गाइडेड/कोड दृष्टिकोण2,,6,,7,,88,9, 9,, 10,11,12,13,14,15.
एक विलक्षण रणनीति के रूप में जीनोम खनन अणुओं के एक या छोटे समूह को एनोटेट करने के प्रयासों की आवश्यकता है; इस प्रकार, इस प्रक्रिया में अंतराल रहते हैं जिसमें अलगाव और संरचना व्याख्या के लिए नए यौगिकों को प्राथमिकता दी जाती है । सिद्धांत रूप में, ये दृष्टिकोण प्रति प्रयोग केवल एक बायोसिंथेटिक मार्ग को लक्षित करते हैं, जिसके परिणामस्वरूप धीमी खोज दर होती है। इस अर्थ में, आणविक नेटवर्किंग दृष्टिकोण के साथ जीएम का उपयोग करना प्राकृतिक उत्पाद अनुसंधान14,,15के लिए एक महत्वपूर्ण अग्रिम का प्रतिनिधित्व करता है।
तरल क्रोमेटोग्राफी-मास स्पेक्ट्रोमेट्री (एलसी-एमएस) की बहुमुखी प्रतिभा, सटीकता और उच्च संवेदनशीलता इसे यौगिक पहचान के लिए एक अच्छी विधि बनाती है। वर्तमान में, कई प्लेटफार्मों ने अलक्षित मेतापोलोमिक्स,16,17,18,,19,,20के लिए एल्गोरिदम और सॉफ्टवेयर सूट का निवेश किया है।, इन कार्यक्रमों के मूल में फीचर डिटेक्शन (पीक पिकिंग)21 और पीक अलाइनमेंट शामिल हैं, जो नमूनों के एक बैच में समान सुविधाओं के मिलान और पैटर्न की खोज की अनुमति देता है। एमएस पैटर्न आधारित एल्गोरिदम22,,23 विशेषता विखंडन पैटर्न की तुलना और मिलान एमएस2 समानताएं आणविक परिवारों संरचनात्मक सुविधाओं को साझा करने पैदा । इन विशेषताओं को तब हाइलाइट और क्लस्टर किया जा सकता है, जो,एमएस2,,24,25के साथ मिलकर एक जटिल जैविक उद्धरण से ज्ञात और अज्ञात अणुओं को तेजी से खोजने की क्षमता प्रदान करता है। इसलिए, एक साथ बड़ी मात्रा में डेटा में निहित कई कीमोटाइप की संरचनात्मक जानकारी हासिल करने के लिए मिलकर एमएस एक बहुमुखी तरीका है।
ग्लोबल नेचुरल प्रोडक्ट्स सोशल मॉलिक्यूलर नेटवर्किंग (जीएनपीएस)26 एल्गोरिदम बहुआयामी वैक्टर के निर्माण के लिए सामान्यीकृत टुकड़ा आयनों तीव्रता का उपयोग करता है, जिसमें समानताएं एक कोसाइन फ़ंक्शन का उपयोग करके तुलना की जाती हैं। विभिन्न माता-पिता आयनों के बीच संबंध को आरेख प्रतिनिधित्व में प्लॉट किया जाता है, जिसमें प्रत्येक विखंडन को नोड (सर्कल) के रूप में कल्पना की जाती है, और प्रत्येक नोड की संबंधितता को किनारे (लाइनों) द्वारा परिभाषित किया जाता है। एक ही स्रोत से अणुओं के वैश्विक दृश्य को आणविक नेटवर्क के रूप में परिभाषित किया गया है। संरचनात्मक रूप से अलग अणु जो विशिष्ट रूप से टुकड़े करते हैं, वे अपने विशिष्ट क्लस्टर या नक्षत्र का निर्माण करेंगे, जबकि संबंधित अणु एक साथ क्लस्टर करते हैं। क्लस्टरिंग कीमोटाइप्स अपने बायोसिंथेटिक मूल के समान संरचनात्मक सुविधाओं के काल्पनिक कनेक्शन की अनुमति देता है।
बीजीएस और उनके छोटे अणु उत्पादों27के बीच बायोइंफॉर्मेटिक्स लिंक बनाते समय कीमोटाइप-टू-जीनोटाइप और जीनोटाइप-टू-कीमोटाइप दृष्टिकोण दोनों का संयोजन शक्तिशाली होता है। इसलिए, एमएस निर्देशित जीनोम खनन एक तेजी से विधि और कम सामग्री लेने वाली रणनीति है, और यह विविध मेटाबोलिक और पर्यावरणीय स्थितियों के तहत एक या अधिक उपभेदों के डब्ल्यूजीएस द्वारा प्रकट माता-पिता आयनों और बायोसिंथेटिक रास्तों को पाटने में मदद करता है।
इस प्रोटोकॉल के कार्यप्रवाहमेंडब्ल्यूजीएस डेटा को बायोसिंथेटिक जीन क्लस्टर एनोटेशन प्लेटफॉर्म जैसे एंटीस्मैश28,29,,30में खिलाना होता है । यह जीनोम द्वारा एन्कोड यौगिकों और यौगिकों के वर्ग की विविधता का अनुमान लगाने में मदद करता है। ब्याज की रासायनिक इकाई को एन्कोडिंग करने वाले बायोसिंथेटिक जीन क्लस्टर को लक्षित करने की रणनीति अपनाई जानी चाहिए, और बीजीसी युक्त जंगली प्रकार के तनाव और/या विषमलोगस तनाव से संस्कृति अर्क का विश्लेषण जीएनपीएस26,,31का उपयोग करसमानताओं के आधार पर संकुल आयनों को उत्पन्न करने के लिए किया जा सकता है । नतीजतन, लक्षित बीजीसी के साथ संबद्ध नए अणुओं की पहचान करना संभव है और डेटाबेस में अनुपलब्ध हैं (मुख्य रूप से अज्ञात एनालॉग, कभी-कभी कम टिटर में उत्पादित)। यह विचार करना प्रासंगिक है कि उपयोगकर्ता इन प्लेटफार्मों में योगदान कर सकते हैं और बायोइन्फॉर्मेटिक्स और एमएस/एमएस डेटा की उपलब्धता तेजी से बढ़ रही है, जो अणुओं के साथ जटिल अर्क के कुशल कनेक्शन का मार्गदर्शन करने के लिए प्रभावी कम्प्यूटेशनल टूल और एल्गोरिदम के निरंतर विकास और उन्नयन के लिए ड्राइविंग कर रही है ।
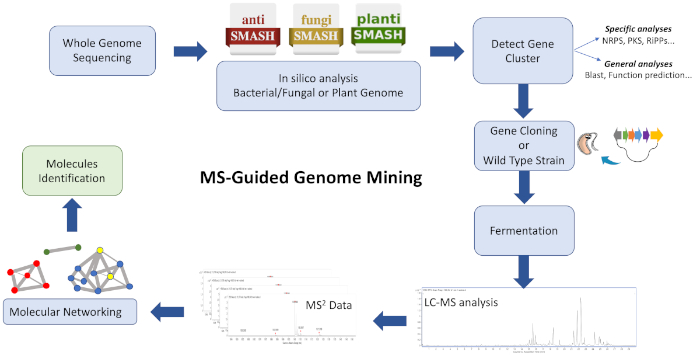
चित्रा 1: पूरे कार्यप्रवाह का अवलोकन। दिखाया गया है कि नए मेटाबोलाइट्स की पहचान करने के लिए वर्णित एमएस-गाइडेड जीनोम खनन दृष्टिकोण में शामिल बायोइंफॉर्मेटिक, क्लोनिंग और आणविक नेटवर्किंग चरणों का एक उदाहरण है। कृपया इस आंकड़े का एक बड़ा संस्करण देखने के लिए यहां क्लिक करें ।
यह प्रोटोकॉल प्राकृतिक उत्पाद खोज पाइपलाइन के लिए प्रारंभिक बिंदु के रूप में जीनोम खनन और आणविक नेटवर्किंग को संयोजित करने के लिए एक त्वरित और कुशल कार्यप्रवाह का वर्णन करता है। यद्यपि कई अनुप्रयोग एक नेटवर्क में एमएस-डिटेक्बल अणुओं की संरचना और संबंधितता की कल्पना करने में सक्षम हैं, संरचनात्मक रूप से समान संकुल अणुओं की कल्पना करने के लिए कई को यहां अपनाया जाता है। इस रणनीति का उपयोग करते हुए, स्ट्रेप्टोमाइस एसपी सीबीएमएआई 2042 के मेटाबोलिक अर्क में देखे गए उपन्यास साइक्लोडिपेप्टाइड उत्पादों की सफलतापूर्वक पहचान की जाती है। जीनोम खनन द्वारा निर्देशित, वैलिनोमाइसिन के लिए पूरे बायोसिंथेटिक जीन क्लस्टर एन्कोडिंग को मान्यता दी जाती है और उत्पादक तनाव स्ट्रेप्टोमाइसेस कोएलिकलर M1146 में क्लोन किया जाता है। अंत में, एमएस पैटर्न आधारित आणविक नेटवर्किंग के बाद, एमएस द्वारा पाए गए अणु ओं को उनके बायोजेनेसिस32के लिए जिम्मेदार बीजीसीएस के साथ सहसंबद्ध किया जाता है।
Protocol
Representative Results
Discussion
इस प्रोटोकॉल का सबसे मजबूत लाभ नए अणुओं, विशेष रूप से संरचनात्मक एनालॉग2की संरचनाओं को स्पष्ट करने के लिए एमएस डेटा के साथ मेटाबोलिक प्रोफाइल और पुल जीनोमिक जानकारी को तेजी से डिक्रिनेट करने ?…
Disclosures
The authors have nothing to disclose.
Acknowledgements
इस अध्ययन के लिए वित्तीय सहायता साओ पाउलो रिसर्च फाउंडेशन द्वारा प्रदान की गई थी – FAPESP (2019/10564-5, 2014/12727-5 और 2014/50249-8 से L.G.O; 2013/12598-8 और 2015/01013-4 से आरएस; और 2019/08853-9 से C.F.F.A. । B.S.P., C.F.F.A., और L.G.O. ने राष्ट्रीय वैज्ञानिक और प्रौद्योगिकीय विकास परिषद से फैलोशिप प्राप्त की-CNPq (205729/2018-5, 162191/2015-4, और 313492/2017-4) । L.G.O. विज्ञान में महिलाओं के लिए कार्यक्रम द्वारा प्रदान की अनुदान सहायता के लिए भी आभारी है (२००८, ब्राजील संस्करण) । सभी लेखकों CAPES स्वीकार करते है (उच्च शिक्षा कर्मियों के सुधार के लिए समन्वय) ब्राजील में स्नातकोत्तर कार्यक्रमों के बाद समर्थन के लिए ।
Materials
| Acetonitrile | Tedia | AA1120-048 | HPLC grade |
| Agar | Oxoid | LP0011 | NA |
| Apramycin | Sigma Aldrich | A2024 | NA |
| Carbenicillin | Sigma Aldrich | C9231 | NA |
| Centrifuge | Eppendorf | NA | 5804 |
| Chloramphenicol | Sigma Aldrich | C3175 | NA |
| Column C18 | Agilent Technologies | NA | ZORBAX RRHD Extend-C18, 80Å, 2.1 x 50 mm, 1.8 µm, 1200 bar pressure limit P/N 757700-902 |
| Kanamycin | Sigma Aldrich | K1377 | NA |
| Manitol P.A.- A.C.S. | Synth | NA | NA |
| Microcentrifuge | Eppendorf | NA | 5418 |
| Nalidixic acid | Sigma Aldrich | N4382 | NA |
| Phusion Flash High-Fidelity PCR Master Mix | ThermoFisher Scientific | F548S | NA |
| Q-TOF mass spectrometer | Agilent technologies | NA | 6550 iFunnel Q-TOF LC/MS |
| Sacarose P.A.- A.C.S. | Synth | NA | NA |
| Shaker/Incubator | Marconi | MA420 | NA |
| Sodium Chloride | Synth | NA | P. A. – ACS |
| Soy extract | NA | NA | NA |
| Sucrose | Synth | NA | P. A. – ACS |
| Thermal Cycles | Eppendorf | NA | Mastercycler Nexus Gradient |
| Thiostrepton | Sigma Aldrich | T8902 | NA |
| Tryptone | Oxoid | LP0042 | NA |
| Tryptone Soy Broth | Oxoid | CM0129 | NA |
| UPLC | Agilent Technologies | NA | 1290 Infinity LC System |
| Yeast extract | Oxoid | LP0021 | NA |
References
- Davies, J. Specialized microbial metabolites: functions and origins. The Journal of Antibiotics. 66 (7), 361-364 (2013).
- Ziemert, N., Alanjary, M., Weber, T. The evolution of genome mining in microbes – a review. Natural Product Reports. 33 (8), 988-1005 (2016).
- Zerikly, M., Challis, G. L. Strategies for the Discovery of New Natural Products by Genome Mining. ChemBioChem. 10 (4), 625-633 (2009).
- Gomez-Escribano, J. P., Bibb, M. J. Heterologous expression of natural product biosynthetic gene clusters in Streptomyces coelicolor: from genome mining to manipulation of biosynthetic pathways. Journal of Industrial Microbiology & Biotechnology. 41 (2), 425-431 (2014).
- Medema, M. H., et al. Minimum Information about a Biosynthetic Gene cluster. Nature Chemical Biology. 11 (9), 625-631 (2015).
- Lautru, S., Deeth, R. J., Bailey, L. M., Challis, G. L. Discovery of a new peptide natural product by Streptomyces coelicolor genome mining. Nature Chemical Biology. 1 (5), 265-269 (2005).
- Chiang, Y. -. M., et al. Molecular Genetic Mining of the Aspergillus Secondary Metabolome: Discovery of the Emericellamide Biosynthetic Pathway. Chemistry & Biology. 15 (6), 527-532 (2008).
- Huang, T., et al. Identification and Characterization of the Pyridomycin Biosynthetic Gene Cluster of Streptomyces pyridomyceticus NRRL B-2517. Journal of Biological Chemistry. 286 (23), 20648-20657 (2011).
- Udwary, D. W., et al. Genome sequencing reveals complex secondary metabolome in the marine actinomycete Salinispora tropica. Proceedings of the National Academy of Sciences. 104 (25), 10376-10381 (2007).
- Gross, H., et al. The Genomisotopic Approach: A Systematic Method to Isolate Products of Orphan Biosynthetic Gene Clusters. Chemistry & Biology. 14 (1), 53-63 (2007).
- Spohn, M., Wohlleben, W., Stegmann, E. Elucidation of the zinc-dependent regulation in Amycolatopsis japonicum enabled the identification of the ethylenediamine-disuccinate ([S,S ]-EDDS) genes. Environmental Microbiology. 18 (4), 1249-1263 (2016).
- Thaker, M. N., Waglechner, N., Wright, G. D. Antibiotic resistance-mediated isolation of scaffold-specific natural product producers. Nature Protocols. 9 (6), 1469-1479 (2014).
- Katz, M., Hover, B. M., Brady, S. F. Culture-independent discovery of natural products from soil metagenomes. Journal of Industrial Microbiology & Biotechnology. 43, 129-141 (2016).
- Quinn, R. A., et al. Molecular Networking as a Drug Discovery, Drug Metabolism, and Precision Medicine Strategy. Trends in Pharmacological Sciences. 38 (2), 143-154 (2017).
- Yang, J. Y., et al. Molecular Networking as a Dereplication Strategy. Journal of Natural Products. 76 (9), 1686-1699 (2013).
- Lommen, A. MetAlign: Interface-Driven, Versatile Metabolomics Tool for Hyphenated Full-Scan Mass Spectrometry Data Preprocessing. Analytical Chemistry. 81 (8), 3079-3086 (2009).
- Katajamaa, M., Miettinen, J., Oresic, M. MZmine: toolbox for processing and visualization of mass spectrometry based molecular profile data. Bioinformatics. 22 (5), 634-636 (2006).
- Pluskal, T., Castillo, S., Villar-Briones, A., Orešič, M. MZmine 2: Modular framework for processing, visualizing, and analyzing mass spectrometry-based molecular profile data. BMC Bioinformatics. 11 (1), 395 (2010).
- Tautenhahn, R., Patti, G. J., Rinehart, D., Siuzdak, G. XCMS Online: A Web-Based Platform to Process Untargeted Metabolomic Data. Analytical Chemistry. 84 (11), 5035-5039 (2012).
- Kuhl, C., Tautenhahn, R., Böttcher, C., Larson, T. R., Neumann, S. CAMERA: An Integrated Strategy for Compound Spectra Extraction and Annotation of Liquid Chromatography/Mass Spectrometry Data Sets. Analytical Chemistry. 84 (1), 283-289 (2012).
- Katajamaa, M., Orešič, M. Data processing for mass spectrometry-based metabolomics. Journal of Chromatography A. 1158, 318-328 (2007).
- Liu, W. -. T., et al. Interpretation of Tandem Mass Spectra Obtained from Cyclic Nonribosomal Peptides. Analytical Chemistry. 81 (11), 4200-4209 (2009).
- Ng, J., et al. Dereplication and de novo sequencing of nonribosomal peptides. Nature Methods. 6 (8), 596-599 (2009).
- Liaw, C., et al. Vitroprocines, new antibiotics against Acinetobacter baumannii, discovered from marine Vibrio sp. QWI-06 using mass-spectrometry-based metabolomics approach. Scientific Reports. 5 (1), 1-11 (2015).
- Kang, K. B., et al. Targeted Isolation of Neuroprotective Dicoumaroyl Neolignans and Lignans from Sageretia theezans Using in Silico Molecular Network Annotation Propagation-Based Dereplication. Journal of Natural Products. 81 (8), 1819-1828 (2018).
- Wang, M., et al. Sharing and community curation of mass spectrometry data with Global Natural Products Social Molecular Networking. Nature Biotechnology. 34 (8), 828-837 (2016).
- Doroghazi, J. R., et al. A roadmap for natural product discovery based on large-scale genomics and metabolomics. Nature Chemical Biology. 10 (11), 963-968 (2014).
- Medema, M. H., et al. antiSMASH: rapid identification, annotation and analysis of secondary metabolite biosynthesis gene clusters in bacterial and fungal genome sequences. Nucleic Acids Research. 39, 339-346 (2011).
- Weber, T., et al. antiSMASH 3.0-a comprehensive resource for the genome mining of biosynthetic gene clusters. Nucleic Acids Research. 43, 237-243 (2015).
- Blin, K., et al. antiSMASH 5.0: updates to the secondary metabolite genome mining pipeline. Nucleic Acids Research. 47, 81-87 (2019).
- Watrous, J., et al. Mass spectral molecular networking of living microbial colonies. Proceedings of the National Academy of Sciences. 109 (26), 1743-1752 (2012).
- Paulo, B. S., Sigrist, R., Angolini, C. F. F., De Oliveira, L. G. New Cyclodepsipeptide Derivatives Revealed by Genome Mining and Molecular Networking. ChemistrySelect. 4 (27), 7785-7790 (2019).
- Gonzaga de Oliveira, L., Sigrist, R., Sachetto Paulo, B., Samborskyy, M. Whole-Genome Sequence of the Endophytic Streptomyces sp. Strain CBMAI 2042, Isolated from Citrus sinensis. Microbiology Resource Announcements. 8 (2), 1-2 (2019).
- Aziz, R. K., et al. The RAST Server: Rapid Annotations using Subsystems Technology. BMC Genomics. 9 (1), 75 (2008).
- Nah, H. -. J., Pyeon, H. -. R., Kang, S. -. H., Choi, S. -. S., Kim, E. -. S. Cloning and Heterologous Expression of a Large-sized Natural Product Biosynthetic Gene Cluster in Streptomyces Species. Frontiers in Microbiology. 8, 1-10 (2017).
- Zhang, J. J., Tang, X., Moore, B. S. Genetic platforms for heterologous expression of microbial natural products. Natural Product Reports. 36 (9), 1313-1332 (2019).
- Alduina, R., et al. Artificial chromosome libraries of Streptomyces coelicolor A3(2) and Planobispora rosea. FEMS Microbiology Letters. 218 (1), 181-186 (2003).
- Jones, A. C., et al. Phage P1-Derived Artificial Chromosomes Facilitate Heterologous Expression of the FK506 Gene Cluster. PLoS One. 8 (7), 69319 (2013).
- Gomez-Escribano, J. P., Bibb, M. J. Engineering Streptomyces coelicolor for heterologous expression of secondary metabolite gene clusters. Microbial Biotechnology. 4 (2), 207-215 (2011).
- Cannell, R. J. P. . Natural Products Isolation. , (1998).
- Kersten, R. D., et al. A mass spectrometry-guided genome mining approach for natural product peptidogenomics. Nature Chemical Biology. 7 (11), 794-802 (2011).
- Kersten, R. D., et al. Glycogenomics as a mass spectrometry-guided genome-mining method for microbial glycosylated molecules. Proceedings of the National Academy of Sciences. 110 (47), 4407-4416 (2013).
- Liu, W., et al. MS/MS-based networking and peptidogenomics guided genome mining revealed the stenothricin gene cluster in Streptomyces roseosporus. The Journal of Antibiotics. 67 (1), 99-104 (2014).
- Duncan, K. R., et al. Molecular Networking and Pattern-Based Genome Mining Improves Discovery of Biosynthetic Gene Clusters and their Products from Salinispora Species. Chemistry & Biology. 22 (4), 460-471 (2015).
- Cao, L., et al. MetaMiner: A Scalable Peptidogenomics Approach for Discovery of Ribosomal Peptide Natural Products with Blind Modifications from Microbial Communities. Cell Systems. , (2019).
- Chen, L. -. Y., Cui, H. -. T., Su, C., Bai, F. -. W., Zhao, X. -. Q. Analysis of the complete genome sequence of a marine-derived strain Streptomyces sp. S063 CGMCC 14582 reveals its biosynthetic potential to produce novel anti-complement agents and peptides. PeerJ. 7 (1), 6122 (2019).
- Kim Tiam, S., et al. Insights into the Diversity of Secondary Metabolites of Planktothrix Using a Biphasic Approach Combining Global Genomics and Metabolomics. Toxins. 11 (9), 498 (2019).
- Özakin, S., Ince, E. Genome and metabolome mining of marine obligate Salinispora strains to discover new natural products. Turkish Journal of Biology. 43 (1), 28-36 (2019).
- Trivella, D. B. B., de Felicio, R. The Tripod for Bacterial Natural Product Discovery: Genome Mining, Silent Pathway Induction, and Mass Spectrometry-Based Molecular Networking. mSystems. 3 (2), 00160 (2018).
- Maansson, M., et al. An Integrated Metabolomic and Genomic Mining Workflow To Uncover the Biosynthetic Potential of Bacteria. mSystems. 1 (3), 1-14 (2016).
- Blin, K., Kim, H. U., Medema, M. H., Weber, T. Recent development of antiSMASH and other computational approaches to mine secondary metabolite biosynthetic gene clusters. Briefings in Bioinformatics. 20 (4), 1103-1113 (2019).
- Fisch, K. M. Biosynthesis of natural products by microbial iterative hybrid PKS-NRPS. RSC Advances. 3 (40), 18228-18247 (2013).
- Tatsuno, S., Arakawa, K., Kinashi, H. Analysis of Modular-iterative Mixed Biosynthesis of Lankacidin by Heterologous Expression and Gene Fusion. The Journal of Antibiotics. 60 (11), 700-708 (2007).
- Helfrich, E. J. N., Piel, J. Biosynthesis of polyketides by trans-AT polyketide synthases. Natural Product Reports. 33 (2), 231-316 (2016).

