Mass Spectrometrie-Guided Genome Mining als Werkzeug zur Aufdeckung neuartiger Naturprodukte
Summary
Hier wird ein Massenspektrometrie-geführtes Genom-Mining-Protokoll erstellt und beschrieben. Es basiert auf Genomsequenzinformationen und LC-MS/MS-Analysen und zielt darauf ab, die Identifizierung von Molekülen aus komplexen mikrobiellen und pflanzlichen Extrakten zu erleichtern.
Abstract
Der chemische Raum, der von natürlichen Produkten bedeckt wird, ist immens und weithin unerkannt. Daher sind bequeme Methoden zur umfassenden Bewertung ihrer Funktionen in der Natur und potenziellen menschlichen Nutzen (z. B. für Anwendungen zur Arzneimittelentdeckung) wünschenswert. Dieses Protokoll beschreibt die Kombination von Genom-Mining (GM) und molekularer Vernetzung (MN), zwei zeitgenössischen Ansätzen, die genclustercodierte Anmerkungen in der sequenziierten Gesamtheit des Genoms mit chemischen Struktursignaturen aus rohen metabolischen Extrakten abgleichen. Dies ist der erste Schritt zur Entdeckung neuer natürlicher Wesen. Diese Konzepte werden, wenn sie zusammen angewendet werden, hier als MS-geführter Genomabbau definiert. Bei dieser Methode sind die Hauptkomponenten zuvor (mit MN) gekennzeichnet, und strukturell verwandte neue Kandidaten werden mit Genomsequenzanmerkungen (mit GM) verknüpft. Die Kombination von GM und MN ist eine gewinnbringende Strategie, um neue Molekül-Rückgrate anzusprechen oder metabolische Profile zu ernten, um Analoga aus bereits bekannten Verbindungen zu identifizieren.
Introduction
Untersuchungen des Sekundärstoffwechsels bestehen häufig darin, Rohextrakte auf spezifische biologische Aktivitäten zu untersuchen, gefolgt von Reinigung, Identifizierung und Charakterisierung der Bestandteile aktiver Fraktionen. Dieser Prozess hat sich als effizient erwiesen und die Isolierung mehrerer chemischer Einheiten gefördert. Heutzutage wird dies jedoch als undurchführbar angesehen, vor allem aufgrund der hohen Wiederentdeckungsraten. Als die pharmazeutische Industrie revolutionierte, ohne die Rollen und Funktionen spezialisierter Metaboliten zu kennen, wurde ihre Identifizierung unter Laborbedingungen durchgeführt, die die Natur nicht genau darstellten1. Heute gibt es ein besseres Verständnis von natürlichen Signaleinflüssen, Sekretion und das Vorhandensein der meisten Ziele bei nicht nachweisbar niedrigen Konzentrationen. Darüber hinaus wird die Regulierung des Prozesses der akademischen Gemeinschaft und der pharmazeutischen Industrie helfen, dieses Wissen zu nutzen. Es wird auch der Forschung zugute kommen, die die direkte Isolierung von Metaboliten im Zusammenhang mit stillen biosynthetischen Genclustern (BGCs)2betrifft.
In diesem Zusammenhang haben Fortschritte bei der genomischen Sequenzierung das Interesse an screening-Mikroorganismenmetaboliten erneut geweckt. Dies liegt daran, dass die Analyse der genomischen Informationen von nicht entdeckten biosynthetischen Clustern Gene aufdecken kann, die neuartige Verbindungen kodieren, die nicht beobachtet oder unter Laborbedingungen produziert werden. Viele mikrobielle ganze Genomprojekte oder Entwürfe sind heute verfügbar, und die Zahl wächst jedes Jahr, was massive Aussichten für die Entdeckung neuer bioaktiver Moleküle durch Genomabbau3,4bietet.
Der Atlas der biosynthetischen Gencluster ist die derzeit größte Sammlung automatisch abgebauter Gencluster als Bestandteil der Integrated Microbial Genomes Platform des Joint Genome Institute (JGI IMG-ABC)2. Zuletzt hat die Standardisierungsinitiative Minimum Information for Biosynthetic Gene Clusters (MIBiG) die manuelle Reannotation von BGCs gefördert und einen hochgradig kuratierten Referenzdatensatz 5 zur Verfügunggestellt. Heutzutage stehen viele Werkzeuge zur Verfügung, um das rechnerische Mining genetischer Daten und deren Verbindung zu bekannten sekundären Metaboliten zu ermöglichen. Außerdem wurden verschiedene Strategien für den Zugang zu neuen bioaktiven Naturprodukten entwickelt (z. B. heterologe Expression, Zielgenlöschung, In-vitro-Rekonstitution, genomische Sequenz, isotopengeführtes Screening [genomisotopen Ansatz], Manipulation lokaler und globaler Regulierungsbehörden, resistenzzielbasierter Bergbau, kulturunabhängiger Bergbau und in jüngerer Zeit MS-geführte/code-Ansätze2,6,7,8,9, 10,11,12,13,14,15).
Genomabbau als einzigartige Strategie erfordert Anstrengungen, um eine einzelne oder kleine Gruppe von Molekülen zu kommentieren; So bleiben Lücken im Prozess, in denen neue Verbindungen für Isolation und Strukturaufklärung priorisiert werden. Im Prinzip zielen diese Ansätze nur auf einen biosynthetischen Pfad pro Experiment ab, was zu einer langsamen Entdeckungsrate führt. In diesem Sinne stellt die Verwendung von GM zusammen mit einem molekularen Vernetzungsansatz einen wichtigen Fortschritt für die Naturproduktforschungdar 14,15.
Die Vielseitigkeit, Genauigkeit und hohe Empfindlichkeit der Flüssigchromatographie-Massenspektrometrie (LC-MS) machen es zu einer guten Methode zur zusammengesetzten Identifizierung. Derzeit haben mehrere Plattformen Algorithmen und Software-Suiten für ungezielte Metabolomik16,17,18,19,20investiert. Der Kern dieser Programme umfasst die Feature-Erkennung (Peak Picking)21 und die Spitzenausrichtung, die die Übereinstimmung identischer Features über einen Stapel von Samples und die Suche nach Mustern ermöglicht. MS-Muster-basierte Algorithmen22,23 vergleichen charakteristische Fragmentierungsmuster und stimmen MS2-Ähnlichkeiten überein, die molekulare Familien mit strukturellen Merkmalen erzeugen. Diese Merkmale können dann hervorgehoben und gebündelt werden, was die Fähigkeit verleiht, bekannte und unbekannte Moleküle aus einem komplexen biologischen Extrakt durch Tandem MS2,24,25schnell zu entdecken. Daher ist Tandem MS eine vielseitige Methode, um strukturelle Informationen über mehrere Chemotypen zu erhalten, die in einer großen Datenmenge gleichzeitig enthalten sind.
Der Global Natural Products Social Molecular Networking (GNPS)26-Algorithmus verwendet die normalisierte Fragmentionen-Intensität, um multidimensionale Vektoren zu konstruieren, bei denen Ähnlichkeiten mit einer Kosinusfunktion verglichen werden. Die Beziehung zwischen verschiedenen übergeordneten Ionen wird in einer Diagrammdarstellung dargestellt, in der jede Fragmentierung als Knoten (Kreise) visualisiert wird und die Verwandtschaft jedes Knotens durch eine Kante (Linien) definiert wird. Die globale Visualisierung von Molekülen aus einer Hand wird als molekulares Netzwerk definiert. Strukturell divergierende Moleküle, die auf einzigartige Weise fragmentieren, bilden ihren eigenen spezifischen Cluster oder ihre eigene Konstellation, während sich verwandte Moleküle zusammenschließen. Clustering-Chemotypen ermöglichen die hypothetische Verbindung ähnlicher struktureller Merkmale mit ihren biosynthetischen Ursprüngen.
Die Kombination sowohl von Chemotyp-zu-Genotyp- als auch von Genotyp-zu-Chemotyp-Ansätzen ist eine starke Wirkung, wenn es darum geht, Bioinformatik-Verbindungen zwischen BGCs und ihren kleinen Molekülprodukten herzustellen27. Daher ist MS-geführter Genomabbau eine schnelle Methode und eine strategie mit geringem Materialverbrauch, und es hilft, Elternionen und biosynthetische Wege zu überbrücken, die von WGS einer oder mehrerer Stämme unter verschiedenen metabolischen und ökologischen Bedingungen aufgedeckt wurden.
Der Workflow dieses Protokolls (Abbildung 1) besteht darin, WGS-Daten in eine biosynthetische Gencluster-Anmerkungsplattform wie antiSMASH28,29,30einzuspeisen. Es hilft, die Vielfalt der Verbindungen und Klasse der Verbindungen durch das Genom kodiert zu schätzen. Es muss eine Strategie zur Zielart ierung eines biosynthetischen Genclusters angenommen werden, der für eine chemische Einheit von Interesse kodiert, und Kulturextrakte aus einem WildenStamm und/oder heterologen Stamm, der den BGC enthält, können analysiert werden, um clusterierte Ionen auf der Grundlage von Ähnlichkeiten mit GNPS26,31zu erzeugen. Folglich ist es möglich, neue Moleküle zu identifizieren, die mit dem zielgerichteten BGC assoziiert werden und in der Datenbank nicht verfügbar sind (hauptsächlich unbekannte Analoga, manchmal in niedrigen Tistern produziert). Es ist wichtig zu berücksichtigen, dass Benutzer zu diesen Plattformen beitragen können und dass die Verfügbarkeit von Bioinformatik und MS/MS-Daten rapide zunimmt, was zu einer ständigen Weiterentwicklung und Aktualisierung effektiver Rechenwerkzeuge und Algorithmen führt, um effiziente Verbindungen komplexer Extrakte mit Molekülen zu steuern.
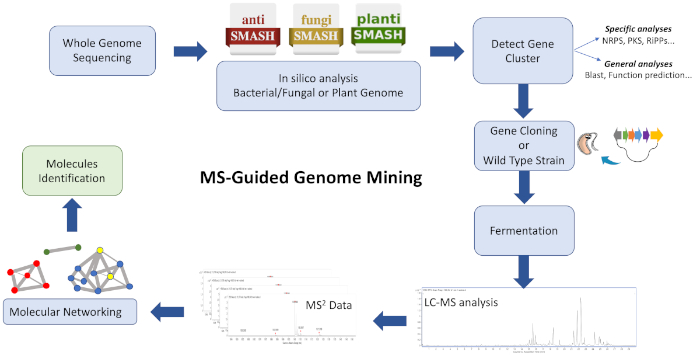
Abbildung 1: Übersicht über den gesamten Workflow. Gezeigt wird eine Illustration der bioinformatischen, klonenden und molekularen Vernetzungsschritte, die im beschriebenen MS-geführten Genom-Mining-Ansatz zur Identifizierung neuer Metaboliten beteiligt sind. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Dieses Protokoll beschreibt einen schnellen und effizienten Workflow, um Genom-Mining und molekulare Vernetzung als Ausgangspunkt für die Pipeline zur Entdeckung natürlicher Produkte zu kombinieren. Obwohl viele Anwendungen in der Lage sind, die Zusammensetzung und Verwandtheit von MS-detektierbaren Molekülen in einem Netzwerk zu visualisieren, werden hier mehrere verwendet, um strukturell ähnliche gruppierte Moleküle zu visualisieren. Mit dieser Strategie, neuartige Cyclodepsipeptid Produkte in metabolischen Extrakten von Streptomyces sp. CBMAI 2042 beobachtet werden erfolgreich identifiziert. Geleitet vom Genombergbau wird der gesamte biosynthetische Gencluster, der für Valinomycine kodiert, erkannt und in den Produzentenstamm Streptomyces coelicolor M1146 geklont. Schließlich korrelieren die von MS nachgewiesenen Moleküle nach einer MS-Muster-basierten molekularen Vernetzung mit BGCs, die für ihre Biogenese verantwortlich sind32.
Protocol
Representative Results
Discussion
Der stärkste Vorteil dieses Protokolls ist seine Fähigkeit, metabolische Profile schnell zu dereplizieren und genomische Informationen mit MS-Daten zu überbrücken, um die Strukturen neuer Moleküle, insbesondere struktureller Analoga2,aufzuklären. Auf der Grundlage genomischer Informationen können verschiedene natürliche Produkte- Chemotypen untersucht werden, wie Polyketide (PK), nichtribosomale Peptide (NRP) und glykosylierte Naturprodukte (BSP) sowie kryptische BGCs. Metamonomisches Scre…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Die finanzielle Unterstützung für diese Studie wurde von der Forschungsstiftung von Sao Paulo – FAPESP (2019/10564-5, 2014/12727-5 und 2014/50249-8 bis L.G.O; 2013/12598-8 und 2015/01013-4 an R.S.; und 2019/08853-9 an C.F.F.A). B.S.P, C.F.F.A. und L.G.O. erhielten Stipendien des National Council for Scientific and Technological Development – CNPq (205729/2018-5, 162191/2015-4 und 313492/2017-4). L.G.O. ist auch dankbar für die Stipendienförderung durch das Programm For Women in Science (2008, Brazilian Edition). Alle Autoren würdigen CAPES (Coordination for the Improvement of Higher Education Personnel) für die Unterstützung der Post-Graduation-Programme in Brasilien.
Materials
| Acetonitrile | Tedia | AA1120-048 | HPLC grade |
| Agar | Oxoid | LP0011 | NA |
| Apramycin | Sigma Aldrich | A2024 | NA |
| Carbenicillin | Sigma Aldrich | C9231 | NA |
| Centrifuge | Eppendorf | NA | 5804 |
| Chloramphenicol | Sigma Aldrich | C3175 | NA |
| Column C18 | Agilent Technologies | NA | ZORBAX RRHD Extend-C18, 80Å, 2.1 x 50 mm, 1.8 µm, 1200 bar pressure limit P/N 757700-902 |
| Kanamycin | Sigma Aldrich | K1377 | NA |
| Manitol P.A.- A.C.S. | Synth | NA | NA |
| Microcentrifuge | Eppendorf | NA | 5418 |
| Nalidixic acid | Sigma Aldrich | N4382 | NA |
| Phusion Flash High-Fidelity PCR Master Mix | ThermoFisher Scientific | F548S | NA |
| Q-TOF mass spectrometer | Agilent technologies | NA | 6550 iFunnel Q-TOF LC/MS |
| Sacarose P.A.- A.C.S. | Synth | NA | NA |
| Shaker/Incubator | Marconi | MA420 | NA |
| Sodium Chloride | Synth | NA | P. A. – ACS |
| Soy extract | NA | NA | NA |
| Sucrose | Synth | NA | P. A. – ACS |
| Thermal Cycles | Eppendorf | NA | Mastercycler Nexus Gradient |
| Thiostrepton | Sigma Aldrich | T8902 | NA |
| Tryptone | Oxoid | LP0042 | NA |
| Tryptone Soy Broth | Oxoid | CM0129 | NA |
| UPLC | Agilent Technologies | NA | 1290 Infinity LC System |
| Yeast extract | Oxoid | LP0021 | NA |
References
- Davies, J. Specialized microbial metabolites: functions and origins. The Journal of Antibiotics. 66 (7), 361-364 (2013).
- Ziemert, N., Alanjary, M., Weber, T. The evolution of genome mining in microbes – a review. Natural Product Reports. 33 (8), 988-1005 (2016).
- Zerikly, M., Challis, G. L. Strategies for the Discovery of New Natural Products by Genome Mining. ChemBioChem. 10 (4), 625-633 (2009).
- Gomez-Escribano, J. P., Bibb, M. J. Heterologous expression of natural product biosynthetic gene clusters in Streptomyces coelicolor: from genome mining to manipulation of biosynthetic pathways. Journal of Industrial Microbiology & Biotechnology. 41 (2), 425-431 (2014).
- Medema, M. H., et al. Minimum Information about a Biosynthetic Gene cluster. Nature Chemical Biology. 11 (9), 625-631 (2015).
- Lautru, S., Deeth, R. J., Bailey, L. M., Challis, G. L. Discovery of a new peptide natural product by Streptomyces coelicolor genome mining. Nature Chemical Biology. 1 (5), 265-269 (2005).
- Chiang, Y. -. M., et al. Molecular Genetic Mining of the Aspergillus Secondary Metabolome: Discovery of the Emericellamide Biosynthetic Pathway. Chemistry & Biology. 15 (6), 527-532 (2008).
- Huang, T., et al. Identification and Characterization of the Pyridomycin Biosynthetic Gene Cluster of Streptomyces pyridomyceticus NRRL B-2517. Journal of Biological Chemistry. 286 (23), 20648-20657 (2011).
- Udwary, D. W., et al. Genome sequencing reveals complex secondary metabolome in the marine actinomycete Salinispora tropica. Proceedings of the National Academy of Sciences. 104 (25), 10376-10381 (2007).
- Gross, H., et al. The Genomisotopic Approach: A Systematic Method to Isolate Products of Orphan Biosynthetic Gene Clusters. Chemistry & Biology. 14 (1), 53-63 (2007).
- Spohn, M., Wohlleben, W., Stegmann, E. Elucidation of the zinc-dependent regulation in Amycolatopsis japonicum enabled the identification of the ethylenediamine-disuccinate ([S,S ]-EDDS) genes. Environmental Microbiology. 18 (4), 1249-1263 (2016).
- Thaker, M. N., Waglechner, N., Wright, G. D. Antibiotic resistance-mediated isolation of scaffold-specific natural product producers. Nature Protocols. 9 (6), 1469-1479 (2014).
- Katz, M., Hover, B. M., Brady, S. F. Culture-independent discovery of natural products from soil metagenomes. Journal of Industrial Microbiology & Biotechnology. 43, 129-141 (2016).
- Quinn, R. A., et al. Molecular Networking as a Drug Discovery, Drug Metabolism, and Precision Medicine Strategy. Trends in Pharmacological Sciences. 38 (2), 143-154 (2017).
- Yang, J. Y., et al. Molecular Networking as a Dereplication Strategy. Journal of Natural Products. 76 (9), 1686-1699 (2013).
- Lommen, A. MetAlign: Interface-Driven, Versatile Metabolomics Tool for Hyphenated Full-Scan Mass Spectrometry Data Preprocessing. Analytical Chemistry. 81 (8), 3079-3086 (2009).
- Katajamaa, M., Miettinen, J., Oresic, M. MZmine: toolbox for processing and visualization of mass spectrometry based molecular profile data. Bioinformatics. 22 (5), 634-636 (2006).
- Pluskal, T., Castillo, S., Villar-Briones, A., Orešič, M. MZmine 2: Modular framework for processing, visualizing, and analyzing mass spectrometry-based molecular profile data. BMC Bioinformatics. 11 (1), 395 (2010).
- Tautenhahn, R., Patti, G. J., Rinehart, D., Siuzdak, G. XCMS Online: A Web-Based Platform to Process Untargeted Metabolomic Data. Analytical Chemistry. 84 (11), 5035-5039 (2012).
- Kuhl, C., Tautenhahn, R., Böttcher, C., Larson, T. R., Neumann, S. CAMERA: An Integrated Strategy for Compound Spectra Extraction and Annotation of Liquid Chromatography/Mass Spectrometry Data Sets. Analytical Chemistry. 84 (1), 283-289 (2012).
- Katajamaa, M., Orešič, M. Data processing for mass spectrometry-based metabolomics. Journal of Chromatography A. 1158, 318-328 (2007).
- Liu, W. -. T., et al. Interpretation of Tandem Mass Spectra Obtained from Cyclic Nonribosomal Peptides. Analytical Chemistry. 81 (11), 4200-4209 (2009).
- Ng, J., et al. Dereplication and de novo sequencing of nonribosomal peptides. Nature Methods. 6 (8), 596-599 (2009).
- Liaw, C., et al. Vitroprocines, new antibiotics against Acinetobacter baumannii, discovered from marine Vibrio sp. QWI-06 using mass-spectrometry-based metabolomics approach. Scientific Reports. 5 (1), 1-11 (2015).
- Kang, K. B., et al. Targeted Isolation of Neuroprotective Dicoumaroyl Neolignans and Lignans from Sageretia theezans Using in Silico Molecular Network Annotation Propagation-Based Dereplication. Journal of Natural Products. 81 (8), 1819-1828 (2018).
- Wang, M., et al. Sharing and community curation of mass spectrometry data with Global Natural Products Social Molecular Networking. Nature Biotechnology. 34 (8), 828-837 (2016).
- Doroghazi, J. R., et al. A roadmap for natural product discovery based on large-scale genomics and metabolomics. Nature Chemical Biology. 10 (11), 963-968 (2014).
- Medema, M. H., et al. antiSMASH: rapid identification, annotation and analysis of secondary metabolite biosynthesis gene clusters in bacterial and fungal genome sequences. Nucleic Acids Research. 39, 339-346 (2011).
- Weber, T., et al. antiSMASH 3.0-a comprehensive resource for the genome mining of biosynthetic gene clusters. Nucleic Acids Research. 43, 237-243 (2015).
- Blin, K., et al. antiSMASH 5.0: updates to the secondary metabolite genome mining pipeline. Nucleic Acids Research. 47, 81-87 (2019).
- Watrous, J., et al. Mass spectral molecular networking of living microbial colonies. Proceedings of the National Academy of Sciences. 109 (26), 1743-1752 (2012).
- Paulo, B. S., Sigrist, R., Angolini, C. F. F., De Oliveira, L. G. New Cyclodepsipeptide Derivatives Revealed by Genome Mining and Molecular Networking. ChemistrySelect. 4 (27), 7785-7790 (2019).
- Gonzaga de Oliveira, L., Sigrist, R., Sachetto Paulo, B., Samborskyy, M. Whole-Genome Sequence of the Endophytic Streptomyces sp. Strain CBMAI 2042, Isolated from Citrus sinensis. Microbiology Resource Announcements. 8 (2), 1-2 (2019).
- Aziz, R. K., et al. The RAST Server: Rapid Annotations using Subsystems Technology. BMC Genomics. 9 (1), 75 (2008).
- Nah, H. -. J., Pyeon, H. -. R., Kang, S. -. H., Choi, S. -. S., Kim, E. -. S. Cloning and Heterologous Expression of a Large-sized Natural Product Biosynthetic Gene Cluster in Streptomyces Species. Frontiers in Microbiology. 8, 1-10 (2017).
- Zhang, J. J., Tang, X., Moore, B. S. Genetic platforms for heterologous expression of microbial natural products. Natural Product Reports. 36 (9), 1313-1332 (2019).
- Alduina, R., et al. Artificial chromosome libraries of Streptomyces coelicolor A3(2) and Planobispora rosea. FEMS Microbiology Letters. 218 (1), 181-186 (2003).
- Jones, A. C., et al. Phage P1-Derived Artificial Chromosomes Facilitate Heterologous Expression of the FK506 Gene Cluster. PLoS One. 8 (7), 69319 (2013).
- Gomez-Escribano, J. P., Bibb, M. J. Engineering Streptomyces coelicolor for heterologous expression of secondary metabolite gene clusters. Microbial Biotechnology. 4 (2), 207-215 (2011).
- Cannell, R. J. P. . Natural Products Isolation. , (1998).
- Kersten, R. D., et al. A mass spectrometry-guided genome mining approach for natural product peptidogenomics. Nature Chemical Biology. 7 (11), 794-802 (2011).
- Kersten, R. D., et al. Glycogenomics as a mass spectrometry-guided genome-mining method for microbial glycosylated molecules. Proceedings of the National Academy of Sciences. 110 (47), 4407-4416 (2013).
- Liu, W., et al. MS/MS-based networking and peptidogenomics guided genome mining revealed the stenothricin gene cluster in Streptomyces roseosporus. The Journal of Antibiotics. 67 (1), 99-104 (2014).
- Duncan, K. R., et al. Molecular Networking and Pattern-Based Genome Mining Improves Discovery of Biosynthetic Gene Clusters and their Products from Salinispora Species. Chemistry & Biology. 22 (4), 460-471 (2015).
- Cao, L., et al. MetaMiner: A Scalable Peptidogenomics Approach for Discovery of Ribosomal Peptide Natural Products with Blind Modifications from Microbial Communities. Cell Systems. , (2019).
- Chen, L. -. Y., Cui, H. -. T., Su, C., Bai, F. -. W., Zhao, X. -. Q. Analysis of the complete genome sequence of a marine-derived strain Streptomyces sp. S063 CGMCC 14582 reveals its biosynthetic potential to produce novel anti-complement agents and peptides. PeerJ. 7 (1), 6122 (2019).
- Kim Tiam, S., et al. Insights into the Diversity of Secondary Metabolites of Planktothrix Using a Biphasic Approach Combining Global Genomics and Metabolomics. Toxins. 11 (9), 498 (2019).
- Özakin, S., Ince, E. Genome and metabolome mining of marine obligate Salinispora strains to discover new natural products. Turkish Journal of Biology. 43 (1), 28-36 (2019).
- Trivella, D. B. B., de Felicio, R. The Tripod for Bacterial Natural Product Discovery: Genome Mining, Silent Pathway Induction, and Mass Spectrometry-Based Molecular Networking. mSystems. 3 (2), 00160 (2018).
- Maansson, M., et al. An Integrated Metabolomic and Genomic Mining Workflow To Uncover the Biosynthetic Potential of Bacteria. mSystems. 1 (3), 1-14 (2016).
- Blin, K., Kim, H. U., Medema, M. H., Weber, T. Recent development of antiSMASH and other computational approaches to mine secondary metabolite biosynthetic gene clusters. Briefings in Bioinformatics. 20 (4), 1103-1113 (2019).
- Fisch, K. M. Biosynthesis of natural products by microbial iterative hybrid PKS-NRPS. RSC Advances. 3 (40), 18228-18247 (2013).
- Tatsuno, S., Arakawa, K., Kinashi, H. Analysis of Modular-iterative Mixed Biosynthesis of Lankacidin by Heterologous Expression and Gene Fusion. The Journal of Antibiotics. 60 (11), 700-708 (2007).
- Helfrich, E. J. N., Piel, J. Biosynthesis of polyketides by trans-AT polyketide synthases. Natural Product Reports. 33 (2), 231-316 (2016).

