L’extraction du génome guidée par spectrométrie de masse comme outil pour découvrir de nouveaux produits naturels
Summary
Un protocole d’extraction du génome guidé par spectrométrie de masse est établi et décrit ici. Il est basé sur l’information sur la séquence génomique et l’analyse LC-MS/MS et vise à faciliter l’identification des molécules à partir d’extraits microbiens et végétaux complexes.
Abstract
L’espace chimique couvert par les produits naturels est immense et largement méconnu. Par conséquent, des méthodologies pratiques pour effectuer une évaluation de grande envergure de leurs fonctions dans la nature et les avantages humains potentiels (p. ex., pour les applications de découverte de médicaments) sont souhaitées. Ce protocole décrit la combinaison de l’extraction du génome (GM) et du réseautage moléculaire (MN), deux approches contemporaines qui correspondent aux annotations codées par les grappes génétiques dans le séquençage du génome entier avec des signatures de structure chimique provenant d’extraits métaboliques bruts. C’est la première étape vers la découverte de nouvelles entités naturelles. Ces concepts, lorsqu’ils sont appliqués ensemble, sont définis ici comme l’exploitation du génome guidée par la SP. Dans cette méthode, les composants principaux sont auparavant désignés (à l’aide de MN), et les nouveaux candidats structurellement liés sont associés à des annotations de séquences génomiques (à l’aide de GM). La combinaison de GM et de MN est une stratégie rentable visant à cibler de nouvelles colonnes dorsales de molécules ou à récolter des profils métaboliques afin d’identifier les analogues des composés déjà connus.
Introduction
Les études sur le métabolisme secondaire consistent souvent à filtrer des extraits bruts pour des activités biologiques spécifiques suivies de la purification, de l’identification et de la caractérisation des constituants appartenant à des fractions actives. Ce processus s’est avéré efficace, favorisant l’isolement de plusieurs entités chimiques. Cependant, aujourd’hui, cela est considéré comme irréalisable, principalement en raison des taux élevés de redécouverte. Comme l’industrie pharmaceutique a révolutionné sans connaître les rôles et les fonctions des métabolites spécialisés, leur identification a été effectuée dans des conditions de laboratoire qui ne représentaient pas exactement la nature1. Aujourd’hui, il y a une meilleure compréhension des influences naturelles de signalisation, de la sécrétion, et de la présence de la plupart des cibles à des concentrations anormalement basses. De plus, la réglementation du processus aidera la communauté universitaire et l’industrie pharmaceutique à tirer parti de ces connaissances. Il bénéficiera également à la recherche impliquant l’isolement direct des métabolites liés aux grappes de gènes biosynthétiques silencieuses (BGC)2.
Dans ce contexte, les progrès du séquençage génomique ont renouvelé leur intérêt pour le dépistage des métabolites micro-organismes. C’est parce que l’analyse de l’information génomique des amas biosynthétiques non découverts peut révéler des gènes codant de nouveaux composés non observés ou produits dans des conditions de laboratoire. De nombreux projets ou projets de génome entier microbien sont disponibles aujourd’hui, et le nombre augmente chaque année, offrant des perspectives massives pour découvrir de nouvelles molécules bioactives grâce à l’exploitation minière du génome3,4.
L’Atlas of Biosynthetic Gene Clusters est la plus grande collection actuelle de grappes de gènes automatiquement extraites en tant que composant de la plate-forme intégrée des génomes microbiens de l’Institut mixte du génome (JGI IMG-ABC)2. Plus récemment, l’Initiative de normalisation des grappes de gènes biosynthétiques (MIBiG) a favorisé la reannotation manuelle des BGC, fournissant un ensemble de données de référence très organisé5. Aujourd’hui, de nombreux outils sont disponibles pour permettre l’extraction informatique des données génétiques et leur connexion aux métabolites secondaires connus. Différentes stratégies ont également été élaborées pour accéder à de nouveaux produits naturels bioactifs (c.-à-d. l’expression hétérogène, la suppression des gènes cibles, la reconstitution in vitro, la séquence génomique, le dépistage guidé par isotopes [approche génoto-américaine], la manipulation des régulateurs locaux et mondiaux, l’exploitation minière axée sur les cibles de résistance, l’exploitation minière indépendante de la culture et, plus récemment, les approches de ms-guided/code2,6,7,8,9, 10,11,12,13,14,15).
L’extraction du génome comme stratégie singulière nécessite des efforts pour annoter un seul ou un petit groupe de molécules; ainsi, des lacunes subsistent dans le processus dans lequel de nouveaux composés sont priorisés pour l’isolement et l’élucidation de la structure. En principe, ces approches ne ciblent qu’une seule voie biosynthétique par expérience, ce qui entraîne un faible taux de découverte. En ce sens, l’utilisation de GM avec une approche de réseautage moléculaire représente une avancée importante pour la recherche sur les produits naturels14,15.
La polyvalence, la précision et la sensibilité élevée de la spectrométrie de masse chromatographie liquide (LC-MS) en font une bonne méthode d’identification composée. Actuellement, plusieurs plates-formes ont investi des algorithmes et des suites logicielles pour métabolomiques non ciblés16,17,18,19,20. Le cœur de ces programmes comprend la détection des fonctionnalités (pic de sélection)21 et l’alignement de pointe, ce qui permet de faire correspondre des caractéristiques identiques à travers un lot d’échantillons et de rechercher des modèles. Les algorithmes basés sur les modèles de SP22,23 comparent les modèles caractéristiques de fragmentation et correspondent aux similitudes de MS2 générant des familles moléculaires partageant des caractéristiques structurelles. Ces caractéristiques peuvent ensuite être mises en évidence et regroupées, conférant la possibilité de découvrir rapidement des molécules connues et inconnues à partir d’un extrait biologique complexe par tandem MS2,24,25. Par conséquent, la SP tandem est une méthode polyvalente pour obtenir des informations structurelles de plusieurs chemotypes contenus dans une grande quantité de données simultanément.
L’algorithme Global Natural Products Social Molecular Networking (GNPS)26 utilise l’intensité normalisée des ions fragmentés pour construire des vecteurs multidimensionnels, dans lesquels les similitudes sont comparées à l’aide d’une fonction cosine. La relation entre les différents ions parentaux est tracée dans une représentation de diagramme, dans laquelle chaque fragmentation est visualisée comme un nœud (cercles), et la relation de chaque nœud est définie par un bord (lignes). La visualisation globale des molécules à partir d’une seule source est définie comme un réseau moléculaire. Des molécules structurellement divergentes qui se fragmentent de façon unique formeront leur propre amas ou constellation spécifique, tandis que les molécules apparentées s’assemblent. Les chemotypes de regroupement permettent la connexion hypothétique de caractéristiques structurelles similaires à leurs origines biosynthétiques.
La combinaison des approches chemotype-génotype et génotype-chemotype est puissante lors de la création de liens bioinformatiques entre les BGC et leurs produits à petites molécules27. Par conséquent, l’extraction du génome guidée par ms est une méthode rapide et une stratégie peu consommatrice de matériaux, et elle aide à combler les ions parentaux et les voies biosynthétiques révélées par WGS d’une ou plusieurs souches dans diverses conditions métaboliques et environnementales.
Le flux de travail de ce protocole (figure 1) consiste à alimenter les données WGS dans une plate-forme d’annotation de grappes génétiques biosynthétiques telles que l’antiSMASH28,29,30. Il permet d’estimer la variété des composés et la classe des composés codés par le génome. Une stratégie visant à cibler un groupe génétique biosynthétique codant une entité chimique d’intérêt doit être adoptée, et des extraits de culture d’une souche de type sauvage et/ou d’une souche hétérologue contenant le BGC peuvent être analysés pour générer des ions groupés basés sur des similitudes à l’aide duPNBS 26,31. Par conséquent, il est possible d’identifier de nouvelles molécules qui s’associent au BGC ciblé et ne sont pas disponibles dans la base de données (principalement des analogues inconnus, parfois produits en bas de la vie). Il est pertinent de considérer que les utilisateurs peuvent contribuer à ces plates-formes et que la disponibilité des données bioinformatiques et MS/MS augmente rapidement, conduisant à un développement constant et la mise à niveau d’outils et d’algorithmes informatiques efficaces pour guider les connexions efficaces d’extraits complexes avec des molécules.
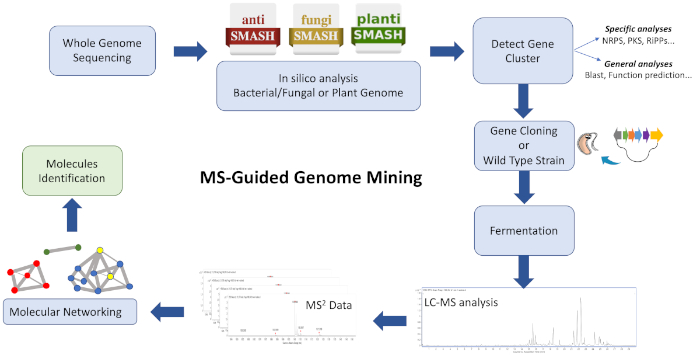
Figure 1 : Aperçu de l’ensemble du flux de travail. On y voit une illustration des étapes bioinformatiques, clonage et de réseautage moléculaire impliquées dans l’approche décrite d’extraction du génome guidée par ms pour identifier de nouveaux métabolites. S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.
Ce protocole décrit un flux de travail rapide et efficace pour combiner l’extraction du génome et le réseautage moléculaire comme point de départ pour le pipeline naturel de découverte de produits. Bien que de nombreuses applications soient capables de visualiser la composition et la dataabilité des molécules détectables par la SP dans un réseau, plusieurs sont adoptées ici pour visualiser des molécules groupées structurellement similaires. En utilisant cette stratégie, de nouveaux produits de cyclodepsipeptide observés dans les extraits métaboliques de Streptomyces sp. CBMAI 2042 sont identifiés avec succès. Guidé par l’extraction du génome, l’ensemble du codage de faisceau de gènes biosynthétique pour les valinomycines est reconnu et cloné dans la souche productrice Streptomyces coelicolor M1146. Enfin, à la suite d’un réseau moléculaire basé sur le modèle de SP, les molécules détectées par LAS sont corrélées avec les BGC responsables de leur biogenèse32.
Protocol
Representative Results
Discussion
L’avantage le plus fort de ce protocole est sa capacité à déposifier rapidement les profils métaboliques et à combler l’information génomique avec les données de la SP afin d’élucider les structures de nouvelles molécules, en particulier les analogues structurels2. Sur la base d’informations génomiques, différents produits naturels chemotypes peuvent être étudiés, tels que les polyketides (PK), peptides nonribosomal (NRP), et les produits naturels glycosylés (PNB), ainsi que…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Le soutien financier apporté à cette étude a été fourni par la Fondation de recherche de Sao Paulo – FAPESP (2019/10564-5, 2014/12727-5 et 2014/50249-8 à L.G.O; 2013/12598-8 et 2015/01013-4 à R.S.; et 2019/08853-9 à C.F.F.A). B.S.P, C.F.F.A., et L.G.O. ont reçu des bourses du Conseil national pour le développement scientifique et technologique – CNPq (205729/2018-5, 162191/2015-4, et 313492/2017-4). L.G.O. est également reconnaissant pour le soutien de subvention fourni par le programme Pour les femmes en sciences (2008, Édition brésilienne). Tous les auteurs reconnaissent capeS (Coordination pour l’amélioration du personnel de l’enseignement supérieur) pour soutenir les programmes post-diplôme au Brésil.
Materials
| Acetonitrile | Tedia | AA1120-048 | HPLC grade |
| Agar | Oxoid | LP0011 | NA |
| Apramycin | Sigma Aldrich | A2024 | NA |
| Carbenicillin | Sigma Aldrich | C9231 | NA |
| Centrifuge | Eppendorf | NA | 5804 |
| Chloramphenicol | Sigma Aldrich | C3175 | NA |
| Column C18 | Agilent Technologies | NA | ZORBAX RRHD Extend-C18, 80Å, 2.1 x 50 mm, 1.8 µm, 1200 bar pressure limit P/N 757700-902 |
| Kanamycin | Sigma Aldrich | K1377 | NA |
| Manitol P.A.- A.C.S. | Synth | NA | NA |
| Microcentrifuge | Eppendorf | NA | 5418 |
| Nalidixic acid | Sigma Aldrich | N4382 | NA |
| Phusion Flash High-Fidelity PCR Master Mix | ThermoFisher Scientific | F548S | NA |
| Q-TOF mass spectrometer | Agilent technologies | NA | 6550 iFunnel Q-TOF LC/MS |
| Sacarose P.A.- A.C.S. | Synth | NA | NA |
| Shaker/Incubator | Marconi | MA420 | NA |
| Sodium Chloride | Synth | NA | P. A. – ACS |
| Soy extract | NA | NA | NA |
| Sucrose | Synth | NA | P. A. – ACS |
| Thermal Cycles | Eppendorf | NA | Mastercycler Nexus Gradient |
| Thiostrepton | Sigma Aldrich | T8902 | NA |
| Tryptone | Oxoid | LP0042 | NA |
| Tryptone Soy Broth | Oxoid | CM0129 | NA |
| UPLC | Agilent Technologies | NA | 1290 Infinity LC System |
| Yeast extract | Oxoid | LP0021 | NA |
References
- Davies, J. Specialized microbial metabolites: functions and origins. The Journal of Antibiotics. 66 (7), 361-364 (2013).
- Ziemert, N., Alanjary, M., Weber, T. The evolution of genome mining in microbes – a review. Natural Product Reports. 33 (8), 988-1005 (2016).
- Zerikly, M., Challis, G. L. Strategies for the Discovery of New Natural Products by Genome Mining. ChemBioChem. 10 (4), 625-633 (2009).
- Gomez-Escribano, J. P., Bibb, M. J. Heterologous expression of natural product biosynthetic gene clusters in Streptomyces coelicolor: from genome mining to manipulation of biosynthetic pathways. Journal of Industrial Microbiology & Biotechnology. 41 (2), 425-431 (2014).
- Medema, M. H., et al. Minimum Information about a Biosynthetic Gene cluster. Nature Chemical Biology. 11 (9), 625-631 (2015).
- Lautru, S., Deeth, R. J., Bailey, L. M., Challis, G. L. Discovery of a new peptide natural product by Streptomyces coelicolor genome mining. Nature Chemical Biology. 1 (5), 265-269 (2005).
- Chiang, Y. -. M., et al. Molecular Genetic Mining of the Aspergillus Secondary Metabolome: Discovery of the Emericellamide Biosynthetic Pathway. Chemistry & Biology. 15 (6), 527-532 (2008).
- Huang, T., et al. Identification and Characterization of the Pyridomycin Biosynthetic Gene Cluster of Streptomyces pyridomyceticus NRRL B-2517. Journal of Biological Chemistry. 286 (23), 20648-20657 (2011).
- Udwary, D. W., et al. Genome sequencing reveals complex secondary metabolome in the marine actinomycete Salinispora tropica. Proceedings of the National Academy of Sciences. 104 (25), 10376-10381 (2007).
- Gross, H., et al. The Genomisotopic Approach: A Systematic Method to Isolate Products of Orphan Biosynthetic Gene Clusters. Chemistry & Biology. 14 (1), 53-63 (2007).
- Spohn, M., Wohlleben, W., Stegmann, E. Elucidation of the zinc-dependent regulation in Amycolatopsis japonicum enabled the identification of the ethylenediamine-disuccinate ([S,S ]-EDDS) genes. Environmental Microbiology. 18 (4), 1249-1263 (2016).
- Thaker, M. N., Waglechner, N., Wright, G. D. Antibiotic resistance-mediated isolation of scaffold-specific natural product producers. Nature Protocols. 9 (6), 1469-1479 (2014).
- Katz, M., Hover, B. M., Brady, S. F. Culture-independent discovery of natural products from soil metagenomes. Journal of Industrial Microbiology & Biotechnology. 43, 129-141 (2016).
- Quinn, R. A., et al. Molecular Networking as a Drug Discovery, Drug Metabolism, and Precision Medicine Strategy. Trends in Pharmacological Sciences. 38 (2), 143-154 (2017).
- Yang, J. Y., et al. Molecular Networking as a Dereplication Strategy. Journal of Natural Products. 76 (9), 1686-1699 (2013).
- Lommen, A. MetAlign: Interface-Driven, Versatile Metabolomics Tool for Hyphenated Full-Scan Mass Spectrometry Data Preprocessing. Analytical Chemistry. 81 (8), 3079-3086 (2009).
- Katajamaa, M., Miettinen, J., Oresic, M. MZmine: toolbox for processing and visualization of mass spectrometry based molecular profile data. Bioinformatics. 22 (5), 634-636 (2006).
- Pluskal, T., Castillo, S., Villar-Briones, A., Orešič, M. MZmine 2: Modular framework for processing, visualizing, and analyzing mass spectrometry-based molecular profile data. BMC Bioinformatics. 11 (1), 395 (2010).
- Tautenhahn, R., Patti, G. J., Rinehart, D., Siuzdak, G. XCMS Online: A Web-Based Platform to Process Untargeted Metabolomic Data. Analytical Chemistry. 84 (11), 5035-5039 (2012).
- Kuhl, C., Tautenhahn, R., Böttcher, C., Larson, T. R., Neumann, S. CAMERA: An Integrated Strategy for Compound Spectra Extraction and Annotation of Liquid Chromatography/Mass Spectrometry Data Sets. Analytical Chemistry. 84 (1), 283-289 (2012).
- Katajamaa, M., Orešič, M. Data processing for mass spectrometry-based metabolomics. Journal of Chromatography A. 1158, 318-328 (2007).
- Liu, W. -. T., et al. Interpretation of Tandem Mass Spectra Obtained from Cyclic Nonribosomal Peptides. Analytical Chemistry. 81 (11), 4200-4209 (2009).
- Ng, J., et al. Dereplication and de novo sequencing of nonribosomal peptides. Nature Methods. 6 (8), 596-599 (2009).
- Liaw, C., et al. Vitroprocines, new antibiotics against Acinetobacter baumannii, discovered from marine Vibrio sp. QWI-06 using mass-spectrometry-based metabolomics approach. Scientific Reports. 5 (1), 1-11 (2015).
- Kang, K. B., et al. Targeted Isolation of Neuroprotective Dicoumaroyl Neolignans and Lignans from Sageretia theezans Using in Silico Molecular Network Annotation Propagation-Based Dereplication. Journal of Natural Products. 81 (8), 1819-1828 (2018).
- Wang, M., et al. Sharing and community curation of mass spectrometry data with Global Natural Products Social Molecular Networking. Nature Biotechnology. 34 (8), 828-837 (2016).
- Doroghazi, J. R., et al. A roadmap for natural product discovery based on large-scale genomics and metabolomics. Nature Chemical Biology. 10 (11), 963-968 (2014).
- Medema, M. H., et al. antiSMASH: rapid identification, annotation and analysis of secondary metabolite biosynthesis gene clusters in bacterial and fungal genome sequences. Nucleic Acids Research. 39, 339-346 (2011).
- Weber, T., et al. antiSMASH 3.0-a comprehensive resource for the genome mining of biosynthetic gene clusters. Nucleic Acids Research. 43, 237-243 (2015).
- Blin, K., et al. antiSMASH 5.0: updates to the secondary metabolite genome mining pipeline. Nucleic Acids Research. 47, 81-87 (2019).
- Watrous, J., et al. Mass spectral molecular networking of living microbial colonies. Proceedings of the National Academy of Sciences. 109 (26), 1743-1752 (2012).
- Paulo, B. S., Sigrist, R., Angolini, C. F. F., De Oliveira, L. G. New Cyclodepsipeptide Derivatives Revealed by Genome Mining and Molecular Networking. ChemistrySelect. 4 (27), 7785-7790 (2019).
- Gonzaga de Oliveira, L., Sigrist, R., Sachetto Paulo, B., Samborskyy, M. Whole-Genome Sequence of the Endophytic Streptomyces sp. Strain CBMAI 2042, Isolated from Citrus sinensis. Microbiology Resource Announcements. 8 (2), 1-2 (2019).
- Aziz, R. K., et al. The RAST Server: Rapid Annotations using Subsystems Technology. BMC Genomics. 9 (1), 75 (2008).
- Nah, H. -. J., Pyeon, H. -. R., Kang, S. -. H., Choi, S. -. S., Kim, E. -. S. Cloning and Heterologous Expression of a Large-sized Natural Product Biosynthetic Gene Cluster in Streptomyces Species. Frontiers in Microbiology. 8, 1-10 (2017).
- Zhang, J. J., Tang, X., Moore, B. S. Genetic platforms for heterologous expression of microbial natural products. Natural Product Reports. 36 (9), 1313-1332 (2019).
- Alduina, R., et al. Artificial chromosome libraries of Streptomyces coelicolor A3(2) and Planobispora rosea. FEMS Microbiology Letters. 218 (1), 181-186 (2003).
- Jones, A. C., et al. Phage P1-Derived Artificial Chromosomes Facilitate Heterologous Expression of the FK506 Gene Cluster. PLoS One. 8 (7), 69319 (2013).
- Gomez-Escribano, J. P., Bibb, M. J. Engineering Streptomyces coelicolor for heterologous expression of secondary metabolite gene clusters. Microbial Biotechnology. 4 (2), 207-215 (2011).
- Cannell, R. J. P. . Natural Products Isolation. , (1998).
- Kersten, R. D., et al. A mass spectrometry-guided genome mining approach for natural product peptidogenomics. Nature Chemical Biology. 7 (11), 794-802 (2011).
- Kersten, R. D., et al. Glycogenomics as a mass spectrometry-guided genome-mining method for microbial glycosylated molecules. Proceedings of the National Academy of Sciences. 110 (47), 4407-4416 (2013).
- Liu, W., et al. MS/MS-based networking and peptidogenomics guided genome mining revealed the stenothricin gene cluster in Streptomyces roseosporus. The Journal of Antibiotics. 67 (1), 99-104 (2014).
- Duncan, K. R., et al. Molecular Networking and Pattern-Based Genome Mining Improves Discovery of Biosynthetic Gene Clusters and their Products from Salinispora Species. Chemistry & Biology. 22 (4), 460-471 (2015).
- Cao, L., et al. MetaMiner: A Scalable Peptidogenomics Approach for Discovery of Ribosomal Peptide Natural Products with Blind Modifications from Microbial Communities. Cell Systems. , (2019).
- Chen, L. -. Y., Cui, H. -. T., Su, C., Bai, F. -. W., Zhao, X. -. Q. Analysis of the complete genome sequence of a marine-derived strain Streptomyces sp. S063 CGMCC 14582 reveals its biosynthetic potential to produce novel anti-complement agents and peptides. PeerJ. 7 (1), 6122 (2019).
- Kim Tiam, S., et al. Insights into the Diversity of Secondary Metabolites of Planktothrix Using a Biphasic Approach Combining Global Genomics and Metabolomics. Toxins. 11 (9), 498 (2019).
- Özakin, S., Ince, E. Genome and metabolome mining of marine obligate Salinispora strains to discover new natural products. Turkish Journal of Biology. 43 (1), 28-36 (2019).
- Trivella, D. B. B., de Felicio, R. The Tripod for Bacterial Natural Product Discovery: Genome Mining, Silent Pathway Induction, and Mass Spectrometry-Based Molecular Networking. mSystems. 3 (2), 00160 (2018).
- Maansson, M., et al. An Integrated Metabolomic and Genomic Mining Workflow To Uncover the Biosynthetic Potential of Bacteria. mSystems. 1 (3), 1-14 (2016).
- Blin, K., Kim, H. U., Medema, M. H., Weber, T. Recent development of antiSMASH and other computational approaches to mine secondary metabolite biosynthetic gene clusters. Briefings in Bioinformatics. 20 (4), 1103-1113 (2019).
- Fisch, K. M. Biosynthesis of natural products by microbial iterative hybrid PKS-NRPS. RSC Advances. 3 (40), 18228-18247 (2013).
- Tatsuno, S., Arakawa, K., Kinashi, H. Analysis of Modular-iterative Mixed Biosynthesis of Lankacidin by Heterologous Expression and Gene Fusion. The Journal of Antibiotics. 60 (11), 700-708 (2007).
- Helfrich, E. J. N., Piel, J. Biosynthesis of polyketides by trans-AT polyketide synthases. Natural Product Reports. 33 (2), 231-316 (2016).

