Mass Spectrometry-Guided Genome Mining come strumento per scoprire nuovi prodotti naturali
Summary
Un protocollo di estrazione del genoma guidato dalla spettrometria di massa è stabilito e descritto qui. Si basa sulle informazioni sulla sequenza genomica e sull’analisi LC-MS/MS e mira a facilitare l’identificazione di molecole da complessi estratti microbici e vegetali.
Abstract
Lo spazio chimico coperto da prodotti naturali è immenso e ampiamente non riconosciuto. Pertanto, sono desiderate metodologie convenienti per eseguire una valutazione ad ampio raggio delle loro funzioni in natura e potenziali benefici umani (ad esempio, per applicazioni di scoperta di farmaci). Questo protocollo descrive la combinazione di estrazione del genoma (GM) e networking molecolare (MN), due approcci contemporanei che corrispondono alle annotazioni con codifica a cluster genico nel sequenziamento dell’intero genoma con le firme della struttura chimica da estratti metabolici grezzi. Questo è il primo passo verso la scoperta di nuove entità naturali. Questi concetti, se applicati insieme, sono qui definiti come estrazione genomica guidata da MS. In questo metodo, i componenti principali sono designati in precedenza (utilizzando MN) e i nuovi candidati strutturalmente correlati sono associati alle annotazioni della sequenza del genoma (utilizzando GM). La combinazione di GM e MN è una strategia redditizia per colpire nuove spinadorsali molecolari o raccogliere profili metabolici al fine di identificare analoghi da composti già noti.
Introduction
Le indagini sul metabolismo secondario spesso consistono nello screening di estratti grezzi per specifiche attività biologiche seguite da purificazione, identificazione e caratterizzazione dei costituenti appartenenti a frazioni attive. Questo processo si è dimostrato efficiente, promuovendo l’isolamento di diverse entità chimiche. Tuttavia, al giorno d’oggi questo è visto come irrealizzabile, principalmente a causa degli alti tassi di riscoperta. Come l’industria farmaceutica rivoluzionato senza conoscenza dei ruoli e le funzioni di metaboliti specializzati, la loro identificazione è stata effettuata in condizioni di laboratorio che non rappresentavano con precisione la natura1. Oggi, c’è una migliore comprensione delle influenze di segnalazione naturale, secrezione, e la presenza della maggior parte degli obiettivi a concentrazioni non rilevabili basse. Inoltre, la regolamentazione del processo aiuterà la comunità accademica e l’industria farmaceutica a trarre vantaggio da queste conoscenze. Ne beneficeranno anche la ricerca che coinvolgerà l’isolamento diretto dei metaboliti legati ai cluster genici biosintetici silenziosi (BCC)2.
In questo contesto, i progressi nel sequenziamento genomico hanno rinnovato l’interesse per i metaboliti dei microrganismi di screening. Questo perché l’analisi delle informazioni genomiche degli ammassi biosintetici scoperti può rivelare geni che codificano nuovi composti non osservati o prodotti in condizioni di laboratorio. Molti progetti o correnti d’aria microbici dell’intero genoma sono oggi disponibili, e il numero è in crescita ogni anno, fornendo enormi prospettive per scoprire nuove molecole bioattive attraverso l’estrazione del genoma3,4.
L’Atlante dei cluster di geni biosintetici è la più grande collezione di cluster genici estratti automaticamente come componente della piattaforma integrata dei genomi microbici del Joint Genome Institute (JGI IMG-ABC)2. Più recentemente, la Minimum Information for Biosynthetic Gene Clusters (MIBiG) Standardization Initiative ha promosso la riannotazione manuale dei BMC, fornendo un set di dati di riferimento altamente curato5. Al giorno d’oggi, sono disponibili molti strumenti per consentire l’estrazione computazionale dei dati genetici e la loro connessione ai metaboliti secondari noti. Sono state inoltre sviluppate diverse strategie per accedere a nuovi prodotti naturali bioattivi (ad esempio, espressione etetologa, cancellazione genica mirata, ricostituzione in vitro, sequenza genomica, screening guidato da isotopi [approccio genomitopico], manipolazione delle autorità di regolamentazione locali e globali, estrazione basata su target di resistenza, estrazione indipendente dalla cultura e, più recentemente, approccio al codiceMS 2,6,7,8,9, 10,11,12,13,14,15).
L’estrazione del genoma come strategia singolare richiede sforzi per annotare un singolo o un piccolo gruppo di molecole; pertanto, permangono lacune nel processo in cui i nuovi composti sono prioritari per l’isolamento e la chiarificazione della struttura. In linea di principio, questi approcci si rivolgono a un solo percorso biosintetico per esperimento, con conseguente rallentamento del tasso di scoperta. In questo senso, l’utilizzo di GM insieme a un approccio di rete molecolare rappresenta un importante progresso per la ricerca sui prodotti naturali14,15.
La versatilità, la precisione e l’elevata sensibilità della spettrometria cromatografica-massa liquida (LC-MS) lo rendono un buon metodo per l’identificazione composta. Attualmente, diverse piattaforme hanno investito algoritmi e suite software per metabolomica non mirata16,17,18,19,20.20 Il nucleo di questi programmi include il rilevamento delle funzionalità (picco di raccolta)21 e l’allineamento di picco, che consente la corrispondenza di funzionalità identiche in un batch di campioni e la ricerca di modelli. Gli algoritmi basati su modelli MS22,,23 confrontano i modelli di frammentazione caratteristici e corrispondono alle somiglianze ms2 generando famiglie molecolari che condividono caratteristiche strutturali. Queste caratteristiche possono quindi essere evidenziate e raggruppate, conferendo la capacità di scoprire rapidamente molecole note e sconosciute da un complesso estratto biologico in tandem MS2,24,25. Pertanto, la SM in tandem è un metodo versatile per ottenere informazioni strutturali su diversi chemiotipi contenuti contemporaneamente in una grande quantità di dati.
L’algoritmo Global Natural Products Social Molecular Networking (GNPS)26 utilizza l’intensità degli ioni di frammento normalizzati per costruire vettori multidimensionali, in cui le somiglianze vengono confrontate utilizzando una funzione coseno. La relazione tra i diversi ioni padre viene tracciata in una rappresentazione del diagramma, in cui ogni frammentazione viene visualizzata come nodo (cerchi) e la correlazione di ogni nodo è definita da uno spigolo (linee). La visualizzazione globale delle molecole provenienti da un’unica fonte è definita come una rete molecolare. Molecole strutturalmente divergenti che si frammentano in modo univoco formeranno il proprio ammasso o costellazione specifica, mentre le molecole correlate si raggruppano. I chemiotipi di clustering consentono l’ipotetica connessione di caratteristiche strutturali simili alle loro origini biosintetiche.
La combinazione di approcci chemiotipo-genotipo e genotipo è potente quando si creano collegamenti bioinformatici tra BCC e i loro piccoli prodotti molecolari27. Pertanto, l’estrazione del genoma guidata dalla MS è un metodo rapido e una strategia a basso consumo di materiali e aiuta a colmare gli ioni dei genitori e le vie biosintetiche rivelate dal WGS di uno o più ceppi in diverse condizioni metaboliche e ambientali.
Il flusso di lavoro di questo protocollo (Figura 1) consiste nell’inserire i dati WGS in una piattaforma di annotazione del cluster di geni biosintetici come antiSMASH28,29,30. Aiuta a stimare la varietà di composti e la classe di composti codificati dal genoma. Deve essere adottata una strategia per colpire un cluster di geni biosintetici che codifica un’entità chimica di interesse, e gli estratti di coltura da un ceppo di tipo selvaggio e/o ceppo eteboloso contenente il BGC possono essere analizzati per generare ioni raggruppati in base alle somiglianze utilizzando GNPS26,31. Di conseguenza, è possibile identificare nuove molecole che si associano al BGC mirato e non sono disponibili nel database (principalmente analoghi sconosciuti, a volte prodotti in elementi bassi). È importante considerare che gli utenti possono contribuire a queste piattaforme e che la disponibilità di bioinformatica e dati MS/MS sta aumentando rapidamente, spingendo a uno sviluppo costante e l’aggiornamento di strumenti e algoritmi computazionali efficaci per guidare connessioni efficienti di estratti complessi con molecole.
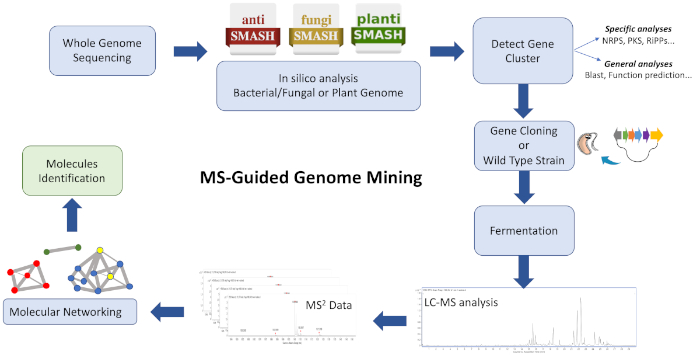
Figura 1: Panoramica dell’intero flusso di lavoro. È mostrato un’illustrazione delle fasi bioinformatiche, clonazione e networking molecolare coinvolte nell’approccio descritto nell’estrazione del genoma guidato dalla Ms per identificare nuovi metaboliti. Fare clic qui per visualizzare una versione più grande di questa figura.
Questo protocollo descrive un flusso di lavoro rapido ed efficiente per combinare l’estrazione del genoma e la rete molecolare come punto di partenza per la pipeline di scoperta naturale dei prodotti. Anche se molte applicazioni sono in grado di visualizzare la composizione e la relatedness delle molecole rilevabili dalla MS in una rete, diverse sono adottate qui per visualizzare molecole raggruppate strutturalmente simili. Utilizzando questa strategia, vengono identificati con successo nuovi prodotti ciclodepsipeptide osservati negli estratti metabolici di Streptomyces sp. CBMAI 2042. Guidato dall’estrazione del genoma, l’intera codifica del cluster genico biosintetico per le valiomiciche è riconosciuta e clonata nel ceppo produttore Streptomices coelicolor M1146. Infine, a seguito di una rete molecolare basata su modelli di SM, le molecole rilevate dalla SM sono correlate con i BMC responsabili della loro biogenesi32.
Protocol
Representative Results
Discussion
Il vantaggio più forte di questo protocollo è la sua capacità di dereplicare rapidamente i profili metabolici e le informazioni genomiche del ponte con i dati MS al fine di chiarire le strutture di nuove molecole, in particolare analoghi strutturali2. Sulla base di informazioni genomiche, è possibile studiare chemiotipi di prodotti naturali diversi, come i polionidi (PK), i peptidi non ribonsuici (NRP) e i prodotti naturali glicosillati (GNP), nonché gli screening metabolomici criptici. Così…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Il sostegno finanziario a questo studio è stato fornito dalla Fondazione ricerca di San Paolo – FAPESP (2019/10564-5, 2014/12727-5 e 2014/50249-8 a L.G.O; 2013/12598-8 e 2015/01013-4 a R.S.; e 2019/08853-9 a C.F.F.A). B.S.P, C.F.F.A. e L.G.O. hanno ricevuto borse di studio dal Consiglio nazionale per lo sviluppo scientifico e tecnologico – CNPq (205729/2018-5, 162191/2015-4 e 313492/2017-4). L.G.O. è anche grato per il sostegno alla borsa di studio fornito dal programma For Women in Science (2008, edizione brasiliana). Tutti gli autori riconoscono CAPES (Coordinamento per il Miglioramento del Personale per l’Istruzione Superiore) per aver sostenuto i programmi post-laurea in Brasile.
Materials
| Acetonitrile | Tedia | AA1120-048 | HPLC grade |
| Agar | Oxoid | LP0011 | NA |
| Apramycin | Sigma Aldrich | A2024 | NA |
| Carbenicillin | Sigma Aldrich | C9231 | NA |
| Centrifuge | Eppendorf | NA | 5804 |
| Chloramphenicol | Sigma Aldrich | C3175 | NA |
| Column C18 | Agilent Technologies | NA | ZORBAX RRHD Extend-C18, 80Å, 2.1 x 50 mm, 1.8 µm, 1200 bar pressure limit P/N 757700-902 |
| Kanamycin | Sigma Aldrich | K1377 | NA |
| Manitol P.A.- A.C.S. | Synth | NA | NA |
| Microcentrifuge | Eppendorf | NA | 5418 |
| Nalidixic acid | Sigma Aldrich | N4382 | NA |
| Phusion Flash High-Fidelity PCR Master Mix | ThermoFisher Scientific | F548S | NA |
| Q-TOF mass spectrometer | Agilent technologies | NA | 6550 iFunnel Q-TOF LC/MS |
| Sacarose P.A.- A.C.S. | Synth | NA | NA |
| Shaker/Incubator | Marconi | MA420 | NA |
| Sodium Chloride | Synth | NA | P. A. – ACS |
| Soy extract | NA | NA | NA |
| Sucrose | Synth | NA | P. A. – ACS |
| Thermal Cycles | Eppendorf | NA | Mastercycler Nexus Gradient |
| Thiostrepton | Sigma Aldrich | T8902 | NA |
| Tryptone | Oxoid | LP0042 | NA |
| Tryptone Soy Broth | Oxoid | CM0129 | NA |
| UPLC | Agilent Technologies | NA | 1290 Infinity LC System |
| Yeast extract | Oxoid | LP0021 | NA |
References
- Davies, J. Specialized microbial metabolites: functions and origins. The Journal of Antibiotics. 66 (7), 361-364 (2013).
- Ziemert, N., Alanjary, M., Weber, T. The evolution of genome mining in microbes – a review. Natural Product Reports. 33 (8), 988-1005 (2016).
- Zerikly, M., Challis, G. L. Strategies for the Discovery of New Natural Products by Genome Mining. ChemBioChem. 10 (4), 625-633 (2009).
- Gomez-Escribano, J. P., Bibb, M. J. Heterologous expression of natural product biosynthetic gene clusters in Streptomyces coelicolor: from genome mining to manipulation of biosynthetic pathways. Journal of Industrial Microbiology & Biotechnology. 41 (2), 425-431 (2014).
- Medema, M. H., et al. Minimum Information about a Biosynthetic Gene cluster. Nature Chemical Biology. 11 (9), 625-631 (2015).
- Lautru, S., Deeth, R. J., Bailey, L. M., Challis, G. L. Discovery of a new peptide natural product by Streptomyces coelicolor genome mining. Nature Chemical Biology. 1 (5), 265-269 (2005).
- Chiang, Y. -. M., et al. Molecular Genetic Mining of the Aspergillus Secondary Metabolome: Discovery of the Emericellamide Biosynthetic Pathway. Chemistry & Biology. 15 (6), 527-532 (2008).
- Huang, T., et al. Identification and Characterization of the Pyridomycin Biosynthetic Gene Cluster of Streptomyces pyridomyceticus NRRL B-2517. Journal of Biological Chemistry. 286 (23), 20648-20657 (2011).
- Udwary, D. W., et al. Genome sequencing reveals complex secondary metabolome in the marine actinomycete Salinispora tropica. Proceedings of the National Academy of Sciences. 104 (25), 10376-10381 (2007).
- Gross, H., et al. The Genomisotopic Approach: A Systematic Method to Isolate Products of Orphan Biosynthetic Gene Clusters. Chemistry & Biology. 14 (1), 53-63 (2007).
- Spohn, M., Wohlleben, W., Stegmann, E. Elucidation of the zinc-dependent regulation in Amycolatopsis japonicum enabled the identification of the ethylenediamine-disuccinate ([S,S ]-EDDS) genes. Environmental Microbiology. 18 (4), 1249-1263 (2016).
- Thaker, M. N., Waglechner, N., Wright, G. D. Antibiotic resistance-mediated isolation of scaffold-specific natural product producers. Nature Protocols. 9 (6), 1469-1479 (2014).
- Katz, M., Hover, B. M., Brady, S. F. Culture-independent discovery of natural products from soil metagenomes. Journal of Industrial Microbiology & Biotechnology. 43, 129-141 (2016).
- Quinn, R. A., et al. Molecular Networking as a Drug Discovery, Drug Metabolism, and Precision Medicine Strategy. Trends in Pharmacological Sciences. 38 (2), 143-154 (2017).
- Yang, J. Y., et al. Molecular Networking as a Dereplication Strategy. Journal of Natural Products. 76 (9), 1686-1699 (2013).
- Lommen, A. MetAlign: Interface-Driven, Versatile Metabolomics Tool for Hyphenated Full-Scan Mass Spectrometry Data Preprocessing. Analytical Chemistry. 81 (8), 3079-3086 (2009).
- Katajamaa, M., Miettinen, J., Oresic, M. MZmine: toolbox for processing and visualization of mass spectrometry based molecular profile data. Bioinformatics. 22 (5), 634-636 (2006).
- Pluskal, T., Castillo, S., Villar-Briones, A., Orešič, M. MZmine 2: Modular framework for processing, visualizing, and analyzing mass spectrometry-based molecular profile data. BMC Bioinformatics. 11 (1), 395 (2010).
- Tautenhahn, R., Patti, G. J., Rinehart, D., Siuzdak, G. XCMS Online: A Web-Based Platform to Process Untargeted Metabolomic Data. Analytical Chemistry. 84 (11), 5035-5039 (2012).
- Kuhl, C., Tautenhahn, R., Böttcher, C., Larson, T. R., Neumann, S. CAMERA: An Integrated Strategy for Compound Spectra Extraction and Annotation of Liquid Chromatography/Mass Spectrometry Data Sets. Analytical Chemistry. 84 (1), 283-289 (2012).
- Katajamaa, M., Orešič, M. Data processing for mass spectrometry-based metabolomics. Journal of Chromatography A. 1158, 318-328 (2007).
- Liu, W. -. T., et al. Interpretation of Tandem Mass Spectra Obtained from Cyclic Nonribosomal Peptides. Analytical Chemistry. 81 (11), 4200-4209 (2009).
- Ng, J., et al. Dereplication and de novo sequencing of nonribosomal peptides. Nature Methods. 6 (8), 596-599 (2009).
- Liaw, C., et al. Vitroprocines, new antibiotics against Acinetobacter baumannii, discovered from marine Vibrio sp. QWI-06 using mass-spectrometry-based metabolomics approach. Scientific Reports. 5 (1), 1-11 (2015).
- Kang, K. B., et al. Targeted Isolation of Neuroprotective Dicoumaroyl Neolignans and Lignans from Sageretia theezans Using in Silico Molecular Network Annotation Propagation-Based Dereplication. Journal of Natural Products. 81 (8), 1819-1828 (2018).
- Wang, M., et al. Sharing and community curation of mass spectrometry data with Global Natural Products Social Molecular Networking. Nature Biotechnology. 34 (8), 828-837 (2016).
- Doroghazi, J. R., et al. A roadmap for natural product discovery based on large-scale genomics and metabolomics. Nature Chemical Biology. 10 (11), 963-968 (2014).
- Medema, M. H., et al. antiSMASH: rapid identification, annotation and analysis of secondary metabolite biosynthesis gene clusters in bacterial and fungal genome sequences. Nucleic Acids Research. 39, 339-346 (2011).
- Weber, T., et al. antiSMASH 3.0-a comprehensive resource for the genome mining of biosynthetic gene clusters. Nucleic Acids Research. 43, 237-243 (2015).
- Blin, K., et al. antiSMASH 5.0: updates to the secondary metabolite genome mining pipeline. Nucleic Acids Research. 47, 81-87 (2019).
- Watrous, J., et al. Mass spectral molecular networking of living microbial colonies. Proceedings of the National Academy of Sciences. 109 (26), 1743-1752 (2012).
- Paulo, B. S., Sigrist, R., Angolini, C. F. F., De Oliveira, L. G. New Cyclodepsipeptide Derivatives Revealed by Genome Mining and Molecular Networking. ChemistrySelect. 4 (27), 7785-7790 (2019).
- Gonzaga de Oliveira, L., Sigrist, R., Sachetto Paulo, B., Samborskyy, M. Whole-Genome Sequence of the Endophytic Streptomyces sp. Strain CBMAI 2042, Isolated from Citrus sinensis. Microbiology Resource Announcements. 8 (2), 1-2 (2019).
- Aziz, R. K., et al. The RAST Server: Rapid Annotations using Subsystems Technology. BMC Genomics. 9 (1), 75 (2008).
- Nah, H. -. J., Pyeon, H. -. R., Kang, S. -. H., Choi, S. -. S., Kim, E. -. S. Cloning and Heterologous Expression of a Large-sized Natural Product Biosynthetic Gene Cluster in Streptomyces Species. Frontiers in Microbiology. 8, 1-10 (2017).
- Zhang, J. J., Tang, X., Moore, B. S. Genetic platforms for heterologous expression of microbial natural products. Natural Product Reports. 36 (9), 1313-1332 (2019).
- Alduina, R., et al. Artificial chromosome libraries of Streptomyces coelicolor A3(2) and Planobispora rosea. FEMS Microbiology Letters. 218 (1), 181-186 (2003).
- Jones, A. C., et al. Phage P1-Derived Artificial Chromosomes Facilitate Heterologous Expression of the FK506 Gene Cluster. PLoS One. 8 (7), 69319 (2013).
- Gomez-Escribano, J. P., Bibb, M. J. Engineering Streptomyces coelicolor for heterologous expression of secondary metabolite gene clusters. Microbial Biotechnology. 4 (2), 207-215 (2011).
- Cannell, R. J. P. . Natural Products Isolation. , (1998).
- Kersten, R. D., et al. A mass spectrometry-guided genome mining approach for natural product peptidogenomics. Nature Chemical Biology. 7 (11), 794-802 (2011).
- Kersten, R. D., et al. Glycogenomics as a mass spectrometry-guided genome-mining method for microbial glycosylated molecules. Proceedings of the National Academy of Sciences. 110 (47), 4407-4416 (2013).
- Liu, W., et al. MS/MS-based networking and peptidogenomics guided genome mining revealed the stenothricin gene cluster in Streptomyces roseosporus. The Journal of Antibiotics. 67 (1), 99-104 (2014).
- Duncan, K. R., et al. Molecular Networking and Pattern-Based Genome Mining Improves Discovery of Biosynthetic Gene Clusters and their Products from Salinispora Species. Chemistry & Biology. 22 (4), 460-471 (2015).
- Cao, L., et al. MetaMiner: A Scalable Peptidogenomics Approach for Discovery of Ribosomal Peptide Natural Products with Blind Modifications from Microbial Communities. Cell Systems. , (2019).
- Chen, L. -. Y., Cui, H. -. T., Su, C., Bai, F. -. W., Zhao, X. -. Q. Analysis of the complete genome sequence of a marine-derived strain Streptomyces sp. S063 CGMCC 14582 reveals its biosynthetic potential to produce novel anti-complement agents and peptides. PeerJ. 7 (1), 6122 (2019).
- Kim Tiam, S., et al. Insights into the Diversity of Secondary Metabolites of Planktothrix Using a Biphasic Approach Combining Global Genomics and Metabolomics. Toxins. 11 (9), 498 (2019).
- Özakin, S., Ince, E. Genome and metabolome mining of marine obligate Salinispora strains to discover new natural products. Turkish Journal of Biology. 43 (1), 28-36 (2019).
- Trivella, D. B. B., de Felicio, R. The Tripod for Bacterial Natural Product Discovery: Genome Mining, Silent Pathway Induction, and Mass Spectrometry-Based Molecular Networking. mSystems. 3 (2), 00160 (2018).
- Maansson, M., et al. An Integrated Metabolomic and Genomic Mining Workflow To Uncover the Biosynthetic Potential of Bacteria. mSystems. 1 (3), 1-14 (2016).
- Blin, K., Kim, H. U., Medema, M. H., Weber, T. Recent development of antiSMASH and other computational approaches to mine secondary metabolite biosynthetic gene clusters. Briefings in Bioinformatics. 20 (4), 1103-1113 (2019).
- Fisch, K. M. Biosynthesis of natural products by microbial iterative hybrid PKS-NRPS. RSC Advances. 3 (40), 18228-18247 (2013).
- Tatsuno, S., Arakawa, K., Kinashi, H. Analysis of Modular-iterative Mixed Biosynthesis of Lankacidin by Heterologous Expression and Gene Fusion. The Journal of Antibiotics. 60 (11), 700-708 (2007).
- Helfrich, E. J. N., Piel, J. Biosynthesis of polyketides by trans-AT polyketide synthases. Natural Product Reports. 33 (2), 231-316 (2016).

