Mass Spectrometry-Guided Genome Mining als een instrument om nieuwe natuurlijke producten te ontdekken
Summary
Een massaspectrometrie-geleide genoom mijnbouw protocol is hier vastgesteld en beschreven. Het is gebaseerd op genoomsequentie-informatie en LC-MS/MS-analyse en heeft tot doel de identificatie van moleculen uit complexe microbiële en plantenextracten te vergemakkelijken.
Abstract
De chemische ruimte bedekt met natuurlijke producten is immens en op grote schaal niet herkend. Daarom zijn handige methoden om hun functies in de natuur en potentiële menselijke voordelen (bijvoorbeeld voor toepassingen voor medicijnontdekking) te evalueren gewenst. Dit protocol beschrijft de combinatie van genoommijnbouw (GM) en moleculaire netwerken (MN), twee hedendaagse benaderingen die gencluster-gecodeerde annotaties in hele genoomsequencing matchen met chemische structuurhandtekeningen van ruwe metabole extracten. Dit is de eerste stap in de richting van de ontdekking van nieuwe natuurlijke entiteiten. Deze concepten, wanneer ze samen worden toegepast, worden hier gedefinieerd als MS-geleide genoommijnbouw. In deze methode worden de belangrijkste componenten eerder aangewezen (met behulp van MN), en structureel gerelateerde nieuwe kandidaten worden geassocieerd met genoomsequentieannotaties (met behulp van GM). Het combineren van GM en MN is een winstgevende strategie om nieuwe molecuulbackbones aan te pakken of metabole profielen te oogsten om analogen van reeds bekende verbindingen te identificeren.
Introduction
Onderzoeken van secundair metabolisme bestaan vaak uit het screenen van ruwe extracten voor specifieke biologische activiteiten, gevolgd door zuivering, identificatie en karakterisering van de bestanddelen die behoren tot actieve fracties. Dit proces is efficiënt gebleken en heeft de isolatie van verschillende chemische entiteiten bevorderd. Tegenwoordig wordt dit echter als onhaalbaar gezien, vooral vanwege de hoge herontdekkingspercentages. Aangezien de farmaceutische industrie zonder kennis van de rollen en de functies van gespecialiseerde metabolieten een revolutie teweegbracht, werd hun identificatie uitgevoerd onder laboratoriumomstandigheden die niet nauwkeurig aardvertegenwoordigen 1. Vandaag de dag is er een beter begrip van natuurlijke signalering seinen invloeden, afscheiding, en de aanwezigheid van de meeste doelen bij niet op te sporen lage concentraties. Bovendien zal de regulering van het proces de academische gemeenschap en de farmaceutische industrie helpen om van deze kennis gebruik te maken. Het zal ook ten goede komen aan onderzoek naar directe isolatie van metabolieten in verband met stille biosynthetische genclusters (BGC’s)2.
In deze context hebben de vooruitgang in genomische sequencing hernieuwde interesse in het screenen van micro-organismen metabolieten. Dit komt omdat het analyseren van de genomische informatie van onbedekte biosynthetische clusters kan onthullen genen coderen nieuwe verbindingen niet waargenomen of geproduceerd onder laboratoriumomstandigheden. Veel microbiële hele genoom projecten of ontwerpen zijn beschikbaar vandaag, en het aantal groeit elk jaar, het verstrekken van enorme vooruitzichten voor het blootleggen van nieuwe bioactieve moleculen door middel van genoom mijnbouw3,4.
De Atlas van Biosynthetische Gen Clusters is de huidige grootste verzameling van automatisch gedolzegenclusters als onderdeel van het Integrated Microbial Genomes Platform van het Joint Genome Institute (JGI IMG-ABC)2. Meest recent heeft het Minimum Information for Biosynthetic Gene Clusters (MIBiG) Standardization Initiative de handmatige reannotatie van BGC’s bevorderd, met een sterk samengestelde referentiedataset5. Tegenwoordig zijn er tal van instrumenten beschikbaar om computationele mijnbouw van genetische gegevens en hun verbinding met bekende secundaire metabolieten mogelijk te maken. Er zijn ook verschillende strategieën ontwikkeld om toegang te krijgen tot nieuwe bioactieve natuurlijke producten (d.w.z. heterologe expressie, doelgenverwijdering, in vitro reconstructie, genomische sequentie, isotopengeleide screening [genomisotopische benadering], manipulatie van lokale en mondiale regelgevers, op resistentie gebaseerde mijnbouw, cultuuronafhankelijke mijnbouw en, meer recentelijk, MS-geleide/codebenaderingen2,6,7,8,9, 10,11,12,13,14,15).
Genoommining als enkelvoudstrategie vereist inspanningen om een enkele of kleine groep moleculen te annoteren; er blijven dus hiaten in het proces bestaan waarin nieuwe verbindingen prioriteit krijgen voor isolatie en structuuropheldering. In principe zijn deze benaderingen slechts gericht op één biosynthetisch traject per experiment, wat resulteert in een langzame ontdekkingssnelheid. In die zin is het gebruik van GM samen met een moleculaire netwerkbenadering een belangrijke vooruitgang voor het onderzoek naar natuurlijke producten14,15.
De veelzijdigheid, nauwkeurigheid en hoge gevoeligheid van vloeibare chromatografie-massaspectrometrie (LC-MS) maken het een goede methode voor samengestelde identificatie. Momenteel hebben verschillende platforms geïnvesteerd algoritmen en software suites voor ongerichte metabolomics16,17,18,19,20. De kern van deze programma’s omvat functiedetectie (peak picking)21 en piekuitlijning, waardoor identieke functies in een batch van monsters kunnen worden gematcht en naar patronen wordt gezocht. MS-patroongebaseerde algoritmen22,23 vergelijken karakteristieke fragmentatiepatronen en matchen MS2-overeenkomsten die moleculaire families genereren die structurele kenmerken delen. Deze kenmerken kunnen vervolgens worden gemarkeerd en geclusterd, waardoor de mogelijkheid om snel te ontdekken bekende en onbekende moleculen uit een complex biologisch extract door tandem MS2,24,25. Daarom is tandem MS een veelzijdige methode om structurele informatie te verkrijgen over verschillende chemotypes in een grote hoeveelheid gegevens tegelijk.
Het Global Natural Products Social Molecular Networking (GNPS)26 algoritme gebruikt de genormaliseerde fragmentionenintensiteit om multidimensionale vectoren te construeren, waarbij gelijkenissen worden vergeleken met behulp van een cosinefunctie. De relatie tussen verschillende bovenliggende ionen wordt uitgezet in een diagramweergave, waarbij elke fragmentatie wordt gevisualiseerd als een knooppunt (cirkels) en de verwantheid van elk knooppunt wordt gedefinieerd door een rand (lijnen). De globale visualisatie van moleculen uit één bron wordt gedefinieerd als een moleculair netwerk. Structureel uiteenlopende moleculen die uniek fragmenteren, vormen hun eigen specifieke cluster of constellatie, terwijl verwante moleculen samenclusteren. Clustering chemotypes maakt het mogelijk de hypothetische verbinding van soortgelijke structurele kenmerken aan hun biosynthetische oorsprong.
Het combineren van zowel chemotype-to-genotype en genotype-to-chemotype benaderingen is krachtig bij het creëren van bio-informatica verbindingen tussen BGCs en hun kleine molecuul producten27. Daarom is MS-geleide genoommijnbouw een snelle methode en een lage materiaalverbruikende strategie, en het helpt bij het overbruggen van ouderionen en biosynthetische trajecten die door WGS van een of meer stammen onder diverse metabolische en milieuomstandigheden worden onthuld.
De workflow van dit protocol (figuur 1) bestaat uit het voeden van WGS-gegevens in een biosynthetisch genclusterannotatieplatform zoals antiSMASH28,29,30. Het helpt bij het schatten van de verscheidenheid van verbindingen en de klasse van verbindingen gecodeerd door het genoom. Er moet een strategie worden aangenomen om zich te richten op een biosynthetisch gencluster dat een chemische entiteit van belang codeert, en kweekextracten van een wilde soortstam en/of heterologe stam die de BGC bevat, kunnen worden geanalyseerd om geclusterde ionen te genereren op basis van gelijkenissen met GNPS26,31. Bijgevolg is het mogelijk om nieuwe moleculen te identificeren die zich associëren met de beoogde BGC en niet beschikbaar zijn in de database (voornamelijk onbekende analogen, soms geproduceerd in lage titers). Het is relevant om te bedenken dat gebruikers kunnen bijdragen aan deze platforms en dat de beschikbaarheid van bioinformatica en MS/MS-gegevens snel toeneemt, wat leidt tot een constante ontwikkeling en upgrade van effectieve computationele tools en algoritmen om efficiënte verbindingen van complexe extracten met moleculen te begeleiden.
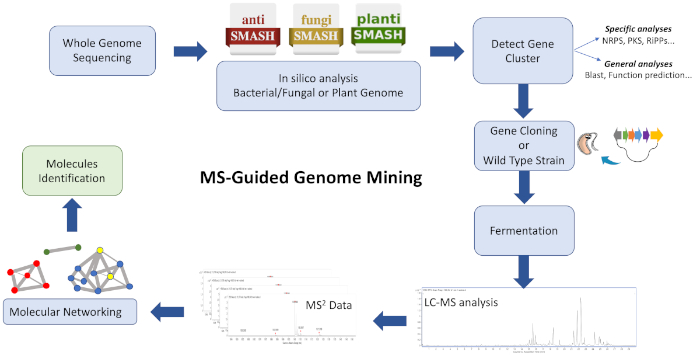
Figuur 1: Overzicht van de gehele werkstroom. Getoond is een illustratie van de bio-informatica, klonen, en moleculaire netwerkstappen die betrokken zijn bij de beschreven MS-geleide genoom mijnbouw aanpak om nieuwe metabolieten te identificeren. Klik hier om een grotere versie van dit cijfer te bekijken.
Dit protocol beschrijft een snelle en efficiënte workflow om genoommining en moleculaire netwerken te combineren als uitgangspunt voor de pijplijn voor de ontdekking van natuurlijke producten. Hoewel veel toepassingen in staat zijn om de samenstelling en verwantschap van MS-detecteerbare moleculen in één netwerk te visualiseren, worden er hier verschillende aangenomen om structureel vergelijkbare geclusterde moleculen te visualiseren. Met behulp van deze strategie, nieuwe cyclodepsipeptide producten waargenomen in metabole extracten van Streptomyces sp. CBMAI 2042 zijn met succes geïdentificeerd. Geleid door genoommijnbouw wordt de hele biosynthetische genclustercodering voor valinomycines erkend en gekloond in de producentenstam Streptomyces coelicolor M1146. Ten slotte, na een MS-patroon gebaseerde moleculaire netwerken, de moleculen gedetecteerd door MS zijn gecorreleerd met BGCs die verantwoordelijk zijn voor hun biogenese32.
Protocol
Representative Results
Discussion
Het sterkste voordeel van dit protocol is het vermogen om metabole profielen snel te derepliceren en genomische informatie te overbruggen met MS-gegevens om de structuren van nieuwe moleculen, met name structureleanalogen,op te helderen 2 . Op basis van genomische informatie kunnen verschillende natuurlijke producten worden onderzocht, zoals polyketides (PK), nonribosomal peptiden (NRP) en glycosylated natural products (BNP), evenals cryptische BGCs. Metabolomic screening levert bewijs van geactiv…
Disclosures
The authors have nothing to disclose.
Acknowledgements
De financiële steun voor deze studie werd verstrekt door São Paulo Research Foundation – FAPESP (2019/10564-5, 2014/12727-5 en 2014/50249-8 tot L.G.O; 2013/12598-8 en 2015/01013-4 naar R.S.; en 2019/08853-9 naar C.F.F.A). B.S.P, C.F.F.A. en L.G.O. ontvingen beurzen van de Nationale Raad voor Wetenschappelijke en Technologische Ontwikkeling – CNPq (205729/2018-5, 162191/2015-4 en 313492/2017-4). L.G.O. is ook dankbaar voor de subsidiesteun van het programma For Women in Science (2008, Brazilian Edition). Alle auteurs erkennen CAPES (Coördinatie voor de verbetering van het hoger onderwijs Personeel) voor de ondersteuning van de post-afstuderen programma’s in Brazilië.
Materials
| Acetonitrile | Tedia | AA1120-048 | HPLC grade |
| Agar | Oxoid | LP0011 | NA |
| Apramycin | Sigma Aldrich | A2024 | NA |
| Carbenicillin | Sigma Aldrich | C9231 | NA |
| Centrifuge | Eppendorf | NA | 5804 |
| Chloramphenicol | Sigma Aldrich | C3175 | NA |
| Column C18 | Agilent Technologies | NA | ZORBAX RRHD Extend-C18, 80Å, 2.1 x 50 mm, 1.8 µm, 1200 bar pressure limit P/N 757700-902 |
| Kanamycin | Sigma Aldrich | K1377 | NA |
| Manitol P.A.- A.C.S. | Synth | NA | NA |
| Microcentrifuge | Eppendorf | NA | 5418 |
| Nalidixic acid | Sigma Aldrich | N4382 | NA |
| Phusion Flash High-Fidelity PCR Master Mix | ThermoFisher Scientific | F548S | NA |
| Q-TOF mass spectrometer | Agilent technologies | NA | 6550 iFunnel Q-TOF LC/MS |
| Sacarose P.A.- A.C.S. | Synth | NA | NA |
| Shaker/Incubator | Marconi | MA420 | NA |
| Sodium Chloride | Synth | NA | P. A. – ACS |
| Soy extract | NA | NA | NA |
| Sucrose | Synth | NA | P. A. – ACS |
| Thermal Cycles | Eppendorf | NA | Mastercycler Nexus Gradient |
| Thiostrepton | Sigma Aldrich | T8902 | NA |
| Tryptone | Oxoid | LP0042 | NA |
| Tryptone Soy Broth | Oxoid | CM0129 | NA |
| UPLC | Agilent Technologies | NA | 1290 Infinity LC System |
| Yeast extract | Oxoid | LP0021 | NA |
References
- Davies, J. Specialized microbial metabolites: functions and origins. The Journal of Antibiotics. 66 (7), 361-364 (2013).
- Ziemert, N., Alanjary, M., Weber, T. The evolution of genome mining in microbes – a review. Natural Product Reports. 33 (8), 988-1005 (2016).
- Zerikly, M., Challis, G. L. Strategies for the Discovery of New Natural Products by Genome Mining. ChemBioChem. 10 (4), 625-633 (2009).
- Gomez-Escribano, J. P., Bibb, M. J. Heterologous expression of natural product biosynthetic gene clusters in Streptomyces coelicolor: from genome mining to manipulation of biosynthetic pathways. Journal of Industrial Microbiology & Biotechnology. 41 (2), 425-431 (2014).
- Medema, M. H., et al. Minimum Information about a Biosynthetic Gene cluster. Nature Chemical Biology. 11 (9), 625-631 (2015).
- Lautru, S., Deeth, R. J., Bailey, L. M., Challis, G. L. Discovery of a new peptide natural product by Streptomyces coelicolor genome mining. Nature Chemical Biology. 1 (5), 265-269 (2005).
- Chiang, Y. -. M., et al. Molecular Genetic Mining of the Aspergillus Secondary Metabolome: Discovery of the Emericellamide Biosynthetic Pathway. Chemistry & Biology. 15 (6), 527-532 (2008).
- Huang, T., et al. Identification and Characterization of the Pyridomycin Biosynthetic Gene Cluster of Streptomyces pyridomyceticus NRRL B-2517. Journal of Biological Chemistry. 286 (23), 20648-20657 (2011).
- Udwary, D. W., et al. Genome sequencing reveals complex secondary metabolome in the marine actinomycete Salinispora tropica. Proceedings of the National Academy of Sciences. 104 (25), 10376-10381 (2007).
- Gross, H., et al. The Genomisotopic Approach: A Systematic Method to Isolate Products of Orphan Biosynthetic Gene Clusters. Chemistry & Biology. 14 (1), 53-63 (2007).
- Spohn, M., Wohlleben, W., Stegmann, E. Elucidation of the zinc-dependent regulation in Amycolatopsis japonicum enabled the identification of the ethylenediamine-disuccinate ([S,S ]-EDDS) genes. Environmental Microbiology. 18 (4), 1249-1263 (2016).
- Thaker, M. N., Waglechner, N., Wright, G. D. Antibiotic resistance-mediated isolation of scaffold-specific natural product producers. Nature Protocols. 9 (6), 1469-1479 (2014).
- Katz, M., Hover, B. M., Brady, S. F. Culture-independent discovery of natural products from soil metagenomes. Journal of Industrial Microbiology & Biotechnology. 43, 129-141 (2016).
- Quinn, R. A., et al. Molecular Networking as a Drug Discovery, Drug Metabolism, and Precision Medicine Strategy. Trends in Pharmacological Sciences. 38 (2), 143-154 (2017).
- Yang, J. Y., et al. Molecular Networking as a Dereplication Strategy. Journal of Natural Products. 76 (9), 1686-1699 (2013).
- Lommen, A. MetAlign: Interface-Driven, Versatile Metabolomics Tool for Hyphenated Full-Scan Mass Spectrometry Data Preprocessing. Analytical Chemistry. 81 (8), 3079-3086 (2009).
- Katajamaa, M., Miettinen, J., Oresic, M. MZmine: toolbox for processing and visualization of mass spectrometry based molecular profile data. Bioinformatics. 22 (5), 634-636 (2006).
- Pluskal, T., Castillo, S., Villar-Briones, A., Orešič, M. MZmine 2: Modular framework for processing, visualizing, and analyzing mass spectrometry-based molecular profile data. BMC Bioinformatics. 11 (1), 395 (2010).
- Tautenhahn, R., Patti, G. J., Rinehart, D., Siuzdak, G. XCMS Online: A Web-Based Platform to Process Untargeted Metabolomic Data. Analytical Chemistry. 84 (11), 5035-5039 (2012).
- Kuhl, C., Tautenhahn, R., Böttcher, C., Larson, T. R., Neumann, S. CAMERA: An Integrated Strategy for Compound Spectra Extraction and Annotation of Liquid Chromatography/Mass Spectrometry Data Sets. Analytical Chemistry. 84 (1), 283-289 (2012).
- Katajamaa, M., Orešič, M. Data processing for mass spectrometry-based metabolomics. Journal of Chromatography A. 1158, 318-328 (2007).
- Liu, W. -. T., et al. Interpretation of Tandem Mass Spectra Obtained from Cyclic Nonribosomal Peptides. Analytical Chemistry. 81 (11), 4200-4209 (2009).
- Ng, J., et al. Dereplication and de novo sequencing of nonribosomal peptides. Nature Methods. 6 (8), 596-599 (2009).
- Liaw, C., et al. Vitroprocines, new antibiotics against Acinetobacter baumannii, discovered from marine Vibrio sp. QWI-06 using mass-spectrometry-based metabolomics approach. Scientific Reports. 5 (1), 1-11 (2015).
- Kang, K. B., et al. Targeted Isolation of Neuroprotective Dicoumaroyl Neolignans and Lignans from Sageretia theezans Using in Silico Molecular Network Annotation Propagation-Based Dereplication. Journal of Natural Products. 81 (8), 1819-1828 (2018).
- Wang, M., et al. Sharing and community curation of mass spectrometry data with Global Natural Products Social Molecular Networking. Nature Biotechnology. 34 (8), 828-837 (2016).
- Doroghazi, J. R., et al. A roadmap for natural product discovery based on large-scale genomics and metabolomics. Nature Chemical Biology. 10 (11), 963-968 (2014).
- Medema, M. H., et al. antiSMASH: rapid identification, annotation and analysis of secondary metabolite biosynthesis gene clusters in bacterial and fungal genome sequences. Nucleic Acids Research. 39, 339-346 (2011).
- Weber, T., et al. antiSMASH 3.0-a comprehensive resource for the genome mining of biosynthetic gene clusters. Nucleic Acids Research. 43, 237-243 (2015).
- Blin, K., et al. antiSMASH 5.0: updates to the secondary metabolite genome mining pipeline. Nucleic Acids Research. 47, 81-87 (2019).
- Watrous, J., et al. Mass spectral molecular networking of living microbial colonies. Proceedings of the National Academy of Sciences. 109 (26), 1743-1752 (2012).
- Paulo, B. S., Sigrist, R., Angolini, C. F. F., De Oliveira, L. G. New Cyclodepsipeptide Derivatives Revealed by Genome Mining and Molecular Networking. ChemistrySelect. 4 (27), 7785-7790 (2019).
- Gonzaga de Oliveira, L., Sigrist, R., Sachetto Paulo, B., Samborskyy, M. Whole-Genome Sequence of the Endophytic Streptomyces sp. Strain CBMAI 2042, Isolated from Citrus sinensis. Microbiology Resource Announcements. 8 (2), 1-2 (2019).
- Aziz, R. K., et al. The RAST Server: Rapid Annotations using Subsystems Technology. BMC Genomics. 9 (1), 75 (2008).
- Nah, H. -. J., Pyeon, H. -. R., Kang, S. -. H., Choi, S. -. S., Kim, E. -. S. Cloning and Heterologous Expression of a Large-sized Natural Product Biosynthetic Gene Cluster in Streptomyces Species. Frontiers in Microbiology. 8, 1-10 (2017).
- Zhang, J. J., Tang, X., Moore, B. S. Genetic platforms for heterologous expression of microbial natural products. Natural Product Reports. 36 (9), 1313-1332 (2019).
- Alduina, R., et al. Artificial chromosome libraries of Streptomyces coelicolor A3(2) and Planobispora rosea. FEMS Microbiology Letters. 218 (1), 181-186 (2003).
- Jones, A. C., et al. Phage P1-Derived Artificial Chromosomes Facilitate Heterologous Expression of the FK506 Gene Cluster. PLoS One. 8 (7), 69319 (2013).
- Gomez-Escribano, J. P., Bibb, M. J. Engineering Streptomyces coelicolor for heterologous expression of secondary metabolite gene clusters. Microbial Biotechnology. 4 (2), 207-215 (2011).
- Cannell, R. J. P. . Natural Products Isolation. , (1998).
- Kersten, R. D., et al. A mass spectrometry-guided genome mining approach for natural product peptidogenomics. Nature Chemical Biology. 7 (11), 794-802 (2011).
- Kersten, R. D., et al. Glycogenomics as a mass spectrometry-guided genome-mining method for microbial glycosylated molecules. Proceedings of the National Academy of Sciences. 110 (47), 4407-4416 (2013).
- Liu, W., et al. MS/MS-based networking and peptidogenomics guided genome mining revealed the stenothricin gene cluster in Streptomyces roseosporus. The Journal of Antibiotics. 67 (1), 99-104 (2014).
- Duncan, K. R., et al. Molecular Networking and Pattern-Based Genome Mining Improves Discovery of Biosynthetic Gene Clusters and their Products from Salinispora Species. Chemistry & Biology. 22 (4), 460-471 (2015).
- Cao, L., et al. MetaMiner: A Scalable Peptidogenomics Approach for Discovery of Ribosomal Peptide Natural Products with Blind Modifications from Microbial Communities. Cell Systems. , (2019).
- Chen, L. -. Y., Cui, H. -. T., Su, C., Bai, F. -. W., Zhao, X. -. Q. Analysis of the complete genome sequence of a marine-derived strain Streptomyces sp. S063 CGMCC 14582 reveals its biosynthetic potential to produce novel anti-complement agents and peptides. PeerJ. 7 (1), 6122 (2019).
- Kim Tiam, S., et al. Insights into the Diversity of Secondary Metabolites of Planktothrix Using a Biphasic Approach Combining Global Genomics and Metabolomics. Toxins. 11 (9), 498 (2019).
- Özakin, S., Ince, E. Genome and metabolome mining of marine obligate Salinispora strains to discover new natural products. Turkish Journal of Biology. 43 (1), 28-36 (2019).
- Trivella, D. B. B., de Felicio, R. The Tripod for Bacterial Natural Product Discovery: Genome Mining, Silent Pathway Induction, and Mass Spectrometry-Based Molecular Networking. mSystems. 3 (2), 00160 (2018).
- Maansson, M., et al. An Integrated Metabolomic and Genomic Mining Workflow To Uncover the Biosynthetic Potential of Bacteria. mSystems. 1 (3), 1-14 (2016).
- Blin, K., Kim, H. U., Medema, M. H., Weber, T. Recent development of antiSMASH and other computational approaches to mine secondary metabolite biosynthetic gene clusters. Briefings in Bioinformatics. 20 (4), 1103-1113 (2019).
- Fisch, K. M. Biosynthesis of natural products by microbial iterative hybrid PKS-NRPS. RSC Advances. 3 (40), 18228-18247 (2013).
- Tatsuno, S., Arakawa, K., Kinashi, H. Analysis of Modular-iterative Mixed Biosynthesis of Lankacidin by Heterologous Expression and Gene Fusion. The Journal of Antibiotics. 60 (11), 700-708 (2007).
- Helfrich, E. J. N., Piel, J. Biosynthesis of polyketides by trans-AT polyketide synthases. Natural Product Reports. 33 (2), 231-316 (2016).

