用于植物/微生物分子相互作用和信号的同时系统分析的水培共培养系统
Summary
所描述的水培共培养系统支持具有金属筛网的完整植物,并将其与细菌共培养。然后可以单独收集植物组织,细菌和分泌的分子用于下游分析,同时允许研究植物宿主和相互作用的微生物或微生物群的分子响应。
Abstract
模拟天然植物 – 微生物相互作用的实验设计对于描绘复杂的植物 – 微生物信号传导过程非常重要。 拟南芥 – 根癌土壤杆菌 提供了一个优秀的模型系统来研究细菌发病和植物相互作用。以前对植物 – 土壤杆菌相互作用的研究在很大程度上依赖于植物细胞悬浮培养物,植物的人造伤害,或通过合成化学品人工诱导微生物毒力因子或植物防御。然而,这些方法与植物中的自然信号不同,其中植物和微生物以空间和时间的方式识别和响应。这项工作提出了一个水培共培养系统,其中完整的植物由金属筛网支持并与农杆菌共培养。在这种共培养系统中,没有合成的植物激素或诱导微生物的化学物质补充抗性毒性或植物防御。水培共培养系统非常类似于天然植物-微生物相互作用和在植物信号动态平衡。植物根部可以从含有农杆菌的培养基中分离出来,同时有效地研究植物宿主和相互作用的微生物的信号传导和反应。在任何给定的时间点/间隔,可以分别收集植物组织或细菌用于各种“omics”分析,证明该系统的功效和功效。水培共培养系统可以容易地适应于研究:1)不同植物 – 微生物系统的相互信号转导,2)植物宿主与多种微生物物种( 即微生物菌群或微生物群落)之间的信号传导,3)营养物质和化学物质如何牵连在植物微生物信号传导中,以及4)微生物如何与植物宿主相互作用,并有助于植物对生物的耐受性非生物压力。
Introduction
植物相关微生物在生物地球化学循环,生物修复,减缓气候变化,植物生长和健康以及植物对生物和非生物胁迫的耐受方面起重要作用。微生物与两个直接通过植物细胞壁接触,并通过化学分泌间接和信令1,2,3株植物相互作用。作为无生物,植物已经开发出直接和间接的机制来抵抗病原体的感染。直接防御包括结构防御和防御蛋白的表达,而间接防御包括次级代谢产物的植物的生产和生物拮抗对侵入病原体4,5的吸引力。植物来源的根系分泌物,分泌物,粘液,粘液和裂解物改变根际的物理化学性质,以吸引或排斥微生物往其主人6 。根分泌的化学成分是物种特异性的,从而作为选择性过滤器,其允许某些能够识别这些化合物的微生物在根际6中繁殖。因此,相容的微生物物种可以被刺激以激活和增强它们的相互关系,这是为了植物宿主1的益处或损害。
在根际理解植物-微生物相互作用的关键是增强植物生产力和生态系统功能,因为大多数微生物和化学暴露的发生在根部结构和土壤-空气界面2,6,7,8。然而,考虑地下植物 – 微生物的相互作用和互惠的反应是一个挑战,因为它有趣的复杂和动态的性质,以及在紧密可控的生长条件下缺乏具有天然根结构和植物形态的合适的实验模型。作为研究最多的植物病原体之一, 农杆菌感染了广泛的农业和园艺重要植物,包括樱桃,苹果,梨,葡萄和玫瑰9 。 农杆菌是了解植物病原体相互作用的一个重要的模式生物,是在植物转化和植物工程10,11,12,13,14的有力工具。
分子植物 – 农杆菌相互作用已经被很好地研究了几十年,目前对农杆菌致病性的理解是广泛的9 ,F“> 11,15,16。 土壤杆菌的致病性在很大程度上归因于感知植物衍生的信号,从而导致在其毒力方案和细胞-细胞通信的精细调制的其演进能力,所谓群体感应17. 土壤杆菌毒力程序由几种根际信号调节,涉及两组双组分系统,即ChvG / I系统和VirA / G系统,根际酸性条件激活了chvG / I , virA / G的转录,以及涉及农杆菌致病性的其他几个基因,包括virE0 , virE1 , virH1 , virH2和VI型分泌系统(T6SS)的基因18。植物衍生的酚类化合物,包括乙酰丁香酮(4'-羟基-3',5 '二甲氧基苯乙酮),激活VirA / G 2成分系统通过磷酸化信号传导机制19 。 VirA / G然后激活整个vir调节子,导致将来自其肿瘤诱导(Ti)质粒的称为转移DNA(T-DNA)的〜20kb细菌DNA片段转移并整合到植物核16中 。 T-DNA携带负责合成植物激素吲哚-3-乙酸(IAA)( iaaM和iaaH )和细胞分裂素( ipt )的基因 ,一旦在植物细胞中表达,就会产生大量的这些植物激素。这导致异常组织增殖和植物肿瘤的发展,被称为冠瘿病,这是植物9,11,20中的慢性和复苏问题。 IAA还与水杨酸和γ-氨基丁酸共同作用以抑制农杆菌毒力或减少农杆菌米群体感应(QS)17,21,22。为了对抗这种压制,T-DNA还携带基因冠瘿碱的合成,其激活农杆菌群体感应来促进土壤杆菌的致病性,并且还用作对于该病原体22,23的营养源。
尽管对农杆菌 –植物相互作用的总体了解以及由此产生的T-DNA转移到植物宿主中,但在相互作用的初始阶段的复合信号传导事件了解甚少。这部分是由于传统方法调查农杆菌植物信号传导的局限性。植物细胞悬浮培养和人工位点特异性伤害通常用于研究分子植物 – 微生物相互作用24 ,。EF“> 26,27然而,细胞悬浮液缺乏典型植物形态学,特别是,植物悬浮细胞不具有根结构和根渗出物,其是用于激活微生物趋化性和毒力28,29非常重要的植物形态学的维持。和根部结构已经通过人为伤害植物,这有利于特定于站点的感染,从而导致在检测诱导的植物防御相关基因的直接感染植物组织30,31寻址的。然而,人工伤人是从病原体感染性质显著不同,特别是作为伤人导致茉莉酸(JA)的积累,其全身地与天然植物信令和防御26干涉。另外,合成的化学物质通常用于人工诱导植物宿主反应或病原体毒力。虽然反射在植物中的浓度这样的化学化合物的补充是可行的,例如补充不占根分泌物扩散逐渐进入周围根际,其产生由微生物28,32感测的趋化梯度。鉴于常规方法研究植物 – 微生物相互作用的局限性,所获得的数据的准确性和深度可能受到阻碍和限制,而常规方法产生的知识可能不会直接在植物中转化。植物 – 农杆菌信号传导的许多方面尚未完全了解,特别是在相互作用的早期阶段,当疾病症状尚未发展时。
为了修正常规方法的局限性,本文提出了一种廉价,可控的,灵活的水培c摄像系统,允许研究人员在分子植物 – 微生物相互作用的初始阶段更深入地了解复杂的信号和反应途径。水耕已被广泛用来研究植物营养素,根渗出物,生长条件,和金属毒性的植物33,34的影响。水培模型有几个优点,包括小空间要求,各种植物组织的可及性,营养/环境条件的严格控制以及病虫害防治。与琼脂/植物电镀技术相比,水培系统对植物生长的限制也较少,其通常限制2-3周后的生长。重要的是,全植物结构的维护便于必要微生物趋化性和毒力感应8,29自然根分泌。系统描述这里的床比其它的33,34简单,劳动密集程度较低。它使用的部件较少,不需要标准剪刀以外的任何工具。它使用金属网(与尼龙33相反)作为植物生长的强支撑和通过摇动以支持微生物生长的无菌条件下通气的简单方法。此外,该系统可以使用各种尺寸的金属网来支持植物生长,其适应不同植物物种而不限制其根部的宽度。
在这里介绍的水培共培养系统中,植物在无菌水培系统中培养,其中植物根分泌支持接种细菌生长的有机化合物。在这种共培养系统中,补充了人造化学物质,如植物激素,防御诱发剂或毒力诱导化学品,反映了天然细胞在植物 – 微生物相互作用期间进行稳态。利用这种水培共培养系统,可以同时测定农杆菌感染后的拟南芥 Col-0根组织中的基因表达,以及用拟南芥共培养的农杆菌基因的活化。进一步表明,该系统适用于研究农杆菌与植物根系的连接以及植物根系分泌物分布,与土壤杆菌共培养(感染)( 图1 )。
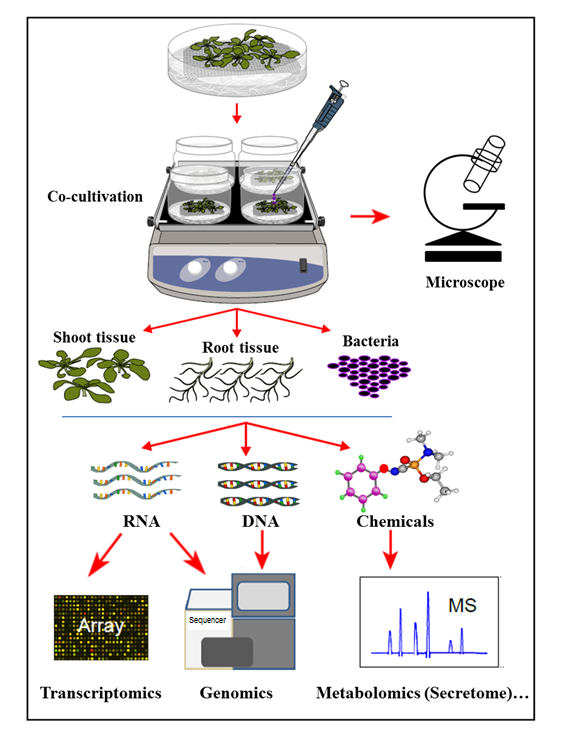
图1:具有样品分析的水培共培养系统概述。将植物生长在网格顶部(在网格上方的芽上),将根浸入水培培养基中,然后用细菌f或共培养。然后分离植物组织和细菌用于同时提取和分析。这个数字已经从参考文献35中修改过。
Protocol
Representative Results
Discussion
鉴于根分泌的逐渐性质, 在植物中产生的毒力诱导化学物质的浓度及其对动植物 – 微生物相互作用的影响发生在空间和时间梯度上。在这种水培共培养系统中,不补充诱导微生物毒力或植物防御的合成植物激素或化学物质。相比之下,使用常规方法,添加合成化学品,如乙酰丁香酮,会产生突然的,人为的浓度峰值。因此,这种水培共培养系统更加模仿自然环境,微生物和宿主植物在空间…
Disclosures
The authors have nothing to disclose.
Acknowledgements
我们要感谢Brian Weselowski和Alexander W. Eastman的帮助和有益的讨论。我们也要感谢Drs。尤金·韦斯特,张敏瑞,沉海涛,崔翠翠,格雷格·索恩为他们的帮助,有益的讨论和对稿件的批判性阅读。该研究由作者作为其职责的一部分由加拿大农业和农业食品加拿大农业和农业发展组织(RBPI号码2555)和成长前进II项目1670号资助。本研究还部分由加拿大自然科学与工程研究理事会(NSERC)发现奖励RGPIN-2015-06052授予中国元。
Materials
| plant seeds (Arabidopsis thaliana Col-0) | Arabidopsis Biological Resource Centre | CS7000 | https://abrc.osu.edu/order-stocks |
| bacteria (Agrobacterium tumefaciens C58) | University of Washington | N/A | |
| labeled bacteria | in-house | optional, depends on downstream analyses | |
| vortex | (various) | ||
| microcentrifuge tubes | (various) | ||
| microcentrifuge | (various) | ||
| 5% sodium hypochlorite | (various) | ||
| double distilled water | (various) | ||
| autoclave | (various) | ||
| micropipette | (various) | ||
| 70 % ethanol | (various) | ||
| Murashige and Skoog (MS) basal salts | Sigma-Aldrich | M5524 | |
| sucrose | (various) | ||
| MES | (various) | ||
| B5 vitamin mix | Sigma-Aldrich | G1019 | |
| phytoagar | (various) | ||
| deep Petri dishes | (various) | ||
| stainless steel mesh | Ferrier Wire Goods Company Ltd | N/A | grade: 304; mesh count: 40 × 40; wire DIA: 0.01 |
| micropore tape, 1 inch | 3M | 1530-1 | |
| diurnal growth chamber | (various) | ||
| cylindrical glass tanks, 100 × 80 mm | Pyrex | 3250 | other sizes can be used, in which case liquid content may need adjustment |
| flow hood | (various) | ||
| forcepts | (various) | ||
| yeast extract | (various) | ||
| tryptone | (various) | ||
| MgSO4 | (various) | ||
| shaking incubator | (various) | ||
| spectrophotometer | (various) | ||
| NaCl | (various) | ||
| shaker | (various) | ||
| scissors | (various) | optional, depends on downstream analyses | |
| fluorescence microscope | (various) | optional, depends on downstream analyses | |
| microscope slides and cover slips | (various) | optional, depends on downstream analyses | |
| nail polish | (various) | optional, depends on downstream analyses | |
| Bacterial RNA extraction kit | (various) | optional, depends on downstream analyses | |
| plant RNA extraction kit (RNeasy Plant Mini Kit) | Qiagen | 74903 or 74904 | optional, depends on downstream analyses |
| material and equipment for RT-qPCR | (various) | optional, depends on downstream analyses | |
| material and equipment for microarray analysis | (various) | optional, depends on downstream analyses | |
| liquid nitrogen | (various) | optional, depends on downstream analyses | |
| mortar and pestle | (various) | optional, depends on downstream analyses | |
| 0.2 µm pore filter | (various) | optional, depends on downstream analyses | |
| 50 mL conical tubes | (various) | optional, depends on downstream analyses | |
| freeze dryer | (various) | optional, depends on downstream analyses | |
| sealable test tubes | (various) | optional, depends on downstream analyses | |
| ethyl acetate | (various) | optional, depends on downstream analyses | |
| nitrogen gas | (various) | optional, depends on downstream analyses | |
| material and equipment for HPLC | (various) | optional, depends on downstream analyses | |
| material and equipment for ESI-TOF-MS | (various) | optional, depends on downstream analyses |
References
- Lambers, H., Mougel, C., Jaillard, B., Hinsinger, P. Plant-microbe-soil interactions in the rhizosphere: An evolutionary perspective. Plant Soil. 321 (1), 83-115 (2009).
- Philippot, L., Raaijmakers, J. M., Lemanceau, P., vander Putten, W. H. Going back to the roots: The microbial ecology of the rhizosphere. Nat. Rev. Microbiol. 11 (11), 789-799 (2013).
- Somers, E., Vanderleyden, J., Srinivasan, M. Rhizosphere bacterial signalling: A Love Parade beneath our feet. Crit. Rev. Microbiol. 30 (4), 205-240 (2004).
- Barah, P., Winge, P., Kusnierczyk, A., Tran, D. H., Bones, A. M. Molecular signatures in Arabidopsis thaliana in response to insect attack and bacterial infection. PLoS ONE. 8 (3), (2013).
- Zhang, J., Zhou, J. -. M. Plant immunity triggered by microbial molecular signatures. Mol. Plant. 3 (5), 783-793 (2010).
- Paterson, E., Gebbing, T., Abel, C., Sim, A., Telfer, G. Rhizodeposition shapes rhizosphere microbial community structure in organic soil. New Phytol. 173 (3), 600-610 (2006).
- Hartmann, A., Schmid, M., van Tuinen, D., Berg, G. Plant-driven selection of microbes. Plant Soil. 321 (1-2), 235-257 (2008).
- Micallef, S. A., Shiaris, M. P., Colon-Carmona, A. Influence of Arabidopsis thaliana accessions on rhizobacterial communities and natural variation in root exudates. J. Exp. Bot. 60 (6), 1729-1742 (2009).
- Burr, T., Otten, L. Crown gall of grape: Biology and disease management. Annu. Rev. Phytopathol. 37, 53-80 (2001).
- Clough, S. J., Bent, A. F. Floral dip: A simplified method for Agrobacterium-mediated transformation of Arabidopsis thaliana. Plant J. 16 (6), 735-743 (1998).
- Binns, A. N., Costantino, P., Spaink, H. P., Kondorosi, A., Hooykaas, P. J. J. The Agrobacterium oncogenes. The Rhizobiaceae. , (1998).
- Gheysen, G., Angenon, G., Van Montagu, M., Lindsey, K. Agrobacterium-mediated plant transformation: a scientifically intriguing story with significant applications. Transgenic Plant Research. , (1998).
- Valvekens, D., Von Montagu, M. V., Van Lijsebettens, M. Agrobacterium tumefaciens-mediated transformation of Arabidopsis thaliana root explants by using kanamycin selection. Proc. Natl. Acad. Sci. USA. 85 (15), 5536-5540 (1988).
- Krysan, P. J., Young, J. C., Sussman, M. R. T-DNA as an insertional mutagen in Arabidopsis. Plant Cell. 11 (12), 2283 (1999).
- Chilton, M. D., et al. Stable incorporation of plasmid DNA into higher plant cells: The molecular basis of crown gall tumorigenesis. Cell. 11 (2), 263-271 (1977).
- Gelvin, S. B. Agrobacterium in the genomics age. Plant Physiol. 150 (4), 1665-1676 (2009).
- Subramoni, S., Nathoo, N., Klimov, E., Yuan, Z. -. C. Agrobacterium tumefaciens responses to plant-derived signaling molecules. Front. Plant Sci. 5, 322 (2014).
- Yuan, Z., Liu, P., Saenkham, P., Kerr, K., Nester, E. W. Transcriptome profiling and functional analysis of Agrobacterium tumefaciens reveals a general conserved response to acidic conditions (pH 5.5) and a complex acid-mediated signaling involved in Agrobacterium-plant interactions. J. Bacteriol. 190 (2), 494-507 (2008).
- Stachel, S. E., Messens, E., Van Montagu, M., Zambryski, P. Identification of the signal molecules produced by wounded plant cells that activate T-DNA transfer in Agrobacterium tumefaciens. Nature. 318 (6047), 624-629 (1985).
- Memelink, J., de Pater, B. S., Hoge, J. H. C., Schilperoort, R. A. T-DNA hormone biosynthetic genes: Phytohormones and gene expression in plants. Dev. Genet. 8 (5-6), 321-337 (1987).
- Yuan, Z. -. C., et al. The plant signal salicylic acid shuts down expression of the vir regulon and activates quormone-quenching genes in Agrobacterium. Proc. Natl. Acad. Sci. USA. 104 (28), 11790-11795 (2007).
- Yuan, Z. -. C., Haudecoeur, E., Faure, D., Kerr, K. F., Nester, E. W. Comparative transcriptome analysis of Agrobacterium tumefaciens in response to plant signal salicylic acid, indole-3-acetic acid and γ-amino butyric acid reveals signalling cross-talk and Agrobacterium-plant co-evolution. Cell. Microbiol. 10 (11), 2339-2354 (2008).
- Li, P. L., Farrand, S. K. The replicator of the nopaline-type Ti plasmid pTiC58 is a member of the repABC family and is influenced by the TraR-dependent quorum-sensing regulatory system. J. Bacteriol. 182 (1), 179-188 (2000).
- Atkinson, M. M., Huang, J., Knopp, J. A. Hypersensitivity of suspension-cultured tobacco cells to pathogenic bacteria. Phytopathology. 75 (11), 1270-1274 (1985).
- Veena, ., Jiang, H., Doerge, R. W., Gelvin, S. B. Transfer of T-DNA and Vir proteins to plant cells by Agrobacterium tumefaciens induces expression of host genes involved in mediating transformation and suppresses host defense gene expression. Plant J. 35 (2), 219-236 (2003).
- León, J., Rojo, E., Sanchez-Serrano, J. J. Wound signalling in plants. J. Exp. Bot. 52 (354), 1-9 (2001).
- Ditt, R. F., Kerr, K. F., de Figueiredo, P., Delrow, J., Comai, L., Nester, E. W. The Arabidopsis thaliana transcriptome in response to Agrobacterium tumefaciens. Mol. Plant Microbe In. 19 (6), 665-681 (2006).
- Mandimba, G., Heulin, T., Bally, R., Guckert, A., Balandreau, J. Chemotaxis of free-living nitrogen-fixing bacteria towards maize mucilage. Plant Soil. 90 (1-3), 129-139 (1986).
- Rudrappa, T., Czymmek, K. J., Pare, P. W., Bais, H. P. Root-secreted malic acid recruits beneficial soil bacteria. Plant Physiol. 148 (3), 1547-1556 (2008).
- Lee, C. W., et al. Agrobacterium tumefaciens promotes tumor induction by modulating pathogen defense in Arabidopsis thaliana. Plant Cell. 21 (9), 2948-2962 (2009).
- Reymond, P., Weber, H., Damond, M., Farmer, E. E. Differential gene expression in response to mechanical wounding and insect feeding in Arabidopsis. Plant Cell. 12 (5), 707-720 (2000).
- Matthysse, A. G. Attachment of Agrobacterium to plant surfaces. Front. Plant Sci. 5, 252 (2014).
- Alatorre-Cobos, F., et al. An improved, low-cost, hydroponic system for growing Arabidopsis and other plant species under aseptic conditions. BMC Plant Biol. 14 (1), 69 (2014).
- Conn, S. J., et al. Protocol: Optimising hydroponic growth systems for nutritional and physiological analysis of Arabidopsis thaliana and other plants. Plant Methods. 9 (1), 4 (2013).
- Nathoo, N. . Identification of putative plant defense genes using a novel hydroponic co-cultivation technique for studying plant-pathogen interaction. , (2015).
- Shaner, N. C., Campbell, R. E., Steinbach, P. A., Giepmans, B. N., Palmer, A. E., Tsien, R. Y. Improved monomeric red, orange and yellow fluorescent proteins derived from Discosoma sp. red fluorescent protein. Nat. Biotechnol. 22 (12), 1567-1572 (2004).
- Wise, A. A., Liu, H., Binns, A. N. Nucleic acid extraction from Agrobacterium strains. Methods Mol. Bio. 343, 67-76 (2006).
- Nolan, T., Hands, R. E., Bustin, S. A. Quantification of mRNA using real-time RT-PCR. Nat. Protoc. 1 (3), 1559-1582 (2006).
- Salter, M. G., Conlon, H. E. Extraction of plant RNA. Methods Mol. Bio. 362, 309-314 (2007).
- Bao, Y., Wang, S., Yang, X., Li, T., Xia, Y., Meng, X. Metabolomic study of the intervention effects of Shuihonghuazi Formula, a Traditional Chinese Medicinal formulae, on hepatocellular carcinoma (HCC) rats using performance HPLC/ESI-TOF-MS. J. Ethnopharmacol. 198, 468-478 (2017).
- Korves, T. M., Bergelson, J. A developmental response to pathogen infection in Arabidopsis. Plant Physiol. 133 (1), 339-347 (2003).
- Lyons, R., Rusu, A., Stiller, J., Powell, J., Manners, J. M., Kazan, K. Investigating the association between flowering time and defense in the Arabidopsis thaliana-Fusarium oxysporum interaction. PLoS ONE. 10 (6), e0127699 (2015).
- Badri, D. V., Weir, T. L., vander Lelie, D., Vivanco, J. M. Rhizosphere chemical dialogues: Plant-microbe interactions. Curr. Opin. Biotechnol. 20 (6), 642-650 (2009).
- Baerson, S. R., et al. Detoxification and transcriptome response in Arabidopsis seedlings exposed to the allelochemical benzoxazolin-2(3H)-one. J. Biol. Chem. 280 (23), 21867-21881 (2005).

