Un sistema di co-coltivazione idroponica per l'analisi simultanea e sistematica delle interazioni molecolari e della segnalazione delle piante / microbe
Summary
Il sistema di cocultivazione idroponico descritto supporta piante intatte con schermi a maglie metalliche e cocultivala con batteri. I tessuti vegetali, i batteri e le molecole secrete possono quindi essere raccolte separatamente per analisi a valle, consentendo contemporaneamente le risposte molecolari di entrambi gli host vegetali e dei microbi o dei microbiomi interattivi.
Abstract
Un disegno sperimentale che imita le interazioni microbi-vegetali naturali è molto importante per delineare i complessi processi di segnalazione plant-microbe. Arabidopsis thaliana – Agrobacterium tumefaciens Fornisce un eccellente sistema di modelli per studiare la patogenesi batterica e le interazioni vegetali. Studi precedenti sulle interazioni plant- Agrobacterium si sono basate in gran parte sulle colture di sospensione delle cellule vegetali, la ferita artificiale delle piante, o l'induzione artificiale di fattori di virulenza di microbi o difese vegetali da sostanze chimiche sintetiche. Tuttavia, questi metodi sono distinti dalla naturale segnalazione in pianta , dove le piante e i microbi riconoscono e reagiscono in maniera spaziale e temporale. Questo lavoro presenta un sistema di cocultivazione idroponica in cui le piante intatte sono supportate da schermi di maglie metalliche e cocultivate con Agrobacterium . In questo sistema di cocultivazione, nessun fitotormone sintetico o chimico che induce micrÈ integrata la virulenza o la difesa vegetale. Il sistema di cocultivazione idroponica somiglia molto alle interazioni microbi-vegetali naturali e alla homeostasi di segnalazione in planta . Le radici delle piante possono essere separate dal mezzo contenente Agrobacterium e la segnalazione e le risposte sia degli host vegetali che dei microbi interagenti possono essere studiati contemporaneamente e sistematicamente. In qualsiasi momento / intervallo di tempo, i tessuti vegetali oi batteri possono essere raccolti separatamente per varie analisi "omiche", dimostrando il potere e l'efficacia di questo sistema. Il sistema di coltivazione idroponica può essere facilmente adattato per studiare: 1) la segnalazione reciproca di diversi sistemi vegetali-microbici, 2) la segnalazione tra un host vegetale e più specie microbiche ( cioè consorzi microbiali o microbiomi), 3) come sono implicati i nutrienti e le sostanze chimiche Nella segnalazione di microrganismi vegetali e 4) come i microbi interagiscono con gli host vegetali e contribuiscono alla tolleranza vegetale a bioticiR sollecitazioni abiotiche.
Introduction
I microbi associati all'impianto svolgono un ruolo importante nel ciclo biogeochimico, nella bioremediazione, nella mitigazione del cambiamento climatico, nella crescita vegetale e nella salute, e nella tolleranza alle tensioni alle tensioni biotiche e abiotiche. I microrganismi interagiscono con le piante sia direttamente attraverso il contatto cellulare a parete cellulare che indirettamente tramite secrezione chimica e segnalazione 1 , 2 , 3 . Come organismi sessili, le piante hanno sviluppato meccanismi diretti e indiretti per resistere all'infezione da agenti patogeni. Le difese dirette comprendono le difese strutturali e l'espressione delle proteine di difesa, mentre le difese indirette includono la produzione secondaria del metabolita vegetale e l'attrazione degli organismi antagonisti agli invasori patogeni 4 , 5 . Gli essudati radici derivanti dalle piante, le secrezioni, le mucillagini, i mucigel e i lisati alterano le proprietà fisico-chimiche della rizosfera per attirare o respingereMicrobi verso i loro ospiti 6 . La composizione chimica della secrezione radicale è specie-specifica, servendo così come un filtro selettivo che consente a certi microrganismi in grado di riconoscere tali composti a fiorire nella rizosfera 6 . Pertanto, le specie microbiche compatibili possono essere stimolate ad attivare e migliorare le loro associazioni, sia a vantaggio che a detrimento dell'ospite vegetale 1 .
La comprensione delle interazioni tra pianta e microbe nella rizosfera è fondamentale per aumentare la produttività delle piante e il funzionamento dell'ecosistema, in quanto la maggior parte delle esposizioni microbiche e chimiche si verifica nella struttura radice e nell'interfaccia del suolo-aria 2 , 6 , 7 , 8 . Tuttavia, l'esame delle interazioni tra piante e micro-piani sotterranee e le risposte reciproche è stata una sfida a causa del suo intrigante Complessa e dinamica e la mancanza di adeguati modelli sperimentali con struttura radicale naturale e morfologia vegetale in condizioni di crescita strettamente controllabili. Come uno dei fitopatogeni più pesantemente studiati, Agrobacterium infetta un'ampia gamma di piante con importanza agricola e orticola, tra cui la ciliegia, la mela, la pera, l'uva e la rosa 9 . Agrobacterium è un organismo di modello importante per la comprensione delle interazioni patogeno-patogeno ed è un potente strumento di trasformazione delle piante e di ingegneria degli impianti 10 , 11 , 12 , 13 , 14 .
Le interazioni molecolari delle piante- Agrobacterium sono state ben studiate per diversi decenni e l'attuale comprensione della patogenicità di Agrobacterium è estesa 9 ,F "> 11 , 15 , 16. La patogenicità di Agrobacterium è in gran parte attribuita alle sue capacità evolute di percepire i segnali derivanti dalle piante, con conseguente fine modulazione del suo programma di virulenza e comunicazione cellulare da cellule, il cosiddetto sensore quorum 17 . Il programma di virulenza di Agrobacterium è regolato da diversi segnali disponibili nella rizosfera e coinvolge due set di sistemi a 2 componenti, il sistema ChvG / I e il sistema VirA / G. Le condizioni acide nella rizosfera attivano la trascrizione di chvG / I , virA / G , E molti altri geni coinvolti nella patogenicità di Agrobacterium , compresi virE0 , virE1 , virH1 , virH2 e geni del sistema di secrezione di tipo VI (T6SS) 18 . '-dimetossiacetofenone), attivare la VSistema 2-componente irA / G attraverso i meccanismi di segnalazione della fosforilazione 19 . VirA / G quindi attiva l'intera regolazione vir , con conseguente trasferimento e integrazione di un frammento di DNA batterico ~ 20 kb chiamato DNA di trasferimento (T-DNA) dal suo tumore-inducente (Ti) plasmide nel nucleo di pianta 16 . T-DNA porta i geni responsabili della sintesi degli ormoni vegetali indole-3-acetico (IAA) ( iaaM e iaaH ) e citochinina ( ipt ) e una volta espressi in cellule vegetali, si producono grandi quantità di questi fitotomi. Ciò si traduce in una proliferazione anomala del tessuto e nello sviluppo del tumore delle piante, noto come malattia della corona, che è un problema cronico e risorgente per le piante 9 , 11 , 20 . L'IAA agisce anche collettivamente con acido salicilico e acido gamma-ammino butirrico per reprimere la virulenza di Agrobacterium o per ridurre l' Agrobacteriu M quorum sensing (QS) 17 , 21 , 22 . Per contrastare questa repressione, T-DNA porta anche dei geni per la biosintesi opina, che attiva Agrobacterium quorum sensing per promuovere la patogenicità di Agrobacterium e serve anche come fonte nutritiva per il patogeno 22 , 23 .
Nonostante una comprensione approfondita delle interazioni di Agrobacterium- Plant e del trasferimento di T-DNA risultante nell'ospite della pianta, gli eventi complessi di segnalazione nella fase iniziale dell'interazione sono meno ben compresi. Ciò è dovuto in parte alle limitazioni degli approcci convenzionali per indagare la segnalazione Agrobacterium- Plant. Le colture di sospensione delle cellule vegetali e ferite artificiali specifiche del sito vengono comunemente utilizzate per studiare le interazioni tra microbi e piante molecolari 24 ,. ef "> 26, 27 Tuttavia, sospensioni cellulari mancare tipica morfologia delle piante, in particolare celle di sospensione pianta non hanno strutture radicolari e essudati radicali, che sono molto importanti per l'attivazione chemiotassi microbica e virulenza 28, 29 Il mantenimento della morfologia delle piante. E la struttura delle radici è stata affrontata da piante ferite artificiali che facilitano l'infezione specifica del sito, con conseguente individuazione di geni correlati alla difesa vegetale indotta nel tessuto vegetale direttamente infetto 30 , 31. Tuttavia, ferita artificiale è significativamente diversa dall'infezione patogena in natura , In particolare in quanto ferita porta ad accumulo di acido jasmone (JA) che interferisce sistematicamente con la segnalazione e la difesa naturale delle piante 26. Inoltre, sono tipicamente impiegate sostanze chimiche sintetiche per indurre artificialmente le risposte dell'organismo vegetaleO la virulenza del patogeno. Anche se l'integrazione di tali composti chimici che riflettono concentrazioni in planta è possibile, tale integrazione non rende conto della diffusione di essudati radici gradualmente nella rizosfera circostante, che genera un gradiente chemotattico rilevato dai microbi 28 , 32 . Data la limitazione degli approcci convenzionali per studiare le interazioni tra pianta e microbe, l'accuratezza e la profondità dei dati ottenuti potrebbero essere ostacolati e restrittivi e le conoscenze generate dagli approcci convenzionali non possono essere tradotte direttamente in planta . Molti aspetti della segnalazione di pianta- Agrobacterium non sono ancora pienamente compresi, in particolare nella fase iniziale delle interazioni, quando i sintomi della malattia non sono ancora stati sviluppati.
Per modificare le limitazioni degli approcci convenzionali, questo lavoro presenta un sistema idroponico poco costoso, strettamente controllabile e flessibileSistema di ocultivazione che consente ai ricercatori di acquisire approfondimenti sui complessi percorsi di segnalazione e risposta nella fase iniziale delle interazioni molecolari delle piante-microbe. L'idroponica è stata ampiamente usata per studiare le sostanze nutritive vegetali, gli essudati radici, le condizioni di crescita e gli effetti della tossicità metallica sulle piante 33 , 34 . Ci sono diversi vantaggi di modelli idroponici, tra cui i piccoli requisiti spaziali, l'accessibilità di vari tessuti vegetali, il controllo stretto delle condizioni nutrizionali / ambientali e il controllo di parassiti / malattie. I sistemi idroponici sono anche meno limitanti per la crescita delle piante rispetto alle tecniche di placcatura agar / phytoagar, che in genere limitano la crescita dopo 2-3 settimane. Importante, il mantenimento delle strutture vegetali intere facilita la secrezione naturale della radice necessaria per la chemiotassi microbica e l'induzione della virulenza 8 , 29 . Il sistema descriIl letto qui è più semplice e meno intensivo rispetto alle alternative 33 , 34 . Utilizza meno parti e non richiede strumenti diversi dalle forbici standard. Utilizza la maglia metallica (in contrapposizione al nylon 33 ) come un forte supporto per la crescita delle piante e un semplice metodo di aerazione in condizioni sterili mediante agitazione per supportare la crescita microbica. Inoltre, il sistema può utilizzare maglie metalliche di varie dimensioni per sostenere la crescita vegetale, che accoglie diverse specie vegetali senza limitare la larghezza delle loro radici.
Nel sistema di coltivazione idroponica qui presentato, le piante vengono coltivate in un sistema idroponico sterile dove le radici vegetali secernono composti organici che sostengono la crescita dei batteri inoculati. In questo sistema di cocultivazione non vengono aggiunti prodotti chimici artificiali, come ormoni vegetali, elicitor di difesa o sostanze chimiche che inducono la virulenza, che riflette la cella naturale-signostasi dell'omeostasi durante le interazioni plant-microbe. Con questo sistema di cocultivazione idroponica è stato possibile determinare simultaneamente l'espressione genica nel tessuto radicale Arabidopsis thaliana Col-0 su infezione da Agrobacterium , nonché l'attivazione di geni Agrobacterium dopo cocultivazione con Arabidopsis . È stato inoltre dimostrato che questo sistema è idoneo a studiare l'attaccamento di Agrobacterium alle radici vegetali, nonché al profilo di segreto radicale di pianta, dopo la cocultivazione (infezione) con Agrobacterium ( Figura 1 ).
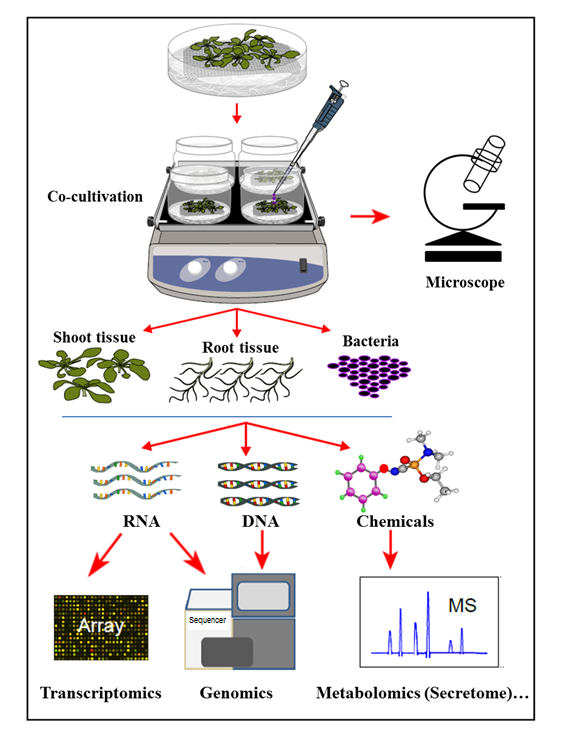
Figura 1: Panoramica del Sistema di Cocultivazione Idroponica, con analisi del campione. Le piante sono coltivate in cima alla maglia (germogli al di sopra della maglia), con le radici immerse in mezzo idroponico che viene quindi inoculato con batteri fO cocultura. I tessuti e i batteri delle piante vengono quindi separati per le estrazioni e le analisi simultanee. Questa cifra è stata modificata dal riferimento 35 .
Protocol
Representative Results
Discussion
Data la natura graduale della secrezione radicale, la concentrazione di sostanze chimiche che inducono la virulenza prodotta in planta ei loro effetti sulle interazioni dinamiche tra pianta e microbici si verificano in gradienti spaziali e temporali. In questo sistema di co-coltivazione idroponica, non è completato alcun fitotormone sintetico o chimico che induce virulenza di microbi o difese delle piante. Al contrario, utilizzando approcci convenzionali, l'aggiunta di sostanze chimiche sintetiche, come il…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Vorremmo ringraziare Brian Weselowski e Alexander W. Eastman per il loro aiuto e la loro utile discussione. Ringraziamo anche i dottori. Eugene W. Nester, Lingrui Zhang, Haitao Shen, Yuhai Cui e Greg Thorn per il loro aiuto, discussioni utili e lettura critica del manoscritto. Questa ricerca è stata finanziata dall'Agricoltura e Agri-Food Canada, dal Growing Forward-AgriFlex (numero RBPI numero 2555) e dal progetto Growing Forward II numero 1670, condotto dagli autori come parte delle loro funzioni. Questo studio è stato anche parzialmente finanziato dalla ZC Yuan di Grant RGPIN-2015-06052 del Ricercatore di Scienze Naturali e di Ingegneria del Canada (NSERC).
Materials
| plant seeds (Arabidopsis thaliana Col-0) | Arabidopsis Biological Resource Centre | CS7000 | https://abrc.osu.edu/order-stocks |
| bacteria (Agrobacterium tumefaciens C58) | University of Washington | N/A | |
| labeled bacteria | in-house | optional, depends on downstream analyses | |
| vortex | (various) | ||
| microcentrifuge tubes | (various) | ||
| microcentrifuge | (various) | ||
| 5% sodium hypochlorite | (various) | ||
| double distilled water | (various) | ||
| autoclave | (various) | ||
| micropipette | (various) | ||
| 70 % ethanol | (various) | ||
| Murashige and Skoog (MS) basal salts | Sigma-Aldrich | M5524 | |
| sucrose | (various) | ||
| MES | (various) | ||
| B5 vitamin mix | Sigma-Aldrich | G1019 | |
| phytoagar | (various) | ||
| deep Petri dishes | (various) | ||
| stainless steel mesh | Ferrier Wire Goods Company Ltd | N/A | grade: 304; mesh count: 40 × 40; wire DIA: 0.01 |
| micropore tape, 1 inch | 3M | 1530-1 | |
| diurnal growth chamber | (various) | ||
| cylindrical glass tanks, 100 × 80 mm | Pyrex | 3250 | other sizes can be used, in which case liquid content may need adjustment |
| flow hood | (various) | ||
| forcepts | (various) | ||
| yeast extract | (various) | ||
| tryptone | (various) | ||
| MgSO4 | (various) | ||
| shaking incubator | (various) | ||
| spectrophotometer | (various) | ||
| NaCl | (various) | ||
| shaker | (various) | ||
| scissors | (various) | optional, depends on downstream analyses | |
| fluorescence microscope | (various) | optional, depends on downstream analyses | |
| microscope slides and cover slips | (various) | optional, depends on downstream analyses | |
| nail polish | (various) | optional, depends on downstream analyses | |
| Bacterial RNA extraction kit | (various) | optional, depends on downstream analyses | |
| plant RNA extraction kit (RNeasy Plant Mini Kit) | Qiagen | 74903 or 74904 | optional, depends on downstream analyses |
| material and equipment for RT-qPCR | (various) | optional, depends on downstream analyses | |
| material and equipment for microarray analysis | (various) | optional, depends on downstream analyses | |
| liquid nitrogen | (various) | optional, depends on downstream analyses | |
| mortar and pestle | (various) | optional, depends on downstream analyses | |
| 0.2 µm pore filter | (various) | optional, depends on downstream analyses | |
| 50 mL conical tubes | (various) | optional, depends on downstream analyses | |
| freeze dryer | (various) | optional, depends on downstream analyses | |
| sealable test tubes | (various) | optional, depends on downstream analyses | |
| ethyl acetate | (various) | optional, depends on downstream analyses | |
| nitrogen gas | (various) | optional, depends on downstream analyses | |
| material and equipment for HPLC | (various) | optional, depends on downstream analyses | |
| material and equipment for ESI-TOF-MS | (various) | optional, depends on downstream analyses |
References
- Lambers, H., Mougel, C., Jaillard, B., Hinsinger, P. Plant-microbe-soil interactions in the rhizosphere: An evolutionary perspective. Plant Soil. 321 (1), 83-115 (2009).
- Philippot, L., Raaijmakers, J. M., Lemanceau, P., vander Putten, W. H. Going back to the roots: The microbial ecology of the rhizosphere. Nat. Rev. Microbiol. 11 (11), 789-799 (2013).
- Somers, E., Vanderleyden, J., Srinivasan, M. Rhizosphere bacterial signalling: A Love Parade beneath our feet. Crit. Rev. Microbiol. 30 (4), 205-240 (2004).
- Barah, P., Winge, P., Kusnierczyk, A., Tran, D. H., Bones, A. M. Molecular signatures in Arabidopsis thaliana in response to insect attack and bacterial infection. PLoS ONE. 8 (3), (2013).
- Zhang, J., Zhou, J. -. M. Plant immunity triggered by microbial molecular signatures. Mol. Plant. 3 (5), 783-793 (2010).
- Paterson, E., Gebbing, T., Abel, C., Sim, A., Telfer, G. Rhizodeposition shapes rhizosphere microbial community structure in organic soil. New Phytol. 173 (3), 600-610 (2006).
- Hartmann, A., Schmid, M., van Tuinen, D., Berg, G. Plant-driven selection of microbes. Plant Soil. 321 (1-2), 235-257 (2008).
- Micallef, S. A., Shiaris, M. P., Colon-Carmona, A. Influence of Arabidopsis thaliana accessions on rhizobacterial communities and natural variation in root exudates. J. Exp. Bot. 60 (6), 1729-1742 (2009).
- Burr, T., Otten, L. Crown gall of grape: Biology and disease management. Annu. Rev. Phytopathol. 37, 53-80 (2001).
- Clough, S. J., Bent, A. F. Floral dip: A simplified method for Agrobacterium-mediated transformation of Arabidopsis thaliana. Plant J. 16 (6), 735-743 (1998).
- Binns, A. N., Costantino, P., Spaink, H. P., Kondorosi, A., Hooykaas, P. J. J. The Agrobacterium oncogenes. The Rhizobiaceae. , (1998).
- Gheysen, G., Angenon, G., Van Montagu, M., Lindsey, K. Agrobacterium-mediated plant transformation: a scientifically intriguing story with significant applications. Transgenic Plant Research. , (1998).
- Valvekens, D., Von Montagu, M. V., Van Lijsebettens, M. Agrobacterium tumefaciens-mediated transformation of Arabidopsis thaliana root explants by using kanamycin selection. Proc. Natl. Acad. Sci. USA. 85 (15), 5536-5540 (1988).
- Krysan, P. J., Young, J. C., Sussman, M. R. T-DNA as an insertional mutagen in Arabidopsis. Plant Cell. 11 (12), 2283 (1999).
- Chilton, M. D., et al. Stable incorporation of plasmid DNA into higher plant cells: The molecular basis of crown gall tumorigenesis. Cell. 11 (2), 263-271 (1977).
- Gelvin, S. B. Agrobacterium in the genomics age. Plant Physiol. 150 (4), 1665-1676 (2009).
- Subramoni, S., Nathoo, N., Klimov, E., Yuan, Z. -. C. Agrobacterium tumefaciens responses to plant-derived signaling molecules. Front. Plant Sci. 5, 322 (2014).
- Yuan, Z., Liu, P., Saenkham, P., Kerr, K., Nester, E. W. Transcriptome profiling and functional analysis of Agrobacterium tumefaciens reveals a general conserved response to acidic conditions (pH 5.5) and a complex acid-mediated signaling involved in Agrobacterium-plant interactions. J. Bacteriol. 190 (2), 494-507 (2008).
- Stachel, S. E., Messens, E., Van Montagu, M., Zambryski, P. Identification of the signal molecules produced by wounded plant cells that activate T-DNA transfer in Agrobacterium tumefaciens. Nature. 318 (6047), 624-629 (1985).
- Memelink, J., de Pater, B. S., Hoge, J. H. C., Schilperoort, R. A. T-DNA hormone biosynthetic genes: Phytohormones and gene expression in plants. Dev. Genet. 8 (5-6), 321-337 (1987).
- Yuan, Z. -. C., et al. The plant signal salicylic acid shuts down expression of the vir regulon and activates quormone-quenching genes in Agrobacterium. Proc. Natl. Acad. Sci. USA. 104 (28), 11790-11795 (2007).
- Yuan, Z. -. C., Haudecoeur, E., Faure, D., Kerr, K. F., Nester, E. W. Comparative transcriptome analysis of Agrobacterium tumefaciens in response to plant signal salicylic acid, indole-3-acetic acid and γ-amino butyric acid reveals signalling cross-talk and Agrobacterium-plant co-evolution. Cell. Microbiol. 10 (11), 2339-2354 (2008).
- Li, P. L., Farrand, S. K. The replicator of the nopaline-type Ti plasmid pTiC58 is a member of the repABC family and is influenced by the TraR-dependent quorum-sensing regulatory system. J. Bacteriol. 182 (1), 179-188 (2000).
- Atkinson, M. M., Huang, J., Knopp, J. A. Hypersensitivity of suspension-cultured tobacco cells to pathogenic bacteria. Phytopathology. 75 (11), 1270-1274 (1985).
- Veena, ., Jiang, H., Doerge, R. W., Gelvin, S. B. Transfer of T-DNA and Vir proteins to plant cells by Agrobacterium tumefaciens induces expression of host genes involved in mediating transformation and suppresses host defense gene expression. Plant J. 35 (2), 219-236 (2003).
- León, J., Rojo, E., Sanchez-Serrano, J. J. Wound signalling in plants. J. Exp. Bot. 52 (354), 1-9 (2001).
- Ditt, R. F., Kerr, K. F., de Figueiredo, P., Delrow, J., Comai, L., Nester, E. W. The Arabidopsis thaliana transcriptome in response to Agrobacterium tumefaciens. Mol. Plant Microbe In. 19 (6), 665-681 (2006).
- Mandimba, G., Heulin, T., Bally, R., Guckert, A., Balandreau, J. Chemotaxis of free-living nitrogen-fixing bacteria towards maize mucilage. Plant Soil. 90 (1-3), 129-139 (1986).
- Rudrappa, T., Czymmek, K. J., Pare, P. W., Bais, H. P. Root-secreted malic acid recruits beneficial soil bacteria. Plant Physiol. 148 (3), 1547-1556 (2008).
- Lee, C. W., et al. Agrobacterium tumefaciens promotes tumor induction by modulating pathogen defense in Arabidopsis thaliana. Plant Cell. 21 (9), 2948-2962 (2009).
- Reymond, P., Weber, H., Damond, M., Farmer, E. E. Differential gene expression in response to mechanical wounding and insect feeding in Arabidopsis. Plant Cell. 12 (5), 707-720 (2000).
- Matthysse, A. G. Attachment of Agrobacterium to plant surfaces. Front. Plant Sci. 5, 252 (2014).
- Alatorre-Cobos, F., et al. An improved, low-cost, hydroponic system for growing Arabidopsis and other plant species under aseptic conditions. BMC Plant Biol. 14 (1), 69 (2014).
- Conn, S. J., et al. Protocol: Optimising hydroponic growth systems for nutritional and physiological analysis of Arabidopsis thaliana and other plants. Plant Methods. 9 (1), 4 (2013).
- Nathoo, N. . Identification of putative plant defense genes using a novel hydroponic co-cultivation technique for studying plant-pathogen interaction. , (2015).
- Shaner, N. C., Campbell, R. E., Steinbach, P. A., Giepmans, B. N., Palmer, A. E., Tsien, R. Y. Improved monomeric red, orange and yellow fluorescent proteins derived from Discosoma sp. red fluorescent protein. Nat. Biotechnol. 22 (12), 1567-1572 (2004).
- Wise, A. A., Liu, H., Binns, A. N. Nucleic acid extraction from Agrobacterium strains. Methods Mol. Bio. 343, 67-76 (2006).
- Nolan, T., Hands, R. E., Bustin, S. A. Quantification of mRNA using real-time RT-PCR. Nat. Protoc. 1 (3), 1559-1582 (2006).
- Salter, M. G., Conlon, H. E. Extraction of plant RNA. Methods Mol. Bio. 362, 309-314 (2007).
- Bao, Y., Wang, S., Yang, X., Li, T., Xia, Y., Meng, X. Metabolomic study of the intervention effects of Shuihonghuazi Formula, a Traditional Chinese Medicinal formulae, on hepatocellular carcinoma (HCC) rats using performance HPLC/ESI-TOF-MS. J. Ethnopharmacol. 198, 468-478 (2017).
- Korves, T. M., Bergelson, J. A developmental response to pathogen infection in Arabidopsis. Plant Physiol. 133 (1), 339-347 (2003).
- Lyons, R., Rusu, A., Stiller, J., Powell, J., Manners, J. M., Kazan, K. Investigating the association between flowering time and defense in the Arabidopsis thaliana-Fusarium oxysporum interaction. PLoS ONE. 10 (6), e0127699 (2015).
- Badri, D. V., Weir, T. L., vander Lelie, D., Vivanco, J. M. Rhizosphere chemical dialogues: Plant-microbe interactions. Curr. Opin. Biotechnol. 20 (6), 642-650 (2009).
- Baerson, S. R., et al. Detoxification and transcriptome response in Arabidopsis seedlings exposed to the allelochemical benzoxazolin-2(3H)-one. J. Biol. Chem. 280 (23), 21867-21881 (2005).

