Een hydroponisch co-cultuursysteem voor gelijktijdige en systematische analyse van plant- / microbe moleculaire interacties en signalering
Summary
Het beschreven hydroponische cocultivatiesysteem ondersteunt intacte planten met metalen maasschermen en cocultivateert ze met bacteriën. Plantweefsel, bacteriën en gescheiden moleculen kunnen vervolgens afzonderlijk worden geoogst voor stroomafwaartse analyses, waarbij tegelijkertijd wordt toegelaten dat de moleculaire reacties van zowel plantaardige hosts als interactieve microben of microbiomen worden onderzocht.
Abstract
Een experimenteel ontwerp dat natuurlijke plant-microbe interacties nabootst, is zeer belangrijk om de complexe plant-microbe signaleringsprocessen te definiëren. Arabidopsis thaliana – Agrobacterium tumefaciens Biedt een uitstekend model systeem om bacteriële pathogenese en plantinteracties te bestuderen. Vorige studies van plant -Agrobacterium interacties hebben grotendeels gebaseerd op plantencel suspensie culturen, het kunstmatige wonden van planten, of de kunstmatige inductie van microbiële virulentie factoren of plantenverdediging door synthetische chemicaliën. Deze methoden onderscheiden zich echter van de natuurlijke signalering in planta , waar planten en microben herkennen en reageren op ruimtelijke en temporele manieren. Dit werk presenteert een hydroponisch cocultivatiesysteem waar intacte planten worden ondersteund door metalen maasschermen en gecocultiveerd met Agrobacterium . In dit cocultivatiesysteem bestaat geen synthetische fytohormoon of chemische stof die micr induceertObalieve virulentie of plantenverdediging wordt aangevuld. Het hydroponische cocultivatiesysteem lijkt op natuurlijke plant-microbe interacties en signaleert homeostase in planta . Plantwortels kunnen gescheiden worden van het medium dat Agrobacterium bevat , en de signalen en reacties van zowel de plantgastheren als de interactieve microben kunnen gelijktijdig en systematisch worden onderzocht. Bij een bepaald tijdstip / interval kunnen plantweefsels of bacteriën afzonderlijk worden geoogst voor diverse "omics" -analyses, die de kracht en werkzaamheid van dit systeem tonen. Het hydroponische cocultivatiesysteem kan gemakkelijk worden aangepast om te bestuderen: 1) de wederzijdse signalering van diverse plantmicrobysystemen, 2) signalering tussen een plantengas en meerdere microbiële soorten ( dwz microbiële consortia of microbiomen), 3) hoe voedingsstoffen en chemicaliën betrokken zijn In plant-microbe signalering, en 4) hoe microben interactie hebben met plantaardige hosts en bijdragen aan plantentolerantie voor biotische oR abiotische stress.
Introduction
Plantgerelateerde microben spelen belangrijke rollen in biogeochemische cycli, bioremediëring, vermindering van klimaatverandering, plantengroei en gezondheid en plantentolerantie tegen biotische en abiotische stress. Micro-organismen interageren met planten, zowel direct door plantencelwandcontact en indirect via chemische afscheiding en signalering 1 , 2 , 3 . Als sessiele organismen hebben planten directe en indirecte mechanismen ontwikkeld om infectie door pathogenen te weerstaan. Directe verdediging omvat structurele verdediging en de expressie van verdedigingseiwitten, terwijl indirecte verdediging de productie van secundaire plantenmetabolieten omvat en de aantrekkingskracht van organismen antagonistisch tegen invallende pathogenen 4 , 5 . Plantaardige wortel exudaten, afscheidingen, mucilages, mucigel en lysaten veranderen de fysisch-chemische eigenschappen van de rhizosfeer om te trekken of af te kerenMicroben naar hun gastheren 6 . De chemische samenstelling van wortelafscheiding is speciesspecifiek, en dient daarbij als selectief filter dat bepaalde micro-organismen in staat stelt om dergelijke verbindingen te herkennen om in de rhizosfeer 6 te bloeien. Zo kunnen compatibele microbiële soorten worden gestimuleerd om hun associaties te activeren en te verbeteren, hetzij tot voordeel of nadelen van de plantengas 1 .
Het begrijpen van plant-microbe-interacties in de rhizosfeer is essentieel voor het verbeteren van de productiviteit van het plant en het functioneren van het ecosysteem, aangezien een meerderheid van de microbiële en chemische blootstelling zich voordoet bij de wortelstructuur en de bodem-luchtinterface 2 , 6 , 7 , 8 . Het onderzoek naar ondergrondse plant-microbe interacties en wederzijdse reacties is echter een uitdaging als gevolg van het intrigerende Complexe en dynamische natuur en het ontbreken van geschikte experimentele modellen met natuurlijke wortelstructuur en plantmorfologie onder strak beheersbare groeivoorwaarden. Als een van de meest bestudeerde fytopathogenen infecteert Agrobacterium een breed scala aan planten met agrarisch en tuinbouw belang, waaronder kersen, appel, peer, druif en roos 9 . Agrobacterium is een belangrijk model organisme voor het interpreteren van plant-pathogeen interacties en is een krachtig instrument in plantentransformatie en planttechniek 10 , 11 , 12 , 13 , 14 .
Moleculaire plant- Agrobacterium interacties zijn al jarenlang bestudeerd en het huidige begrip van Agrobacterium pathogeniciteit is uitgebreid 9 ,F.> 11 , 15 , 16. De pathogeniciteit van Agrobacterium wordt grotendeels toegeschreven aan de evoluerende mogelijkheden van het opnemen van plantaardige afgeleid signalen, wat resulteert in de fijne modulatie van het virulentieprogramma en de cel-naar-celcommunicatie, zogenaamd quorum sensing 17 . Het Agrobacterium virulence programma wordt geregeld door verschillende signalen die beschikbaar zijn in de rhizosfeer en omvatten twee sets van 2-componenten systemen, het ChvG / I-systeem en het VirA / G-systeem. Acide condities in de rhizosfeer activeren de transcriptie van chvG / I , virA / G , En verscheidene andere genen die betrokken zijn bij Agrobacterium pathogeniciteit, waaronder virE0 , virE1 , virH1 , virH2 en genen van het type VI-afzettingssysteem (T6SS) 18. Plantafledde fenolverbindingen, waaronder acetosyringon (4'-hydroxy-3 ', 5 '-dimethoxyacetofenon), activeer de VIrA / G 2-componenten systeem via fosforylatie signaal mechanismen 19 . VirA / G activeert dan het volledige vir- regulon, wat resulteert in de overdracht en integratie van een ~ 20 kb bacterieel DNA fragment genaamd transfer DNA (T-DNA) van zijn tumorinducerende (Ti) plasmide in de plantencel 16 . T-DNA draagt genen die verantwoordelijk zijn voor de synthese van plantaardige hormonen indool-3-azijnzuur (IAA) ( iaaM en iaaH ) en cytokinine ( ipt ), en eenmaal uitgedrukt in plantencellen worden grote hoeveelheden van deze fytohormonen geproduceerd. Dit resulteert in abnormale weefsel proliferatie en ontwikkeling van plantentumor, bekend als kroon galziekte, die een chronisch en resurgent probleem is voor planten 9 , 11 , 20 . IAA handelt ook collectief met salicylzuur en gamma-aminoboterzuur om de Agrobacterium virulentie te onderdrukken of om Agrobacteriu te verminderen M kworum sensing (QS) 17 , 21 , 22 . Om deze onderdrukking tegen te gaan, draagt T-DNA ook genen voor opine biosynthese, die Agrobacterium quorum detecteert om de pathogeniteit van Agrobacterium te bevorderen en dient ook als voedingsbron voor het pathogeen 22 , 23 .
Ondanks een algemeen diepgaand begrip van Agrobacterium- plantinteracties en de resulterende T-DNA-overdracht in de plantaardige gastheer, zijn de complexe signaleringsgebeurtenissen in het beginstadium van interactie minder goed begrepen. Dit komt gedeeltelijk door de beperkingen van conventionele benaderingen om Agrobacterium- plantensignaal te onderzoeken. Plantcelsuspensieculturen en kunstmatige site-specifieke wonden worden vaak gebruikt voor het bestuderen van moleculaire plant-microbe-interacties 24 ,Ef,> 26 , 27. Echter, cel suspensies hebben typische plantmorfologie, in het bijzonder hebben plantaardige suspensiecellen geen wortelstructuren en wortel exudaten, die zeer belangrijk zijn voor het activeren van microbiële chemotaxis en virulentie 28 , 29. Het behoud van plantmorfologie En wortelstructuur is aangepakt door kunstmatig wondende planten, waardoor plaatselijke specifieke infectie wordt vergemakkelijkt, wat resulteert in het detecteren van geïnduceerde plantenverdediging gerelateerde genen in direct geïnfecteerd plantweefsel 30 , 31. Maar kunstmatige wonden is significant verschillend van pathogeeninfectie in de natuur , Met name omdat wonden leidt tot accumulatie van jasmonzuur (JA), die systematisch inbreuk maakt op natuurlijke plantensignalen en verdediging 26. Bovendien worden synthetische chemicaliën typisch gebruikt om kunstmatige reacties kunstmatig in te leidenOf pathogen virulentie. Hoewel de aanvulling van zulke chemische verbindingen die weerspiegelen van concentraties in planta mogelijk is, is deze toevoeging niet in de geringe mate van diffusie van wortel exudaten in de omringende rhizosfeer, die een chemotactische gradiënt genereert door microbes 28 , 32 genereert. Gezien de beperkingen van conventionele benaderingen om plant-microbe-interacties te bestuderen, kan de nauwkeurigheid en diepte van de verkregen gegevens worden belemmerd en restrictief, en de kennis die voortvloeit uit de conventionele benaderingen kan niet direct in planta vertaald worden. Veel aspecten van planten- Agrobacterium signalering zijn nog niet volledig begrepen, vooral in het vroege stadium van interacties, wanneer de ziektebeelden nog niet zijn ontwikkeld.
Om de beperkingen van conventionele benaderingen te wijzigen, presenteert dit werk een goedkope, goed beheersbare en flexibele hydroponische cOcultivatie systeem dat de onderzoekers in staat stelt diepere inzichten te krijgen in de complexe signalering- en reactiewegen bij de eerste fase van moleculaire plant-microbe interacties. Hydroponics is veel gebruikt om plantaardige voedingsstoffen, wortel exudaten, groeivoorwaarden en de effecten van metallische toxiciteit op planten 33 , 34 te bestuderen. Er zijn verschillende voordelen van hydroponische modellen, met inbegrip van de kleine ruimtelijke eisen, de toegankelijkheid van verschillende plantenweefsels, de strakke controle van de nutriënten / milieuomstandigheden en de bestrijding van de plaag / ziekte. Hydroponische systemen zijn ook minder beperkend voor plantengroei in vergelijking met agar / phytoagar plating technieken, die de groei meestal beperken na 2-3 weken. Belangrijk is dat het onderhoud van planten in de planten de natuurlijke wortesecretie vereiste die nodig is voor microbiële chemotaxis en virulentieinductie 8 , 29 . Het systeem beschrijftBed hier is eenvoudiger en minder arbeidsintensief dan de alternatieven 33 , 34 . Het maakt gebruik van minder onderdelen en vereist geen andere gereedschappen dan standaardscharen. Het gebruikt metaalnetwerk (in tegenstelling tot nylon 33 ) als een sterke ondersteuning voor plantengroei en een eenvoudige methode van beluchting onder steriele omstandigheden door schudden om microbiële groei te ondersteunen. Daarnaast kan het systeem metaalmaas van verschillende maten gebruiken om plantengroei te ondersteunen, die diverse plantensoorten herbergt zonder de breedte van hun wortels te beperken.
In het hier gepresenteerde hydroponische cocultivatiesysteem worden planten gekweekt in een steriel hydroponisch systeem waar de plantenwortels organische verbindingen scheiden die de groei van geïmpliceerde bacteriën ondersteunen. In dit cocultivatiesysteem worden geen kunstmatige chemicaliën, zoals plantaardige hormonen, verdedigingselectie, of virulentie-inducerende chemicaliën aangevuld, die de natuurlijke cel weerspiegeltSignalerende homeostase tijdens plant-microbe interacties. Met dit hydroponische cocultivatiesysteem was het mogelijk om gen expressie in Arabidopsis thaliana Col-0 wortelweefsel gelijktijdig te bepalen bij infectie door Agrobacterium , evenals de activatie van Agrobacterium genen bij cocultivatie met Arabidopsis . Verder werd aangetoond dat dit systeem geschikt is om Agrobacterium- bijlage aan plantenwortels te bestuderen, evenals het wortelgeheimprofiel van de plant, bij cocultivatie (infectie) met Agrobacterium ( Figuur 1 ).
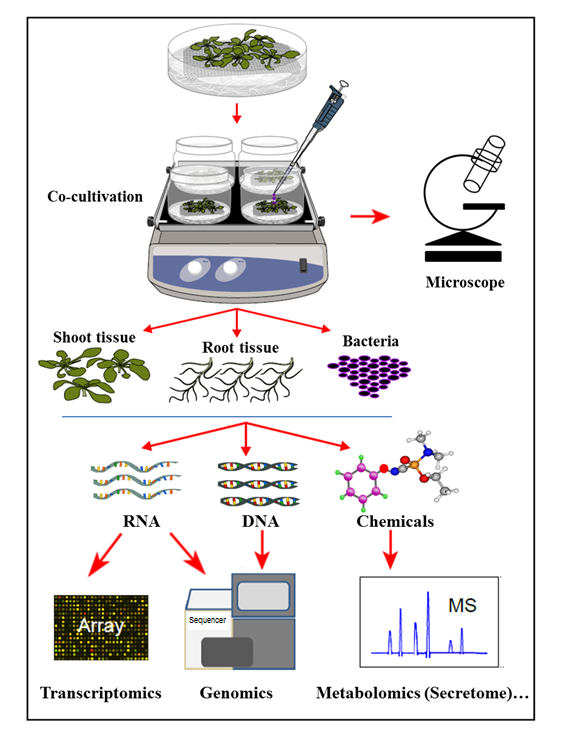
Figuur 1: Overzicht van het Hydroponic Cocultivation System, met voorbeeldanalyses. Planten worden op de maas gekweekt (scheuten boven het maas), met de wortels gedompeld in hydroponisch medium dat vervolgens met bacteriën fOf coculture. Plantweefsels en bacteriën worden vervolgens gescheiden voor gelijktijdige extracties en analyses. Dit cijfer is gewijzigd van referentie 35 .
Protocol
Representative Results
Discussion
Gezien de geleidelijke aard van wortelafscheiding, vinden de concentraties van virulentie-inducerende chemicaliën in planta en hun effecten op dynamische plant-microbe interacties plaats in ruimtelijke en temporale gradiënten. In dit hydroponische co-cultivatie systeem wordt geen synthetische fytohormoon of chemische stof die microbiële virulentie of plantenverdediging veroorzaakt, aangevuld. In tegenstelling hiermee, met behulp van conventionele benaderingen, voegt de toevoeging van synthetische chemicalië…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Wij danken Brian Weselowski en Alexander W. Eastman voor hun hulp en nuttige discussie. We willen ook Drs bedanken. Eugene W. Nester, Lingrui Zhang, Haitao Shen, Yuhai Cui en Greg Thorn voor hun hulp, nuttige discussies en kritisch lezen van het manuscript. Dit onderzoek is gefinancierd door Agriculture and Agri-Food Canada, Growing Forward-AgriFlex (RBPI nummer 2555) en Growing Forward II project nummer 1670, dat door de auteurs als onderdeel van hun taken wordt uitgevoerd. Deze studie werd ook gedeeltelijk gefinancierd door de Natuurwetenschappen en Technische Onderzoeksraad van Canada (NSERC) Discovery Grant RGPIN-2015-06052 toegekend aan ZC Yuan.
Materials
| plant seeds (Arabidopsis thaliana Col-0) | Arabidopsis Biological Resource Centre | CS7000 | https://abrc.osu.edu/order-stocks |
| bacteria (Agrobacterium tumefaciens C58) | University of Washington | N/A | |
| labeled bacteria | in-house | optional, depends on downstream analyses | |
| vortex | (various) | ||
| microcentrifuge tubes | (various) | ||
| microcentrifuge | (various) | ||
| 5% sodium hypochlorite | (various) | ||
| double distilled water | (various) | ||
| autoclave | (various) | ||
| micropipette | (various) | ||
| 70 % ethanol | (various) | ||
| Murashige and Skoog (MS) basal salts | Sigma-Aldrich | M5524 | |
| sucrose | (various) | ||
| MES | (various) | ||
| B5 vitamin mix | Sigma-Aldrich | G1019 | |
| phytoagar | (various) | ||
| deep Petri dishes | (various) | ||
| stainless steel mesh | Ferrier Wire Goods Company Ltd | N/A | grade: 304; mesh count: 40 × 40; wire DIA: 0.01 |
| micropore tape, 1 inch | 3M | 1530-1 | |
| diurnal growth chamber | (various) | ||
| cylindrical glass tanks, 100 × 80 mm | Pyrex | 3250 | other sizes can be used, in which case liquid content may need adjustment |
| flow hood | (various) | ||
| forcepts | (various) | ||
| yeast extract | (various) | ||
| tryptone | (various) | ||
| MgSO4 | (various) | ||
| shaking incubator | (various) | ||
| spectrophotometer | (various) | ||
| NaCl | (various) | ||
| shaker | (various) | ||
| scissors | (various) | optional, depends on downstream analyses | |
| fluorescence microscope | (various) | optional, depends on downstream analyses | |
| microscope slides and cover slips | (various) | optional, depends on downstream analyses | |
| nail polish | (various) | optional, depends on downstream analyses | |
| Bacterial RNA extraction kit | (various) | optional, depends on downstream analyses | |
| plant RNA extraction kit (RNeasy Plant Mini Kit) | Qiagen | 74903 or 74904 | optional, depends on downstream analyses |
| material and equipment for RT-qPCR | (various) | optional, depends on downstream analyses | |
| material and equipment for microarray analysis | (various) | optional, depends on downstream analyses | |
| liquid nitrogen | (various) | optional, depends on downstream analyses | |
| mortar and pestle | (various) | optional, depends on downstream analyses | |
| 0.2 µm pore filter | (various) | optional, depends on downstream analyses | |
| 50 mL conical tubes | (various) | optional, depends on downstream analyses | |
| freeze dryer | (various) | optional, depends on downstream analyses | |
| sealable test tubes | (various) | optional, depends on downstream analyses | |
| ethyl acetate | (various) | optional, depends on downstream analyses | |
| nitrogen gas | (various) | optional, depends on downstream analyses | |
| material and equipment for HPLC | (various) | optional, depends on downstream analyses | |
| material and equipment for ESI-TOF-MS | (various) | optional, depends on downstream analyses |
References
- Lambers, H., Mougel, C., Jaillard, B., Hinsinger, P. Plant-microbe-soil interactions in the rhizosphere: An evolutionary perspective. Plant Soil. 321 (1), 83-115 (2009).
- Philippot, L., Raaijmakers, J. M., Lemanceau, P., vander Putten, W. H. Going back to the roots: The microbial ecology of the rhizosphere. Nat. Rev. Microbiol. 11 (11), 789-799 (2013).
- Somers, E., Vanderleyden, J., Srinivasan, M. Rhizosphere bacterial signalling: A Love Parade beneath our feet. Crit. Rev. Microbiol. 30 (4), 205-240 (2004).
- Barah, P., Winge, P., Kusnierczyk, A., Tran, D. H., Bones, A. M. Molecular signatures in Arabidopsis thaliana in response to insect attack and bacterial infection. PLoS ONE. 8 (3), (2013).
- Zhang, J., Zhou, J. -. M. Plant immunity triggered by microbial molecular signatures. Mol. Plant. 3 (5), 783-793 (2010).
- Paterson, E., Gebbing, T., Abel, C., Sim, A., Telfer, G. Rhizodeposition shapes rhizosphere microbial community structure in organic soil. New Phytol. 173 (3), 600-610 (2006).
- Hartmann, A., Schmid, M., van Tuinen, D., Berg, G. Plant-driven selection of microbes. Plant Soil. 321 (1-2), 235-257 (2008).
- Micallef, S. A., Shiaris, M. P., Colon-Carmona, A. Influence of Arabidopsis thaliana accessions on rhizobacterial communities and natural variation in root exudates. J. Exp. Bot. 60 (6), 1729-1742 (2009).
- Burr, T., Otten, L. Crown gall of grape: Biology and disease management. Annu. Rev. Phytopathol. 37, 53-80 (2001).
- Clough, S. J., Bent, A. F. Floral dip: A simplified method for Agrobacterium-mediated transformation of Arabidopsis thaliana. Plant J. 16 (6), 735-743 (1998).
- Binns, A. N., Costantino, P., Spaink, H. P., Kondorosi, A., Hooykaas, P. J. J. The Agrobacterium oncogenes. The Rhizobiaceae. , (1998).
- Gheysen, G., Angenon, G., Van Montagu, M., Lindsey, K. Agrobacterium-mediated plant transformation: a scientifically intriguing story with significant applications. Transgenic Plant Research. , (1998).
- Valvekens, D., Von Montagu, M. V., Van Lijsebettens, M. Agrobacterium tumefaciens-mediated transformation of Arabidopsis thaliana root explants by using kanamycin selection. Proc. Natl. Acad. Sci. USA. 85 (15), 5536-5540 (1988).
- Krysan, P. J., Young, J. C., Sussman, M. R. T-DNA as an insertional mutagen in Arabidopsis. Plant Cell. 11 (12), 2283 (1999).
- Chilton, M. D., et al. Stable incorporation of plasmid DNA into higher plant cells: The molecular basis of crown gall tumorigenesis. Cell. 11 (2), 263-271 (1977).
- Gelvin, S. B. Agrobacterium in the genomics age. Plant Physiol. 150 (4), 1665-1676 (2009).
- Subramoni, S., Nathoo, N., Klimov, E., Yuan, Z. -. C. Agrobacterium tumefaciens responses to plant-derived signaling molecules. Front. Plant Sci. 5, 322 (2014).
- Yuan, Z., Liu, P., Saenkham, P., Kerr, K., Nester, E. W. Transcriptome profiling and functional analysis of Agrobacterium tumefaciens reveals a general conserved response to acidic conditions (pH 5.5) and a complex acid-mediated signaling involved in Agrobacterium-plant interactions. J. Bacteriol. 190 (2), 494-507 (2008).
- Stachel, S. E., Messens, E., Van Montagu, M., Zambryski, P. Identification of the signal molecules produced by wounded plant cells that activate T-DNA transfer in Agrobacterium tumefaciens. Nature. 318 (6047), 624-629 (1985).
- Memelink, J., de Pater, B. S., Hoge, J. H. C., Schilperoort, R. A. T-DNA hormone biosynthetic genes: Phytohormones and gene expression in plants. Dev. Genet. 8 (5-6), 321-337 (1987).
- Yuan, Z. -. C., et al. The plant signal salicylic acid shuts down expression of the vir regulon and activates quormone-quenching genes in Agrobacterium. Proc. Natl. Acad. Sci. USA. 104 (28), 11790-11795 (2007).
- Yuan, Z. -. C., Haudecoeur, E., Faure, D., Kerr, K. F., Nester, E. W. Comparative transcriptome analysis of Agrobacterium tumefaciens in response to plant signal salicylic acid, indole-3-acetic acid and γ-amino butyric acid reveals signalling cross-talk and Agrobacterium-plant co-evolution. Cell. Microbiol. 10 (11), 2339-2354 (2008).
- Li, P. L., Farrand, S. K. The replicator of the nopaline-type Ti plasmid pTiC58 is a member of the repABC family and is influenced by the TraR-dependent quorum-sensing regulatory system. J. Bacteriol. 182 (1), 179-188 (2000).
- Atkinson, M. M., Huang, J., Knopp, J. A. Hypersensitivity of suspension-cultured tobacco cells to pathogenic bacteria. Phytopathology. 75 (11), 1270-1274 (1985).
- Veena, ., Jiang, H., Doerge, R. W., Gelvin, S. B. Transfer of T-DNA and Vir proteins to plant cells by Agrobacterium tumefaciens induces expression of host genes involved in mediating transformation and suppresses host defense gene expression. Plant J. 35 (2), 219-236 (2003).
- León, J., Rojo, E., Sanchez-Serrano, J. J. Wound signalling in plants. J. Exp. Bot. 52 (354), 1-9 (2001).
- Ditt, R. F., Kerr, K. F., de Figueiredo, P., Delrow, J., Comai, L., Nester, E. W. The Arabidopsis thaliana transcriptome in response to Agrobacterium tumefaciens. Mol. Plant Microbe In. 19 (6), 665-681 (2006).
- Mandimba, G., Heulin, T., Bally, R., Guckert, A., Balandreau, J. Chemotaxis of free-living nitrogen-fixing bacteria towards maize mucilage. Plant Soil. 90 (1-3), 129-139 (1986).
- Rudrappa, T., Czymmek, K. J., Pare, P. W., Bais, H. P. Root-secreted malic acid recruits beneficial soil bacteria. Plant Physiol. 148 (3), 1547-1556 (2008).
- Lee, C. W., et al. Agrobacterium tumefaciens promotes tumor induction by modulating pathogen defense in Arabidopsis thaliana. Plant Cell. 21 (9), 2948-2962 (2009).
- Reymond, P., Weber, H., Damond, M., Farmer, E. E. Differential gene expression in response to mechanical wounding and insect feeding in Arabidopsis. Plant Cell. 12 (5), 707-720 (2000).
- Matthysse, A. G. Attachment of Agrobacterium to plant surfaces. Front. Plant Sci. 5, 252 (2014).
- Alatorre-Cobos, F., et al. An improved, low-cost, hydroponic system for growing Arabidopsis and other plant species under aseptic conditions. BMC Plant Biol. 14 (1), 69 (2014).
- Conn, S. J., et al. Protocol: Optimising hydroponic growth systems for nutritional and physiological analysis of Arabidopsis thaliana and other plants. Plant Methods. 9 (1), 4 (2013).
- Nathoo, N. . Identification of putative plant defense genes using a novel hydroponic co-cultivation technique for studying plant-pathogen interaction. , (2015).
- Shaner, N. C., Campbell, R. E., Steinbach, P. A., Giepmans, B. N., Palmer, A. E., Tsien, R. Y. Improved monomeric red, orange and yellow fluorescent proteins derived from Discosoma sp. red fluorescent protein. Nat. Biotechnol. 22 (12), 1567-1572 (2004).
- Wise, A. A., Liu, H., Binns, A. N. Nucleic acid extraction from Agrobacterium strains. Methods Mol. Bio. 343, 67-76 (2006).
- Nolan, T., Hands, R. E., Bustin, S. A. Quantification of mRNA using real-time RT-PCR. Nat. Protoc. 1 (3), 1559-1582 (2006).
- Salter, M. G., Conlon, H. E. Extraction of plant RNA. Methods Mol. Bio. 362, 309-314 (2007).
- Bao, Y., Wang, S., Yang, X., Li, T., Xia, Y., Meng, X. Metabolomic study of the intervention effects of Shuihonghuazi Formula, a Traditional Chinese Medicinal formulae, on hepatocellular carcinoma (HCC) rats using performance HPLC/ESI-TOF-MS. J. Ethnopharmacol. 198, 468-478 (2017).
- Korves, T. M., Bergelson, J. A developmental response to pathogen infection in Arabidopsis. Plant Physiol. 133 (1), 339-347 (2003).
- Lyons, R., Rusu, A., Stiller, J., Powell, J., Manners, J. M., Kazan, K. Investigating the association between flowering time and defense in the Arabidopsis thaliana-Fusarium oxysporum interaction. PLoS ONE. 10 (6), e0127699 (2015).
- Badri, D. V., Weir, T. L., vander Lelie, D., Vivanco, J. M. Rhizosphere chemical dialogues: Plant-microbe interactions. Curr. Opin. Biotechnol. 20 (6), 642-650 (2009).
- Baerson, S. R., et al. Detoxification and transcriptome response in Arabidopsis seedlings exposed to the allelochemical benzoxazolin-2(3H)-one. J. Biol. Chem. 280 (23), 21867-21881 (2005).

