식물 / 미생물 분자 상호 작용 및 신호의 동시 및 체계적 분석을위한 수경 공동 공동 배양 시스템
Summary
기술 된 수경 공동 배양 시스템은 금속 메쉬 스크린이있는 온전한 식물을지지하고 박테리아와 함께 배양한다. 식물 조직, 박테리아 및 분비 된 분자는 하류 분석을 위해 따로 따로 수확 할 수 있으며 동시에 식물 숙주와 상호 작용하는 미생물 또는 미생물의 분자 반응을 조사 할 수 있습니다.
Abstract
복잡한 식물 – 미생물 신호 처리 과정을 묘사하기 위해서는 자연 식물 – 미생물 상호 작용을 모방 한 실험 설계가 매우 중요합니다. 애기 장대 – Agrobacterium tumefaciens 박테리아의 병인 및 식물 상호 작용을 연구하기위한 우수한 모델 시스템을 제공합니다. 식물 – 아그로 박테 리움 상호 작용에 대한 이전의 연구는 주로 식물 세포 현탁 배양, 식물의 인공 상처 또는 합성 화학 물질에 의한 미생물 독성 인자 또는 식물 방제 의 인공 유도에 크게 의존해왔다. 그러나 이러한 방법은 식물과 미생물이 공간적, 시간적 방식으로인지하고 반응하는 식물의 자연 신호 와 구별된다. 이 작품은 손상되지 않은 식물이 금속 메쉬 스크린에 의해지지되고 아그로 박테 리움 과 함께 배양되는 수경 공동 배양 시스템을 제시합니다. 이 공동 배양 시스템에서, 합성 피토 호르몬 또는 micr을 유도하는 화학 물질obial 독성 또는 식물 방어는 보충된다. hydroponic cocultivation 시스템 은 planta의 자연 식물 – 미생물 상호 작용 및 신호 항상성과 매우 유사합니다. 식물 뿌리는 아그로 박테 리움을 함유하는 배지로부터 분리 될 수 있고, 식물 숙주와 상호 작용하는 미생물의 신호 및 반응은 동시에 체계적으로 조사 될 수있다. 특정 시점 / 간격에서, 식물 조직 또는 박테리아는 다양한 "omics"분석을 위해 별도로 수확 될 수있어이 시스템의 효능과 효과를 입증합니다. 수경 공동 배양 시스템은 1) 다양한 식물 미생물 시스템의 상호 신호 전달, 2) 식물 숙주와 여러 미생물 종 ( 예 : 미생물 협회 또는 미생물) 간의 신호 전달, 3) 영양소와 화학 물질의 관련성 연구 4) 미생물이 식물 숙주와 어떻게 상호 작용하고 생물학적 인 o에 대한 식물 내성에 기여하는지r 비 생물 적 스트레스.
Introduction
식물과 관련된 미생물은 생물 지구 화학적 순환, 생물학적 복원, 기후 변화 완화, 식물 성장 및 건강, 생물학적 및 비 생물 적 스트레스에 대한 식물 내성에 중요한 역할을한다. 미생물은 식물 세포 벽 접촉을 통해 간접적으로 화학 분비 및 신호 1 , 2 , 3을 통해 식물과 상호 작용합니다. 침착성 유기체로서 식물은 병원균에 의한 감염을 막기위한 직접 및 간접적 인 기작을 개발해 왔습니다. 직접 방어에는 구조적 방어와 방어 단백질의 발현이 포함되지만 간접 방어에는 2 차 식물 대사 산물 생성 및 침입 병원체에 대항하는 유기체의 인력이 포함됩니다 4 , 5 . 식물에서 추출한 뿌리 삼출물, 분비물, 점액, 점액 및 용 해물은 rhizosphere의 물리 화학적 성질을 변화시켜 끌어들이거나 밀어 낸다.그들의 숙주에 대한 미생물 6 . 뿌리 분비물의 화학적 조성은 종 특이 적이며, 이로 인해 rhizosphere에서 이러한 화합물을 인식 할 수있는 특정 미생물을 허용하는 선택 필터 역할을합니다 6 . 따라서, 호환 가능한 미생물 종은 자극되어 식물 숙주 1 의 이익 또는 손상에 대한 그들의 결합을 활성화시키고 향상시킬 수있다.
근권에서 식물 – 미생물 상호 작용을 이해하는 것은 미생물 및 화학 물질 노출의 대부분이 뿌리 구조와 토양 – 공기 경계 2 , 6 , 7 , 8 에서 발생하기 때문에 식물 생산성과 생태계 기능을 향상시키는 데 중요합니다. 그러나 지하 식물의 미생물 상호 작용과 상호 반응에 대한 조사는 흥미롭게도 복잡하고 역동적 인 성질 및 단단히 조절 가능한 성장 조건 하에서의 천연 뿌리 구조 및 식물 형태와 적합한 실험 모델의 결여. 가장 많이 연구 phytopathogens 중 하나 인 아그로 박테 리움 체리, 사과, 배, 포도를 포함한 농업과 원예 중요성, 식물의 넓은 범위를 감염, 9 상승했다. 아그로 박테리아 는 식물 – 병원체 상호 작용을 이해하는 중요한 모델 유기체이며 식물 형질 전환 및 식물 공학 10 , 11 , 12 , 13 , 14 에서 강력한 도구입니다.
분자 공장 – 아그로 박테 리움 상호 작용은 물론, 수십 년 동안 연구 및 아그로 박테 리움의 병원성의 현재 이해는 9 광범위 한F "> 11, 15, 16. 아그로 병원성은 대부분 17 센싱 그 독성 프로그램 및 세포 간 통신의 미세 조정의 결과, 식물 유래의 신호를 지각의 발전 능력에 소위 정수를 때문이다. 아그로 박테 리움 독성 프로그램은 rhizosphere에서 이용 가능한 몇 가지 신호에 의해 조절되며 ChvG / I 시스템과 VirA / G 시스템의 2 가지 세트로 구성된다. rhizosphere의 산성 조건은 chvG / I , virA / G 의 전사를 활성화시킨다 , virE0 , virE1 , virH1 , virH2 및 VI 형 분비 시스템 (T6SS) 18의 유전자를 포함한 Agrobacterium 병원성에 관여하는 여러 다른 유전자를 포함한다. 아세토 오손 (acetosyringone) (4'- 히드 록시 -3 ', 5 '- 디메 톡시 아세토 페논), V인산화 신호 전달 메커니즘을 통한 irA / G 2- 성분 시스템 19 . VirA / G는 전체 vir 레 귤론을 활성화시켜 종양 유도 (Ti) 플라스미드에서 전사 핵 (T-DNA)으로 알려진 ~ 20kb의 박테리아 DNA 조각을 식물 핵 16 으로 옮기고 통합시킵니다. T-DNA는 식물 호르몬 인 indole -3-acetic acid (IAA) ( iaaM 과 iaaH )와 cytokinin ( ipt )의 합성에 관여하는 유전자를 가지고 있으며 일단 식물 세포에서 발현되면 많은 양의 식물 호르몬이 생산됩니다. 이것은 비정상적인 조직 증식과 식물 종양 발달을 일으키는데 이는 크라운 담즙병 (crown gall disease)으로 알려져 있으며 이는 식물 9 , 11 , 20 의 만성적이고 재발적인 문제입니다. IAA는 아그로 독성을 억제하거나 감소 Agrobacteriu 살리실산 및 감마 – 아미노 부티르산으로 집단적 행동 m 쿼럼 감지 (QS) ( 17 , 21 , 22) . 이 억압 카운터, T-DNA는 아그로 병원성을 촉진 아그로 정수 감지를 활성화하고, 또한 병원체 22, 23 영양원 역할 의견을 갖다 생합성을위한 유전자를 운반한다.
아그로 박테 리움 – 식물 간 상호 작용에 대한 전반적인 깊은 이해와 식물 숙주로의 T-DNA 전달로 인해 초기 상호 작용에서 복잡한 신호 전달 사건이 덜 잘 이해된다. 이것은 Agrobacterium -plant signaling을 조사하기위한 기존의 접근법의 한계로 인해 부분적으로 발생합니다. 식물 세포 현탁 배양 및 인공 사이트 별 상처는 일반적으로 분자 식물 – 미생물 상호 작용 (24)을 연구하는 데 사용됩니다. EF "> (26), (27)는 그러나, 세포 현탁액은 일반적인 식물의 형태를 부족, 특히 식물 서스펜션 세포가 미생물 화성 및 독성 (28), (29) 활성화를위한 매우 중요한 루트 구조와 뿌리 삼출물을하지 않아도 식물 형태의 유지 보수. 루트 구조는 직접 감염된 식물 조직 (30), (31)에 의한 식물 방어 관련 유전자의 검출 결과, 사이트 별 감염을 용이하게 인위적으로 상처를 입는 식물에 의해 해결되었습니다. 그러나, 인공 상처는 자연의 병원균 감염 큰 차이가 특히 상처가 자스몬 산 (JA) 축적을 유도하여 천연 식물 신호 전달 및 방어를 체계적으로 방해한다 .26 또한 합성 화학 물질은 인공적으로 식물 숙주 반응을 유도하는데 사용된다또는 병원체 독성. 란타에서의 반사 농도와 같은 화학 물질의 보충이 가능하지만, 이러한 보충 서서히 미생물 (28), (32)에 의해 감지 화성 구배를 생성 주변 근권로 루트 배설물의 확산을 고려하지 않는다. 식물 – 미생물 상호 작용, 정확성과 방해하고 제한 될 수 얻은 데이터의 깊이, 그리고 란타에서 직접 번역하지 않을 수 있습니다 기존의 접근 방식에서 생성 된 지식을 연구하는 기존의 접근 방식의 한계를 감안할 때. 식물 – 아그로 박테 리움 신호 전달의 많은 양상은 아직 완전히 이해되지 않았는데, 특히 질병 증상이 아직 나타나지 않은 초기 상호 작용 단계에서 그렇다.
종래의 접근법의 한계를 수정하기 위해,이 연구는 저렴하고, 단단히 조절 가능하며, 유연한 수경 첨가제 인 c연구원이 분자 식물 – 미생물 상호 작용의 초기 단계에서 복잡한 신호 전달 및 반응 경로에 대한 더 깊은 통찰력을 얻을 수 있도록하는 시스템입니다. 수경법은 식물 영양소, 뿌리 삼출물, 성장 조건 및 식물에 금속 독성의 영향을 연구하기 위해 널리 사용되었습니다 33 , 34 . 작은 공간 요구 사항, 다양한 식물 조직의 접근 가능성, 영양 / 환경 조건의 엄격한 제어 및 해충 / 질병 통제를 포함하여 수경법 모델의 몇 가지 장점이 있습니다. 수경 시스템은 한천 / 피토 아르 (phytoagar) 도금 기술과 비교하여 식물 생장을 제한하지 않으며, 2-3 주 후에 성장을 제한합니다. 중요한 것은, 전체 공장 구조의 유지 보수는 미생물 화성 및 독성 유도 (8), (29)에 필요한 천연 루트 분비를 촉진한다. 시스템 설명여기 침대는 대안 33 , 34 보다 간단하고 노동 집약적입니다. 더 적은 부품을 사용하고 표준 가위 이외의 도구를 필요로하지 않습니다. 이 제품은 식물 성장에 대한 강력한 지지제이자 미생물 성장을 지원하기위한 흔들림을 통한 멸균 조건 하에서의 단순 통기 방법 인 금속 망 (나일론 33 과 대조적으로)을 사용합니다. 또한이 시스템은 뿌리의 너비를 제한하지 않으면 서 다양한 식물 종을 수용 할 수있는 식물 성장을 지원하기 위해 다양한 크기의 금속 망을 사용할 수 있습니다.
여기에 제시된 수경 공동 배양 시스템에서, 식물은 접종 된 박테리아의 성장을지지하는 유기 화합물을 식물 뿌리가 분비하는 무균 수경계에서 재배된다. 이 공동 배양 시스템에서는 식물 세포 내 호르몬, 방어력 유발 물질 또는 독성 유발 화학 물질과 같은 인공 화학 물질이 보완되지 않으며 자연 세포를 반영합니다식물 – 미생물 상호 작용 동안 신호 항상성. 이 수경 공동 배양 시스템에 의해, 동시에 유전자를 아그로 박테 리움에 의한 감염에 따라 애기 장대 골-0 뿌리 조직에서의 발현뿐만 아니라, 애기 장대와 공동 배양시 아그로 유전자의 활성을 결정할 수 있었다. 이 시스템은 아그로 박테 리움 ( Agrobacterium )과 동시 배양 (감염)시 식물 뿌리의 분비 프로파일뿐 아니라 식물 뿌리에 대한 아그로 박테 리움의 부착을 연구하는데 적합하다는 것이 추가로 증명되었다 ( 그림 1 ).
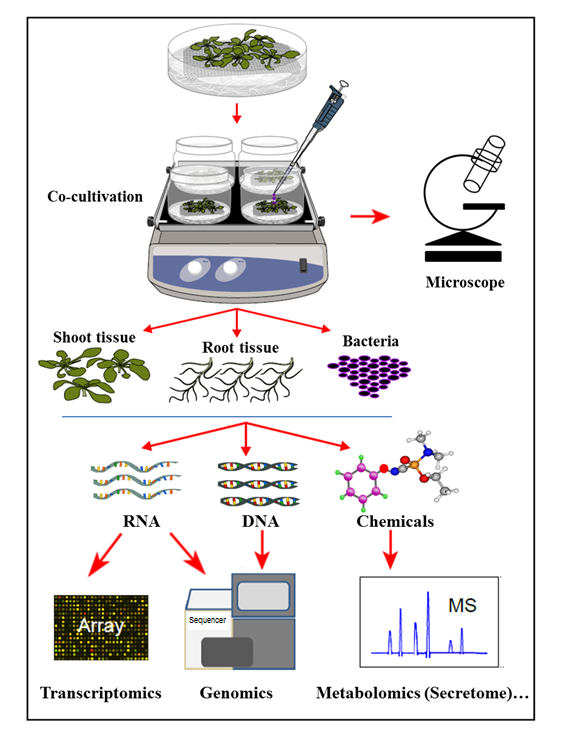
그림 1 : 샘플 분석과 함께 수경 공동 배양 시스템의 개요. 식물은 메쉬 (메쉬 위의 싹)의 꼭대기에서 자랍니다. 수경 재배액에 담근 다음 뿌리를 수경 재배 배지에 넣고 박테리아 f또는 공동 배양. 식물 조직과 박테리아는 동시에 추출 및 분석을 위해 분리됩니다. 이 수치는 참고 35 에서 수정되었습니다.
Protocol
Representative Results
Discussion
뿌리 분비의 점진적인 특성을 감안할 때, planta에서 생산 된 독성 유발 화학 물질의 농도와 역동적 인 식물 – 미생물 상호 작용에 미치는 영향은 공간적 및 시간적 변화에 따라 발생합니다. 이 수경 재성 공동 재배 시스템에는 미생물 독성이나 식물 방어를 유도하는 합성 식물 호르몬이나 화학 물질이 보충되지 않습니다. 대조적으로, 종래의 접근법을 사용하여, 아세토 독 온온 (acetosyr…
Disclosures
The authors have nothing to disclose.
Acknowledgements
그들의 도움과 유용한 토론에 대해 Brian Weselowski와 Alexander W. Eastman에게 감사드립니다. 우리는 또한 Drs. Eugene W. Nester, Lingrui Zhang, Haitao Shen, Yuhai Cui, Greg Thorn의 도움을 받았고 유용한 토론과 원고를 읽었습니다. 이 연구는 농업 및 농업 식품 캐나다 (Agriculture and Agri-Food Canada), Growing Forward-AgriFlex (RBPI 번호 2555) 및 Growing Forward II 프로젝트 번호 1670의 자금 지원을 받아 작성되었습니다. 이 연구는 또한 ZC Yuan에게 수여 된 캐나다 자연 과학 및 공학 연구 협의회 (NSERC) 디스커버리 그랜트 RGPIN-2015-06052에 의해 부분적으로 지원되었습니다.
Materials
| plant seeds (Arabidopsis thaliana Col-0) | Arabidopsis Biological Resource Centre | CS7000 | https://abrc.osu.edu/order-stocks |
| bacteria (Agrobacterium tumefaciens C58) | University of Washington | N/A | |
| labeled bacteria | in-house | optional, depends on downstream analyses | |
| vortex | (various) | ||
| microcentrifuge tubes | (various) | ||
| microcentrifuge | (various) | ||
| 5% sodium hypochlorite | (various) | ||
| double distilled water | (various) | ||
| autoclave | (various) | ||
| micropipette | (various) | ||
| 70 % ethanol | (various) | ||
| Murashige and Skoog (MS) basal salts | Sigma-Aldrich | M5524 | |
| sucrose | (various) | ||
| MES | (various) | ||
| B5 vitamin mix | Sigma-Aldrich | G1019 | |
| phytoagar | (various) | ||
| deep Petri dishes | (various) | ||
| stainless steel mesh | Ferrier Wire Goods Company Ltd | N/A | grade: 304; mesh count: 40 × 40; wire DIA: 0.01 |
| micropore tape, 1 inch | 3M | 1530-1 | |
| diurnal growth chamber | (various) | ||
| cylindrical glass tanks, 100 × 80 mm | Pyrex | 3250 | other sizes can be used, in which case liquid content may need adjustment |
| flow hood | (various) | ||
| forcepts | (various) | ||
| yeast extract | (various) | ||
| tryptone | (various) | ||
| MgSO4 | (various) | ||
| shaking incubator | (various) | ||
| spectrophotometer | (various) | ||
| NaCl | (various) | ||
| shaker | (various) | ||
| scissors | (various) | optional, depends on downstream analyses | |
| fluorescence microscope | (various) | optional, depends on downstream analyses | |
| microscope slides and cover slips | (various) | optional, depends on downstream analyses | |
| nail polish | (various) | optional, depends on downstream analyses | |
| Bacterial RNA extraction kit | (various) | optional, depends on downstream analyses | |
| plant RNA extraction kit (RNeasy Plant Mini Kit) | Qiagen | 74903 or 74904 | optional, depends on downstream analyses |
| material and equipment for RT-qPCR | (various) | optional, depends on downstream analyses | |
| material and equipment for microarray analysis | (various) | optional, depends on downstream analyses | |
| liquid nitrogen | (various) | optional, depends on downstream analyses | |
| mortar and pestle | (various) | optional, depends on downstream analyses | |
| 0.2 µm pore filter | (various) | optional, depends on downstream analyses | |
| 50 mL conical tubes | (various) | optional, depends on downstream analyses | |
| freeze dryer | (various) | optional, depends on downstream analyses | |
| sealable test tubes | (various) | optional, depends on downstream analyses | |
| ethyl acetate | (various) | optional, depends on downstream analyses | |
| nitrogen gas | (various) | optional, depends on downstream analyses | |
| material and equipment for HPLC | (various) | optional, depends on downstream analyses | |
| material and equipment for ESI-TOF-MS | (various) | optional, depends on downstream analyses |
References
- Lambers, H., Mougel, C., Jaillard, B., Hinsinger, P. Plant-microbe-soil interactions in the rhizosphere: An evolutionary perspective. Plant Soil. 321 (1), 83-115 (2009).
- Philippot, L., Raaijmakers, J. M., Lemanceau, P., vander Putten, W. H. Going back to the roots: The microbial ecology of the rhizosphere. Nat. Rev. Microbiol. 11 (11), 789-799 (2013).
- Somers, E., Vanderleyden, J., Srinivasan, M. Rhizosphere bacterial signalling: A Love Parade beneath our feet. Crit. Rev. Microbiol. 30 (4), 205-240 (2004).
- Barah, P., Winge, P., Kusnierczyk, A., Tran, D. H., Bones, A. M. Molecular signatures in Arabidopsis thaliana in response to insect attack and bacterial infection. PLoS ONE. 8 (3), (2013).
- Zhang, J., Zhou, J. -. M. Plant immunity triggered by microbial molecular signatures. Mol. Plant. 3 (5), 783-793 (2010).
- Paterson, E., Gebbing, T., Abel, C., Sim, A., Telfer, G. Rhizodeposition shapes rhizosphere microbial community structure in organic soil. New Phytol. 173 (3), 600-610 (2006).
- Hartmann, A., Schmid, M., van Tuinen, D., Berg, G. Plant-driven selection of microbes. Plant Soil. 321 (1-2), 235-257 (2008).
- Micallef, S. A., Shiaris, M. P., Colon-Carmona, A. Influence of Arabidopsis thaliana accessions on rhizobacterial communities and natural variation in root exudates. J. Exp. Bot. 60 (6), 1729-1742 (2009).
- Burr, T., Otten, L. Crown gall of grape: Biology and disease management. Annu. Rev. Phytopathol. 37, 53-80 (2001).
- Clough, S. J., Bent, A. F. Floral dip: A simplified method for Agrobacterium-mediated transformation of Arabidopsis thaliana. Plant J. 16 (6), 735-743 (1998).
- Binns, A. N., Costantino, P., Spaink, H. P., Kondorosi, A., Hooykaas, P. J. J. The Agrobacterium oncogenes. The Rhizobiaceae. , (1998).
- Gheysen, G., Angenon, G., Van Montagu, M., Lindsey, K. Agrobacterium-mediated plant transformation: a scientifically intriguing story with significant applications. Transgenic Plant Research. , (1998).
- Valvekens, D., Von Montagu, M. V., Van Lijsebettens, M. Agrobacterium tumefaciens-mediated transformation of Arabidopsis thaliana root explants by using kanamycin selection. Proc. Natl. Acad. Sci. USA. 85 (15), 5536-5540 (1988).
- Krysan, P. J., Young, J. C., Sussman, M. R. T-DNA as an insertional mutagen in Arabidopsis. Plant Cell. 11 (12), 2283 (1999).
- Chilton, M. D., et al. Stable incorporation of plasmid DNA into higher plant cells: The molecular basis of crown gall tumorigenesis. Cell. 11 (2), 263-271 (1977).
- Gelvin, S. B. Agrobacterium in the genomics age. Plant Physiol. 150 (4), 1665-1676 (2009).
- Subramoni, S., Nathoo, N., Klimov, E., Yuan, Z. -. C. Agrobacterium tumefaciens responses to plant-derived signaling molecules. Front. Plant Sci. 5, 322 (2014).
- Yuan, Z., Liu, P., Saenkham, P., Kerr, K., Nester, E. W. Transcriptome profiling and functional analysis of Agrobacterium tumefaciens reveals a general conserved response to acidic conditions (pH 5.5) and a complex acid-mediated signaling involved in Agrobacterium-plant interactions. J. Bacteriol. 190 (2), 494-507 (2008).
- Stachel, S. E., Messens, E., Van Montagu, M., Zambryski, P. Identification of the signal molecules produced by wounded plant cells that activate T-DNA transfer in Agrobacterium tumefaciens. Nature. 318 (6047), 624-629 (1985).
- Memelink, J., de Pater, B. S., Hoge, J. H. C., Schilperoort, R. A. T-DNA hormone biosynthetic genes: Phytohormones and gene expression in plants. Dev. Genet. 8 (5-6), 321-337 (1987).
- Yuan, Z. -. C., et al. The plant signal salicylic acid shuts down expression of the vir regulon and activates quormone-quenching genes in Agrobacterium. Proc. Natl. Acad. Sci. USA. 104 (28), 11790-11795 (2007).
- Yuan, Z. -. C., Haudecoeur, E., Faure, D., Kerr, K. F., Nester, E. W. Comparative transcriptome analysis of Agrobacterium tumefaciens in response to plant signal salicylic acid, indole-3-acetic acid and γ-amino butyric acid reveals signalling cross-talk and Agrobacterium-plant co-evolution. Cell. Microbiol. 10 (11), 2339-2354 (2008).
- Li, P. L., Farrand, S. K. The replicator of the nopaline-type Ti plasmid pTiC58 is a member of the repABC family and is influenced by the TraR-dependent quorum-sensing regulatory system. J. Bacteriol. 182 (1), 179-188 (2000).
- Atkinson, M. M., Huang, J., Knopp, J. A. Hypersensitivity of suspension-cultured tobacco cells to pathogenic bacteria. Phytopathology. 75 (11), 1270-1274 (1985).
- Veena, ., Jiang, H., Doerge, R. W., Gelvin, S. B. Transfer of T-DNA and Vir proteins to plant cells by Agrobacterium tumefaciens induces expression of host genes involved in mediating transformation and suppresses host defense gene expression. Plant J. 35 (2), 219-236 (2003).
- León, J., Rojo, E., Sanchez-Serrano, J. J. Wound signalling in plants. J. Exp. Bot. 52 (354), 1-9 (2001).
- Ditt, R. F., Kerr, K. F., de Figueiredo, P., Delrow, J., Comai, L., Nester, E. W. The Arabidopsis thaliana transcriptome in response to Agrobacterium tumefaciens. Mol. Plant Microbe In. 19 (6), 665-681 (2006).
- Mandimba, G., Heulin, T., Bally, R., Guckert, A., Balandreau, J. Chemotaxis of free-living nitrogen-fixing bacteria towards maize mucilage. Plant Soil. 90 (1-3), 129-139 (1986).
- Rudrappa, T., Czymmek, K. J., Pare, P. W., Bais, H. P. Root-secreted malic acid recruits beneficial soil bacteria. Plant Physiol. 148 (3), 1547-1556 (2008).
- Lee, C. W., et al. Agrobacterium tumefaciens promotes tumor induction by modulating pathogen defense in Arabidopsis thaliana. Plant Cell. 21 (9), 2948-2962 (2009).
- Reymond, P., Weber, H., Damond, M., Farmer, E. E. Differential gene expression in response to mechanical wounding and insect feeding in Arabidopsis. Plant Cell. 12 (5), 707-720 (2000).
- Matthysse, A. G. Attachment of Agrobacterium to plant surfaces. Front. Plant Sci. 5, 252 (2014).
- Alatorre-Cobos, F., et al. An improved, low-cost, hydroponic system for growing Arabidopsis and other plant species under aseptic conditions. BMC Plant Biol. 14 (1), 69 (2014).
- Conn, S. J., et al. Protocol: Optimising hydroponic growth systems for nutritional and physiological analysis of Arabidopsis thaliana and other plants. Plant Methods. 9 (1), 4 (2013).
- Nathoo, N. . Identification of putative plant defense genes using a novel hydroponic co-cultivation technique for studying plant-pathogen interaction. , (2015).
- Shaner, N. C., Campbell, R. E., Steinbach, P. A., Giepmans, B. N., Palmer, A. E., Tsien, R. Y. Improved monomeric red, orange and yellow fluorescent proteins derived from Discosoma sp. red fluorescent protein. Nat. Biotechnol. 22 (12), 1567-1572 (2004).
- Wise, A. A., Liu, H., Binns, A. N. Nucleic acid extraction from Agrobacterium strains. Methods Mol. Bio. 343, 67-76 (2006).
- Nolan, T., Hands, R. E., Bustin, S. A. Quantification of mRNA using real-time RT-PCR. Nat. Protoc. 1 (3), 1559-1582 (2006).
- Salter, M. G., Conlon, H. E. Extraction of plant RNA. Methods Mol. Bio. 362, 309-314 (2007).
- Bao, Y., Wang, S., Yang, X., Li, T., Xia, Y., Meng, X. Metabolomic study of the intervention effects of Shuihonghuazi Formula, a Traditional Chinese Medicinal formulae, on hepatocellular carcinoma (HCC) rats using performance HPLC/ESI-TOF-MS. J. Ethnopharmacol. 198, 468-478 (2017).
- Korves, T. M., Bergelson, J. A developmental response to pathogen infection in Arabidopsis. Plant Physiol. 133 (1), 339-347 (2003).
- Lyons, R., Rusu, A., Stiller, J., Powell, J., Manners, J. M., Kazan, K. Investigating the association between flowering time and defense in the Arabidopsis thaliana-Fusarium oxysporum interaction. PLoS ONE. 10 (6), e0127699 (2015).
- Badri, D. V., Weir, T. L., vander Lelie, D., Vivanco, J. M. Rhizosphere chemical dialogues: Plant-microbe interactions. Curr. Opin. Biotechnol. 20 (6), 642-650 (2009).
- Baerson, S. R., et al. Detoxification and transcriptome response in Arabidopsis seedlings exposed to the allelochemical benzoxazolin-2(3H)-one. J. Biol. Chem. 280 (23), 21867-21881 (2005).

