Ein Hydroponisches Co-Kultivierungssystem zur gleichzeitigen und systematischen Analyse von Pflanzen- / Mikroben-Molecular-Wechselwirkungen und Signalgebung
Summary
Das beschriebene hydroponische Kokultivierungssystem unterstützt intakte Pflanzen mit Metallgittersieb und kokultiviert sie mit Bakterien. Pflanzengewebe, Bakterien und sezernierte Moleküle können dann separat für nachgeschaltete Analysen geerntet werden, wobei gleichzeitig die molekularen Reaktionen sowohl der Pflanzenwirte als auch der interagierenden Mikroben oder Mikrobiome untersucht werden können.
Abstract
Ein experimentelles Design, das natürliche Pflanzen-Mikroben-Wechselwirkungen nachahmt, ist sehr wichtig, um die komplexen Plant-Microbe-Signalisierungsprozesse abzugrenzen. Arabidopsis thaliana – Agrobacterium tumefaciens Bietet ein hervorragendes Modellsystem zur Untersuchung bakterieller Pathogenese und Pflanzeninteraktionen. Frühere Untersuchungen von Pflanzen -Agrobacterium- Wechselwirkungen haben sich weitgehend auf Pflanzenzell-Suspensionskulturen, die künstliche Verwundung von Pflanzen oder die künstliche Induktion von mikrobiellen Virulenzfaktoren oder Pflanzenverteidigungen durch synthetische Chemikalien angewiesen. Allerdings unterscheiden sich diese Methoden von der natürlichen Signalisierung in Planta , wo Pflanzen und Mikroben in räumlichen und zeitlichen Manieren erkennen und reagieren. Diese Arbeit präsentiert ein hydroponisches Kokulturierungssystem, bei dem intakte Pflanzen von Metallgittersiebe gestützt und mit Agrobacterium kultiviert werden. In diesem Kokultivierungssystem wird kein synthetisches Phytohormon oder eine Chemikalie, die micr induziertObjektive Virulenz oder Pflanzenverteidigung wird ergänzt. Das hydroponische Kultivierungssystem ähnelt sehr starken Pflanzen-Mikroben-Wechselwirkungen und signalisiert die Homöostase in Planta . Pflanzenwurzeln können aus dem Medium, das Agrobacterium enthält , getrennt werden, und die Signalisierung und Reaktionen sowohl der Pflanzenwirte als auch der wechselwirkenden Mikroben können gleichzeitig und systematisch untersucht werden. Zu jedem gegebenen Zeitpunkt / Intervall können Pflanzengewebe oder Bakterien separat für verschiedene "Omics" -Analysen geerntet werden, was die Leistungsfähigkeit und Wirksamkeit dieses Systems zeigt. Das Hydroponik-Kokulturierungssystem lässt sich leicht an das Studium anpassen: 1) die wechselseitige Signalisierung verschiedener Pflanzen-Mikroben-Systeme, 2) Signalisierung zwischen einem Pflanzenwirt und mehreren mikrobiellen Spezies ( dh mikrobielle Konsortien oder Mikrobiomen), 3) wie Nährstoffe und Chemikalien impliziert werden In der Pflanzenmikroben-Signalisierung und 4) wie die Mikroben mit Pflanzenwirten interagieren und zur Pflanzenverträglichkeit zur Biotik beitragenR abiotische Belastungen.
Introduction
Pflanzen-assoziierte Mikroben spielen eine wichtige Rolle im biogeochemischen Radfahren, Bioremediation, Klimaschutz, Pflanzenwachstum und Gesundheit sowie Pflanzenverträglichkeit gegenüber biotischen und abiotischen Belastungen. Mikroorganismen interagieren mit Pflanzen sowohl direkt durch den Pflanzenzellwandkontakt als auch indirekt über chemische Sekretion und Signalisierung 1 , 2 , 3 . Als sessile Organismen haben Pflanzen direkte und indirekte Mechanismen entwickelt, um einer Infektion durch Pathogene zu widerstehen. Direkte Abwehr umfasst strukturelle Abwehrkräfte und die Expression von Verteidigungsproteinen, während indirekte Verteidigungen sekundäre Pflanzenmetabolitproduktion und die Anziehung von Organismen antagonistisch gegenüber eindringenden Pathogenen 4 , 5 umfassen . Pflanzen-abgeleitete Wurzel-Exsudate, Sekrete, Schleime, Mucigel und Lysate verändern die physikalisch-chemischen Eigenschaften der Rhizosphäre zu gewinnen oder zu stoßenMikroben gegen ihre Gastgeber 6 . Die chemische Zusammensetzung der Wurzelsekretion ist speziesspezifisch und dient so als selektiver Filter, der es ermöglicht, dass bestimmte Mikroorganismen, die in der Lage sind, solche Verbindungen zu erkennen, in der Rhizosphäre 6 zu blühen. So können kompatible mikrobielle Spezies stimuliert werden, um ihre Assoziationen zu aktivieren und zu verstärken, entweder zum Nutzen oder zum Nachteil des Pflanzenwirts 1 .
Das Verständnis von Pflanzen-Mikroben-Wechselwirkungen in der Rhizosphäre ist der Schlüssel zur Steigerung der Pflanzenproduktivität und des Ökosystems, da eine Mehrheit der mikrobiellen und chemischen Exposition an der Wurzelstruktur und der Boden-Luft-Grenzfläche 2 , 6 , 7 , 8 erfolgt . Allerdings ist die Untersuchung von unterirdischen Pflanzen-Mikroben-Wechselwirkungen und wechselseitigen Reaktionen aufgrund ihrer faszinierenden Herausforderung eine Herausforderung Komplexe und dynamische Natur und der Mangel an geeigneten experimentellen Modellen mit natürlicher Wurzelstruktur und Pflanzenmorphologie unter streng kontrollierbaren Wachstumsbedingungen. Als einer der am stärksten untersuchten Phytopathogene infiziert Agrobacterium eine breite Palette von Pflanzen mit landwirtschaftlicher und gartenbaulicher Bedeutung, darunter Kirsche, Apfel, Birne, Traube und Rose 9 . Agrobacterium ist ein wichtiger Modellorganismus für das Verständnis von Pflanzen-Pathogen-Wechselwirkungen und ist ein leistungsfähiges Werkzeug in der Pflanzen-Transformation und Anlagentechnik 10 , 11 , 12 , 13 , 14 .
Molekulare Pflanzen- Agrobacterium- Wechselwirkungen wurden seit mehreren Jahrzehnten gut untersucht, und das aktuelle Verständnis der Agrobacterium- Pathogenität ist umfangreich 9 ,f "> 11, 15, 16. Agrobacterium Pathogenität in seine entwickelte Fähigkeiten des Wahrnehmens von Pflanzen abgeleiteten Signale weitgehend zurückgeführt, in der feine Modulation seiner Virulenz Programm und Zell-zu-Zell – Kommunikation führt, sogenannte quorum sensing 17. Der Das Agrobacterium- Virulenz-Programm wird durch mehrere in der Rhizosphäre verfügbare Signale reguliert und umfasst zwei Sätze von 2-Komponenten-Systemen, das ChvG / I-System und das VirA / G-System. Säurebedingungen in der Rhizosphäre aktivieren die Transkription von chvG / I , virA / G , Und mehrere andere Gene, die an der Agrobacterium- Pathogenität beteiligt sind, einschließlich virE0 , virE1 , virH1 , virH2 und Genen des Typ-VI-Sekretionssystems (T6SS) 18. Pflanzliche Phenolverbindungen, einschließlich Acetosyringon (4'-hydroxy-3 ', 5 '-Dimethoxyacetophenon), aktivieren Sie die V.IrA / G 2-Komponenten-System durch Phosphorylierungs-Signalisierungsmechanismen 19 . VirA / G aktiviert dann das gesamte vir- regulon, was zur Übertragung und Integration eines ~ 20 kb bakteriellen DNA-Fragments namens Transfer-DNA (T-DNA) aus seinem tumorinduzierenden (Ti) -Plasmid in den Pflanzenkern 16 führt . T-DNA trägt Gene, die für die Synthese der Pflanzenhormone Indol-3-essigsäure (IAA) ( iaaM und iaaH ) und Cytokinin ( ipt ) verantwortlich sind und einmal in Pflanzenzellen exprimiert werden, werden große Mengen dieser Phytohormone produziert. Dies führt zu einer abnormen Gewebevermehrung und Pflanzenentumorentwicklung, bekannt als Krone Gallenkrankheit, die ein chronisches und wiederauflebendes Problem für Pflanzen 9 , 11 , 20 ist . IAA wirkt auch gemeinsam mit Salicylsäure und Gamma-Aminobuttersäure, um Agrobacterium- Virulenz zu unterdrücken oder Agrobacteriu zu reduzieren M Quorum Sensing (QS) 17 , 21 , 22 . Um dieser Repression entgegenzuwirken, trägt T-DNA auch Gene für die Opin-Biosynthese, die Agrobacterium quorum-Sensing aktiviert, um die Agrobacterium- Pathogenität zu fördern und dient auch als Nährstoffquelle für den Pathogen 22 , 23 .
Trotz eines insgesamt tiefen Verständnisses von Agrobacterium- Pflanzen-Wechselwirkungen und der resultierenden T-DNA-Übertragung in den Pflanzenwirt sind die komplexen Signalisierungsereignisse im Anfangsstadium der Interaktion weniger gut verstanden. Dies ist teilweise auf die Einschränkungen konventioneller Ansätze zur Untersuchung von Agrobacterium- Plant-Signalisierung zurückzuführen. Pflanzenzell-Suspensionskulturen und künstliche ortsspezifische Verwundungen werden häufig verwendet, um molekulare Pflanzen-Mikroben-Wechselwirkungen zu untersuchen 24 ,Ef "> 26 , 27. Allerdings fehlen Zellsuspensionen einer typischen Pflanzenmorphologie, insbesondere haben Pflanzen-Suspensionszellen keine Wurzelstrukturen und Wurzel-Exsudate, die für die Aktivierung der mikrobiellen Chemotaxis und der Virulenz 28 , 29 sehr wichtig sind. Die Aufrechterhaltung der Pflanzenmorphologie Und die Wurzelstruktur wurde durch künstlich verwundete Pflanzen adressiert, was eine ortsspezifische Infektion erleichtert, was zum Nachweis von induzierten pflanzenverteidigungsbezogenen Genen in direkt infiziertem Pflanzengewebe 30 , 31 führt . Die künstliche Verwundung unterscheidet sich jedoch deutlich von der pathogenen Infektion in der Natur , Insbesondere wenn die Verwundung zu einer Jasmoninsäure (JA) -Akkumulation führt, die systemisch mit der natürlichen Anlagensignalisierung und -verteidigung in Einklang steht 26. Darüber hinaus werden synthetische Chemikalien typischerweise verwendet, um Pflanzenwirtsreaktionen künstlich zu induzierenOder pathogene Virulenz. Obwohl die Ergänzung solcher chemischen Verbindungen, die die Konzentrationen in Planta reflektieren, möglich ist, ist eine solche Ergänzung nicht die Diffusion von Wurzelausscheidungen allmählich in die umgebende Rhizosphäre, die einen von den Mikroben 28 , 32 erfaßten chemotaktischen Gradienten erzeugt. Angesichts der Einschränkungen herkömmlicher Ansätze zur Untersuchung von Pflanzen-Mikroben-Wechselwirkungen können die Genauigkeit und Tiefe der erhaltenen Daten behindert und restriktiv sein und das aus den konventionellen Ansätzen erzeugte Wissen kann nicht direkt in Planta umgesetzt werden . Viele Aspekte der Pflanzen- Agrobacterium- Signalisierung sind noch nicht vollständig verstanden, besonders in der frühen Phase der Wechselwirkungen, wenn die Krankheitssymptome noch nicht entwickelt haben.
Um die Grenzen herkömmlicher Ansätze zu ändern, stellt diese Arbeit eine kostengünstige, streng kontrollierbare und flexible Hydroponik dar. COkultivierungssystem, das es Forschern ermöglicht, tiefer Einblicke in die komplexen Signalisierungs- und Reaktionswege im Anfangsstadium molekularer Pflanzen-Mikroben-Wechselwirkungen zu gewinnen. Hydroponik wurde weithin verwendet, um Pflanzennährstoffe, Wurzel-Exsudate, Wachstumsbedingungen und die Auswirkungen der metallischen Toxizität auf Pflanzen 33 , 34 zu studieren. Es gibt mehrere Vorteile von hydroponischen Modellen, darunter die kleinen räumlichen Anforderungen, die Zugänglichkeit verschiedener Pflanzengewebe, die enge Kontrolle der Nährstoff- / Umweltbedingungen und die Schädlingsbekämpfung. Hydroponische Systeme sind auch weniger begrenzt auf Pflanzenwachstum im Vergleich zu Agar / Phytoagar-Beschichtung Techniken, die in der Regel beschränken Wachstum nach 2-3 Wochen. Wichtig ist, dass die Aufrechterhaltung von Ganzpflanzenstrukturen die für die mikrobielle Chemotaxis und die Virulenzinduktion notwendige natürliche Wurzelsekretion erleichtert 8 , 29 . Das System beschreibtBett hier ist einfacher und weniger arbeitsintensiv als die Alternativen 33 , 34 . Es nutzt weniger Teile und benötigt keine anderen Werkzeuge als Standardscheren. Es verwendet Metallgewebe (im Gegensatz zu Nylon 33 ) als eine starke Unterstützung für Pflanzenwachstum und eine einfache Methode der Belüftung unter sterilen Bedingungen durch Schütteln, um mikrobielles Wachstum zu unterstützen. Darüber hinaus kann das System Metallgewebe in verschiedenen Größen verwenden, um das Pflanzenwachstum zu unterstützen, das verschiedene Pflanzenarten aufnimmt, ohne die Breite ihrer Wurzeln zu beschränken.
In dem hier vorgestellten Hydroponik-Kokultur-System werden Pflanzen in einem sterilen Hydroponik-System angebaut, wo die Pflanzenwurzeln organische Verbindungen absondern, die das Wachstum von geimpften Bakterien unterstützen. In diesem Kokultivierungssystem werden keine künstlichen Chemikalien wie Pflanzenhormone, Verteidigungsauslöser oder virulenzinduzierende Chemikalien ergänzt, was die natürliche Zelle widerspiegelt-signale Homöostase bei Pflanzen-Mikroben-Wechselwirkungen. Mit diesem hydroponischen Kokultivierungssystem war es möglich, die Genexpression in Arabidopsis thaliana Col-0 Wurzelgewebe bei der Infektion durch Agrobacterium sowie die Aktivierung von Agrobacterium- Genen bei der Kokultivierung mit Arabidopsis gleichzeitig zu bestimmen. Es wurde weiter gezeigt, dass dieses System geeignet ist, Agrobacterium- Anlagerung an Pflanzenwurzeln sowie das Pflanzenwurzel-Sekretariat-Profil, bei der Kokultivierung (Infektion) mit Agrobacterium ( Abbildung 1 ) zu untersuchen.
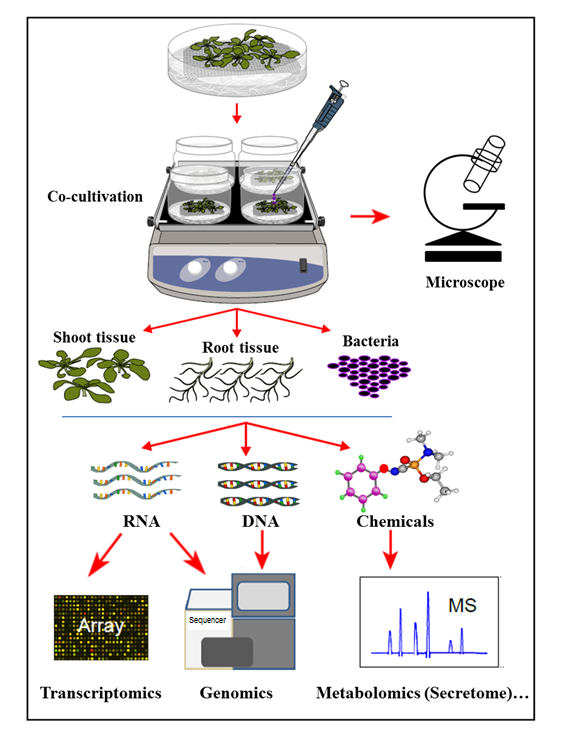
Abbildung 1: Überblick über das Hydroponic Cocultivation System mit Probenanalysen. Pflanzen werden oben auf dem Netz gezüchtet (schießt über dem Netz), wobei die Wurzeln in das hydroponische Medium eintauchen, das dann mit Bakterien geimpft wirdOder Kokultur. Pflanzengewebe und Bakterien werden dann für gleichzeitige Extraktionen und Analysen getrennt. Diese Zahl wurde aus Referenz 35 geändert.
Protocol
Representative Results
Discussion
Angesichts der allmählichen Beschaffenheit der Wurzelsekretion erfolgt die Konzentration der in Planta produzierten Virulenz-induzierenden Chemikalien und deren Auswirkungen auf dynamische Pflanzen-Mikroben-Wechselwirkungen in räumlichen und zeitlichen Gradienten. In diesem hydroponischen Co-Kultivierungssystem wird kein synthetisches Phytohormon oder eine Chemikalie, die mikrobielle Virulenz oder Pflanzenverteidigung induziert, ergänzt. Im Gegensatz dazu, mit konventionellen Ansätzen, die Zugabe von synthe…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Wir danken Brian Weselowski und Alexander W. Eastman für ihre Hilfe und nützliche Diskussion. Wir danken auch Drs. Eugene W. Nester, Lingrui Zhang, Haitao Shen, Yuhai Cui und Greg Thorn für ihre Hilfe, nützliche Diskussionen und kritische Lektüre des Manuskripts. Diese Forschung wurde gefördert durch Landwirtschaft und Agri-Food Kanada, wachsende Forward-AgriFlex (RBPI Nummer 2555) und Growing Forward II Projekt Nummer 1670, durchgeführt von den Autoren als Teil ihrer Aufgaben. Diese Studie wurde auch teilweise von der Naturwissenschaften und Ingenieurwissenschaften Council of Canada (NSERC) Discovery Grant RGPIN-2015-06052 an ZC Yuan vergeben finanziert.
Materials
| plant seeds (Arabidopsis thaliana Col-0) | Arabidopsis Biological Resource Centre | CS7000 | https://abrc.osu.edu/order-stocks |
| bacteria (Agrobacterium tumefaciens C58) | University of Washington | N/A | |
| labeled bacteria | in-house | optional, depends on downstream analyses | |
| vortex | (various) | ||
| microcentrifuge tubes | (various) | ||
| microcentrifuge | (various) | ||
| 5% sodium hypochlorite | (various) | ||
| double distilled water | (various) | ||
| autoclave | (various) | ||
| micropipette | (various) | ||
| 70 % ethanol | (various) | ||
| Murashige and Skoog (MS) basal salts | Sigma-Aldrich | M5524 | |
| sucrose | (various) | ||
| MES | (various) | ||
| B5 vitamin mix | Sigma-Aldrich | G1019 | |
| phytoagar | (various) | ||
| deep Petri dishes | (various) | ||
| stainless steel mesh | Ferrier Wire Goods Company Ltd | N/A | grade: 304; mesh count: 40 × 40; wire DIA: 0.01 |
| micropore tape, 1 inch | 3M | 1530-1 | |
| diurnal growth chamber | (various) | ||
| cylindrical glass tanks, 100 × 80 mm | Pyrex | 3250 | other sizes can be used, in which case liquid content may need adjustment |
| flow hood | (various) | ||
| forcepts | (various) | ||
| yeast extract | (various) | ||
| tryptone | (various) | ||
| MgSO4 | (various) | ||
| shaking incubator | (various) | ||
| spectrophotometer | (various) | ||
| NaCl | (various) | ||
| shaker | (various) | ||
| scissors | (various) | optional, depends on downstream analyses | |
| fluorescence microscope | (various) | optional, depends on downstream analyses | |
| microscope slides and cover slips | (various) | optional, depends on downstream analyses | |
| nail polish | (various) | optional, depends on downstream analyses | |
| Bacterial RNA extraction kit | (various) | optional, depends on downstream analyses | |
| plant RNA extraction kit (RNeasy Plant Mini Kit) | Qiagen | 74903 or 74904 | optional, depends on downstream analyses |
| material and equipment for RT-qPCR | (various) | optional, depends on downstream analyses | |
| material and equipment for microarray analysis | (various) | optional, depends on downstream analyses | |
| liquid nitrogen | (various) | optional, depends on downstream analyses | |
| mortar and pestle | (various) | optional, depends on downstream analyses | |
| 0.2 µm pore filter | (various) | optional, depends on downstream analyses | |
| 50 mL conical tubes | (various) | optional, depends on downstream analyses | |
| freeze dryer | (various) | optional, depends on downstream analyses | |
| sealable test tubes | (various) | optional, depends on downstream analyses | |
| ethyl acetate | (various) | optional, depends on downstream analyses | |
| nitrogen gas | (various) | optional, depends on downstream analyses | |
| material and equipment for HPLC | (various) | optional, depends on downstream analyses | |
| material and equipment for ESI-TOF-MS | (various) | optional, depends on downstream analyses |
References
- Lambers, H., Mougel, C., Jaillard, B., Hinsinger, P. Plant-microbe-soil interactions in the rhizosphere: An evolutionary perspective. Plant Soil. 321 (1), 83-115 (2009).
- Philippot, L., Raaijmakers, J. M., Lemanceau, P., vander Putten, W. H. Going back to the roots: The microbial ecology of the rhizosphere. Nat. Rev. Microbiol. 11 (11), 789-799 (2013).
- Somers, E., Vanderleyden, J., Srinivasan, M. Rhizosphere bacterial signalling: A Love Parade beneath our feet. Crit. Rev. Microbiol. 30 (4), 205-240 (2004).
- Barah, P., Winge, P., Kusnierczyk, A., Tran, D. H., Bones, A. M. Molecular signatures in Arabidopsis thaliana in response to insect attack and bacterial infection. PLoS ONE. 8 (3), (2013).
- Zhang, J., Zhou, J. -. M. Plant immunity triggered by microbial molecular signatures. Mol. Plant. 3 (5), 783-793 (2010).
- Paterson, E., Gebbing, T., Abel, C., Sim, A., Telfer, G. Rhizodeposition shapes rhizosphere microbial community structure in organic soil. New Phytol. 173 (3), 600-610 (2006).
- Hartmann, A., Schmid, M., van Tuinen, D., Berg, G. Plant-driven selection of microbes. Plant Soil. 321 (1-2), 235-257 (2008).
- Micallef, S. A., Shiaris, M. P., Colon-Carmona, A. Influence of Arabidopsis thaliana accessions on rhizobacterial communities and natural variation in root exudates. J. Exp. Bot. 60 (6), 1729-1742 (2009).
- Burr, T., Otten, L. Crown gall of grape: Biology and disease management. Annu. Rev. Phytopathol. 37, 53-80 (2001).
- Clough, S. J., Bent, A. F. Floral dip: A simplified method for Agrobacterium-mediated transformation of Arabidopsis thaliana. Plant J. 16 (6), 735-743 (1998).
- Binns, A. N., Costantino, P., Spaink, H. P., Kondorosi, A., Hooykaas, P. J. J. The Agrobacterium oncogenes. The Rhizobiaceae. , (1998).
- Gheysen, G., Angenon, G., Van Montagu, M., Lindsey, K. Agrobacterium-mediated plant transformation: a scientifically intriguing story with significant applications. Transgenic Plant Research. , (1998).
- Valvekens, D., Von Montagu, M. V., Van Lijsebettens, M. Agrobacterium tumefaciens-mediated transformation of Arabidopsis thaliana root explants by using kanamycin selection. Proc. Natl. Acad. Sci. USA. 85 (15), 5536-5540 (1988).
- Krysan, P. J., Young, J. C., Sussman, M. R. T-DNA as an insertional mutagen in Arabidopsis. Plant Cell. 11 (12), 2283 (1999).
- Chilton, M. D., et al. Stable incorporation of plasmid DNA into higher plant cells: The molecular basis of crown gall tumorigenesis. Cell. 11 (2), 263-271 (1977).
- Gelvin, S. B. Agrobacterium in the genomics age. Plant Physiol. 150 (4), 1665-1676 (2009).
- Subramoni, S., Nathoo, N., Klimov, E., Yuan, Z. -. C. Agrobacterium tumefaciens responses to plant-derived signaling molecules. Front. Plant Sci. 5, 322 (2014).
- Yuan, Z., Liu, P., Saenkham, P., Kerr, K., Nester, E. W. Transcriptome profiling and functional analysis of Agrobacterium tumefaciens reveals a general conserved response to acidic conditions (pH 5.5) and a complex acid-mediated signaling involved in Agrobacterium-plant interactions. J. Bacteriol. 190 (2), 494-507 (2008).
- Stachel, S. E., Messens, E., Van Montagu, M., Zambryski, P. Identification of the signal molecules produced by wounded plant cells that activate T-DNA transfer in Agrobacterium tumefaciens. Nature. 318 (6047), 624-629 (1985).
- Memelink, J., de Pater, B. S., Hoge, J. H. C., Schilperoort, R. A. T-DNA hormone biosynthetic genes: Phytohormones and gene expression in plants. Dev. Genet. 8 (5-6), 321-337 (1987).
- Yuan, Z. -. C., et al. The plant signal salicylic acid shuts down expression of the vir regulon and activates quormone-quenching genes in Agrobacterium. Proc. Natl. Acad. Sci. USA. 104 (28), 11790-11795 (2007).
- Yuan, Z. -. C., Haudecoeur, E., Faure, D., Kerr, K. F., Nester, E. W. Comparative transcriptome analysis of Agrobacterium tumefaciens in response to plant signal salicylic acid, indole-3-acetic acid and γ-amino butyric acid reveals signalling cross-talk and Agrobacterium-plant co-evolution. Cell. Microbiol. 10 (11), 2339-2354 (2008).
- Li, P. L., Farrand, S. K. The replicator of the nopaline-type Ti plasmid pTiC58 is a member of the repABC family and is influenced by the TraR-dependent quorum-sensing regulatory system. J. Bacteriol. 182 (1), 179-188 (2000).
- Atkinson, M. M., Huang, J., Knopp, J. A. Hypersensitivity of suspension-cultured tobacco cells to pathogenic bacteria. Phytopathology. 75 (11), 1270-1274 (1985).
- Veena, ., Jiang, H., Doerge, R. W., Gelvin, S. B. Transfer of T-DNA and Vir proteins to plant cells by Agrobacterium tumefaciens induces expression of host genes involved in mediating transformation and suppresses host defense gene expression. Plant J. 35 (2), 219-236 (2003).
- León, J., Rojo, E., Sanchez-Serrano, J. J. Wound signalling in plants. J. Exp. Bot. 52 (354), 1-9 (2001).
- Ditt, R. F., Kerr, K. F., de Figueiredo, P., Delrow, J., Comai, L., Nester, E. W. The Arabidopsis thaliana transcriptome in response to Agrobacterium tumefaciens. Mol. Plant Microbe In. 19 (6), 665-681 (2006).
- Mandimba, G., Heulin, T., Bally, R., Guckert, A., Balandreau, J. Chemotaxis of free-living nitrogen-fixing bacteria towards maize mucilage. Plant Soil. 90 (1-3), 129-139 (1986).
- Rudrappa, T., Czymmek, K. J., Pare, P. W., Bais, H. P. Root-secreted malic acid recruits beneficial soil bacteria. Plant Physiol. 148 (3), 1547-1556 (2008).
- Lee, C. W., et al. Agrobacterium tumefaciens promotes tumor induction by modulating pathogen defense in Arabidopsis thaliana. Plant Cell. 21 (9), 2948-2962 (2009).
- Reymond, P., Weber, H., Damond, M., Farmer, E. E. Differential gene expression in response to mechanical wounding and insect feeding in Arabidopsis. Plant Cell. 12 (5), 707-720 (2000).
- Matthysse, A. G. Attachment of Agrobacterium to plant surfaces. Front. Plant Sci. 5, 252 (2014).
- Alatorre-Cobos, F., et al. An improved, low-cost, hydroponic system for growing Arabidopsis and other plant species under aseptic conditions. BMC Plant Biol. 14 (1), 69 (2014).
- Conn, S. J., et al. Protocol: Optimising hydroponic growth systems for nutritional and physiological analysis of Arabidopsis thaliana and other plants. Plant Methods. 9 (1), 4 (2013).
- Nathoo, N. . Identification of putative plant defense genes using a novel hydroponic co-cultivation technique for studying plant-pathogen interaction. , (2015).
- Shaner, N. C., Campbell, R. E., Steinbach, P. A., Giepmans, B. N., Palmer, A. E., Tsien, R. Y. Improved monomeric red, orange and yellow fluorescent proteins derived from Discosoma sp. red fluorescent protein. Nat. Biotechnol. 22 (12), 1567-1572 (2004).
- Wise, A. A., Liu, H., Binns, A. N. Nucleic acid extraction from Agrobacterium strains. Methods Mol. Bio. 343, 67-76 (2006).
- Nolan, T., Hands, R. E., Bustin, S. A. Quantification of mRNA using real-time RT-PCR. Nat. Protoc. 1 (3), 1559-1582 (2006).
- Salter, M. G., Conlon, H. E. Extraction of plant RNA. Methods Mol. Bio. 362, 309-314 (2007).
- Bao, Y., Wang, S., Yang, X., Li, T., Xia, Y., Meng, X. Metabolomic study of the intervention effects of Shuihonghuazi Formula, a Traditional Chinese Medicinal formulae, on hepatocellular carcinoma (HCC) rats using performance HPLC/ESI-TOF-MS. J. Ethnopharmacol. 198, 468-478 (2017).
- Korves, T. M., Bergelson, J. A developmental response to pathogen infection in Arabidopsis. Plant Physiol. 133 (1), 339-347 (2003).
- Lyons, R., Rusu, A., Stiller, J., Powell, J., Manners, J. M., Kazan, K. Investigating the association between flowering time and defense in the Arabidopsis thaliana-Fusarium oxysporum interaction. PLoS ONE. 10 (6), e0127699 (2015).
- Badri, D. V., Weir, T. L., vander Lelie, D., Vivanco, J. M. Rhizosphere chemical dialogues: Plant-microbe interactions. Curr. Opin. Biotechnol. 20 (6), 642-650 (2009).
- Baerson, S. R., et al. Detoxification and transcriptome response in Arabidopsis seedlings exposed to the allelochemical benzoxazolin-2(3H)-one. J. Biol. Chem. 280 (23), 21867-21881 (2005).

