Et hydroponisk co-dyrkningssystem til simultan og systematisk analyse af plante / mikrobe molekylære interaktioner og signalering
Summary
Det beskrevne hydroponiske kultiveringssystem understøtter intakte planter med metal mesh skærme og cocultivates dem med bakterier. Plantevæv, bakterier og udskillede molekyler kan derefter høstes separat for nedstrømsanalyser, samtidig med at molekylære reaktioner fra både planteværter og interaktive mikrober eller mikrobiomer kan undersøges.
Abstract
Et eksperimentelt design der efterligner naturlige plante-mikrobe interaktioner er meget vigtigt at afgrænse de komplekse plante-mikrobe signalering processer. Arabidopsis thaliana – Agrobacterium tumefaciens Giver et fremragende model system til at studere bakteriel patogenese og planteinteraktioner. Tidligere undersøgelser af plante- Agrobacterium- interaktioner har i vid udstrækning været afhængige af plantecelle suspensionskulturer, kunstig såring af planter eller kunstig induktion af mikrobielle virulensfaktorer eller plantebeskyttelse af syntetiske kemikalier. Disse metoder adskiller sig imidlertid fra den naturlige signalering i planta , hvor planter og mikrober genkender og reagerer på rumlig og tidsmæssig måde. Dette arbejde præsenterer et hydroponisk kultiveringssystem, hvor intakte planter understøttes af metal mesh skærme og kultiveres med Agrobacterium . I dette kultiveringssystem findes ingen syntetisk phytohormon eller kemikalie, der inducerer micrObial virulence eller planteforsvar er suppleret. Det hydroponiske kultiveringssystem ligner næsten naturlige plante-mikrobe interaktioner og signalering homeostase i planta . Planterødder kan adskilles fra mediet indeholdende Agrobacterium , og signalering og respons fra både værtsværterne og de interaktive mikrober kan undersøges samtidigt og systematisk. Ved et hvilket som helst tidspunkt / interval kan plantevæv eller bakterier høstes separat for forskellige "omics" -analyser, der demonstrerer effekten og effekten af dette system. Det hydroponiske kultiveringssystem kan let tilpasses til at studere: 1) Gensidig signalering af forskellige plante-mikrobesystemer, 2) Signalering mellem en plantevært og flere mikrobielle arter ( dvs. mikrobielle konsortier eller mikrobiomer), 3) hvordan næringsstoffer og kemikalier er impliceret I plante-mikrobe signalering, og 4) hvordan mikrober interagerer med plante værter og bidrage til plantetolerance til biotiske oR abiotiske stress.
Introduction
Planteassocierede mikrober spiller vigtige roller inden for biogeokemisk cykling, bioremediering, afbødning af klimaændringer, plantevækst og sundhed og plantetolerance overfor biotiske og abiotiske påvirkninger. Mikroorganismer interagerer med planter både direkte gennem plantecellevægskontakt og indirekte via kemisk sekretion og signalering 1 , 2 , 3 . Som sessile organismer har planter udviklet direkte og indirekte mekanismer til at modstå infektion med patogener. Direkte forsvar omfatter strukturelle forsvar og udtryk for forsvarsproteiner, mens indirekte forsvar omfatter sekundær plantemetabolitproduktion og tiltrækningen af organismer, der er antagonistiske for invaderende patogener 4 , 5 . Planteafledte rod-exudater, sekreter, slimhinder, mucigel og lysater ændrer rhizosfærens fysisk-kemiske egenskaber for at tiltrække eller afviseMikrober mod deres værter 6 . Den kemiske sammensætning af rodsekretion er artsspecifik og tjener derved som et selektivt filter, der tillader visse mikroorganismer, der er i stand til at genkende sådanne forbindelser for at blomstre i rhizosfæren 6 . Således kan kompatible mikrobielle arter stimuleres til at aktivere og forbedre deres foreninger, enten til fordel for eller skade for planteværten 1 .
Forståelse af plante-mikrobe interaktioner i rhizosfæren er nøglen til at forbedre planteproduktiviteten og økosystemets funktion, da størstedelen af den mikrobielle og kemiske eksponering forekommer ved rodstrukturen og jord-luftgrænsefladen 2 , 6 , 7 , 8 . Undersøgelsen af underjordiske plante-mikrobe-interaktioner og gensidige reaktioner har imidlertid været en udfordring på grund af dens fascinerende Kompleks og dynamisk natur og mangel på egnede eksperimentelle modeller med naturlig rodstruktur og plantemorfologi under tæt kontrollerbare vækstbetingelser. Som en af de mest undersøgte fytopathogener inficerer Agrobacterium en bred vifte af planter med landbrugs- og gartnerisk betydning, herunder kirsebær, æble, pære, drue og rose 9 . Agrobacterium er en vigtig modelorganisme til forståelse af plantepatogeninteraktioner og er et kraftfuldt værktøj i plantetransformation og planteteknik 10 , 11 , 12 , 13 , 14 .
Molekylære plante- Agrobacterium- interaktioner er blevet undersøgt godt i flere årtier, og den nuværende forståelse af Agrobacterium- patogenicitet er omfattende 9 ,F.> 11 , 15 , 16. Agrobacteriumpatogenicitet er i høj grad tilskrevet dets evoluerede evner til at opfatte planteafledte signaler, hvilket resulterer i den fine modulering af dets virulensprogram og celle til celle kommunikation, såkaldt quorum sensing 17 . Agrobacterium virulence-programmet reguleres af flere signaler til rådighed i rhizosfæren og involverer to sæt 2-komponentsystemer, ChvG / I-systemet og VirA / G-systemet. Syrebetingelser i rhizosfæren aktiverer transkriptionen af chvG / I , virA / G , Og flere andre gener involveret i Agrobacterium- patogenicitet, herunder virE0 , virE1 , virH1 , virH2 og gener af type VI-sekretionssystemet (T6SS) 18. Plantafledte phenolforbindelser, herunder acetosyringon (4'-hydroxy-3 ', 5 '-dimethoxyacetophenon), aktiver V'enIrA / G 2-komponentsystem gennem phosphoryleringssignaleringsmekanismer 19 . VirA / G aktiverer derefter hele vir- regulonet, hvilket resulterer i overførsel og integration af et ~ 20 kb bakterielt DNA-fragment kaldet transfer DNA (T-DNA) fra dets tumorinducerende (Ti) plasmid i plantekernen 16 . T-DNA bærer gener ansvarlig for syntesen af plantehormonerne indol-3-eddikesyre (IAA) ( iaaM og iaaH ) og cytokinin ( ipt ) og en gang udtrykt i planteceller produceres store mængder af disse phytohormoner. Dette resulterer i unormal vævsproliferation og udvikling af plantetumor, kendt som kronesygdom, hvilket er et kronisk og genopståeligt problem for planterne 9 , 11 , 20 . IAA virker også kollektivt med salicylsyre og gamma-aminobutyrsyre for at undertrykke Agrobacterium virulens eller for at reducere Agrobacteriu M quorum sensing (QS) 17 , 21 , 22 . For at imødegå denne undertrykkelse bærer T-DNA også gener for opinbiosyntese, som aktiverer Agrobacterium quorum-sensing for at fremme Agrobacterium- patogenicitet og tjener også som en næringsstofkilde for patogenet 22 , 23 .
På trods af en samlet dyb forståelse af Agrobacterium- planteinteraktioner og den resulterende T-DNA-overførsel til planteværten er de komplekse signaleringshændelser i den indledende fase af interaktionen mindre velkendte. Dette skyldes delvist begrænsningerne af konventionelle metoder til undersøgelse af Agrobacterium- plantesignalering. Plantecellesuspensionskulturer og kunstig stedsspecifik såring anvendes almindeligt til at studere molekylære plante-mikrobe interaktioner 24 ,Ef "> 26 , 27. Celleopslæmninger mangler imidlertid typisk plantemorfologi, især plantesuspensionsceller har ikke rotstrukturer og rodeksudater, som er meget vigtige for aktivering af mikrobiell kemotaxis og virulens 28 , 29. Vedligeholdelsen af plantemorfologi Og rodstruktur er blevet behandlet af kunstigt sårende planter, hvilket letter lokalitetsspecifik infektion, hvilket resulterer i påvisning af inducerede planteforsvarrelaterede gener i direkte inficeret plantevæv 30 , 31. Imidlertid er kunstig såring signifikant forskellig fra patogeninfektion i naturen , Især da sår fører til akkumulering af jasmoninsyre (JA), som systematisk interfererer med naturlig plantesignalering og forsvar 26. Desuden anvendes syntetiske kemikalier typisk til kunstigt at fremkalde planteværtsresponserEller patogen virulens. Skønt tilskuddet af sådanne kemiske forbindelser, der reflekterer koncentrationer i planta, er muligt, tager sådanne tilskud ikke hensyn til diffusionen af rod-exudater gradvist ind i den omgivende rhizosfære, hvilket frembringer en kemotaktisk gradient registreret af mikrober 28 , 32 . I betragtning af begrænsningerne af konventionelle fremgangsmåder til undersøgelse af plante-mikrobe-interaktioner kan nøjagtigheden og dybden af de opnåede data være hæmmet og restriktive, og den viden, der genereres ud fra de konventionelle fremgangsmåder, kan ikke oversættes direkte i planta . Mange aspekter af plante- Agrobacterium- signalering er endnu ikke fuldt ud forstået, især i det tidlige stadium af interaktioner, når sygdomssymptomerne endnu ikke er udviklet.
For at ændre begrænsningerne ved konventionelle tilgange præsenterer dette arbejde en billig, tæt kontrollerbar og fleksibel hydroponisk cOdlingssystem, der giver forskere mulighed for at få dybere indsigt i de komplekse signal- og reaktionsveje ved begyndelsen af molekylære plante-mikrobe interaktioner. Hydroponics har været meget anvendt til at studere plantenæringsstoffer, rod-ekssudater, vækstbetingelser og virkningerne af metallisk toksicitet på planterne 33 , 34 . Der er flere fordele ved hydroponiske modeller, herunder de små rumlige krav, tilgængeligheden af forskellige plantevæv, den tætte kontrol af næringsstof / miljøforhold og bekæmpelse af skadegørere / sygdomme. Hydroponiske systemer er også mindre begrænsende for plantevækst i sammenligning med agar / phytoagar plating teknikker, der typisk begrænser vækst efter 2-3 uger. Det er vigtigt, at vedligeholdelsen af helplantekonstruktioner letter den naturlige rodsekretion, der er nødvendig for mikrobiell kemotakse og virulensinduktion 8 , 29 . Systemet beskriverSeng her er enklere og mindre arbejdskrævende end alternativene 33 , 34 . Det bruger færre dele og kræver ikke andre værktøjer end standard saks. Den bruger metalnet (i modsætning til nylon 33 ) som en stærk støtte til plantevækst og en simpel metode til beluftning under sterile betingelser gennem omrystning for at understøtte mikrobiel vækst. Derudover kan systemet bruge metalnet af forskellige størrelser til at understøtte plantevækst, som rummer forskellige plantearter uden at begrænse bredden af deres rødder.
I det hydroponiske kultiveringssystem, der præsenteres her, dyrkes planter i et sterilt hydroponisk system, hvor plantens rødder udskiller organiske forbindelser, der understøtter væksten af podede bakterier. I dette kultiveringssystem suppleres ingen kunstige kemikalier, såsom plantehormoner, forsvarselektronik eller virulensfremkaldende kemikalier, hvilket afspejler den naturlige celleSignalerende homeostase under plante-mikrobe interaktioner. Med dette hydroponiske kultiveringssystem var det muligt at samtidigt bestemme genekspression i Arabidopsis thaliana Col-0 rodvæv efter infektion med Agrobacterium , såvel som aktiveringen af Agrobacterium- gener ved kultivering med Arabidopsis . Det blev yderligere påvist, at dette system er egnet til at studere Agrobacterium- vedhæftning til planterødder såvel som planterodssekretomprofilen ved kultivering (infektion) med Agrobacterium ( Figur 1 ).
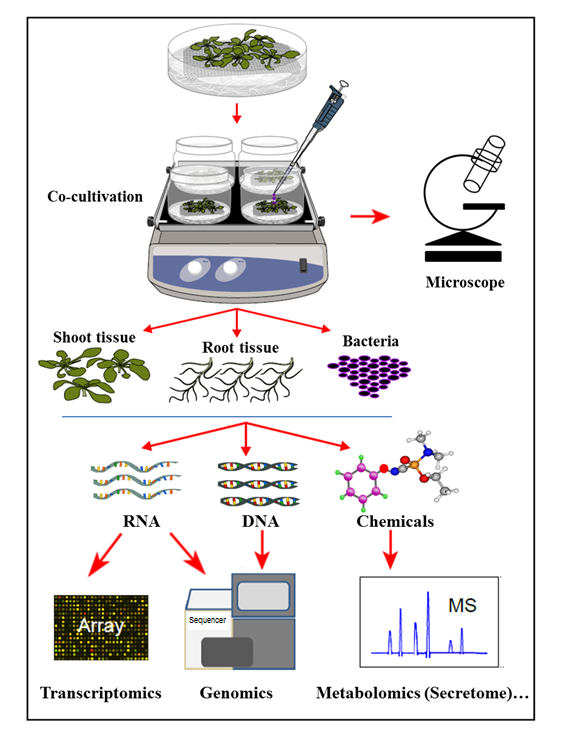
Figur 1: Oversigt over det hydroponiske kultiveringssystem med stikprøveanalyser. Planter dyrkes oven på masken (skud over masken), med rødderne nedsænket i hydroponisk medium, som derefter inokuleres med bakterier fEller kultivering. Plantevæv og bakterier separeres derefter til samtidige ekstraktioner og analyser. Dette tal er blevet ændret fra reference 35 .
Protocol
Representative Results
Discussion
I betragtning af den gradvise karakter af rodsekretion forekommer koncentrationen af virulensfremkaldende kemikalier produceret i planta og deres virkninger på dynamiske plante-mikrobe interaktioner i rumlige og temporale gradienter. I dette hydroponiske co-dyrkningssystem suppleres ingen syntetisk fytohormon eller kemikalie, som inducerer mikrobiell virulens eller plantebeskyttelse. I modsætning hertil skaber tilsætning af syntetiske kemikalier, såsom acetosyringon, en pludselig kunstig stigning i ko…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Vi vil gerne takke Brian Weselowski og Alexander W. Eastman for deres hjælp og nyttige diskussioner. Vi vil også gerne takke Drs. Eugene W. Nester, Lingrui Zhang, Haitao Shen, Yuhai Cui og Greg Thorn for deres hjælp, nyttige diskussioner og kritisk læsning af manuskriptet. Denne forskning blev finansieret af Agriculture and Agri-Food Canada, Growing Forward-AgriFlex (RBPI nummer 2555) og Growing Forward II-projekt nummer 1670, som blev udført af forfatterne som en del af deres opgaver. Denne undersøgelse blev også delvist finansieret af Canadian Science and Engineering Research Council (NSERC) Discovery Grant RGPIN-2015-06052 tildelt ZC Yuan.
Materials
| plant seeds (Arabidopsis thaliana Col-0) | Arabidopsis Biological Resource Centre | CS7000 | https://abrc.osu.edu/order-stocks |
| bacteria (Agrobacterium tumefaciens C58) | University of Washington | N/A | |
| labeled bacteria | in-house | optional, depends on downstream analyses | |
| vortex | (various) | ||
| microcentrifuge tubes | (various) | ||
| microcentrifuge | (various) | ||
| 5% sodium hypochlorite | (various) | ||
| double distilled water | (various) | ||
| autoclave | (various) | ||
| micropipette | (various) | ||
| 70 % ethanol | (various) | ||
| Murashige and Skoog (MS) basal salts | Sigma-Aldrich | M5524 | |
| sucrose | (various) | ||
| MES | (various) | ||
| B5 vitamin mix | Sigma-Aldrich | G1019 | |
| phytoagar | (various) | ||
| deep Petri dishes | (various) | ||
| stainless steel mesh | Ferrier Wire Goods Company Ltd | N/A | grade: 304; mesh count: 40 × 40; wire DIA: 0.01 |
| micropore tape, 1 inch | 3M | 1530-1 | |
| diurnal growth chamber | (various) | ||
| cylindrical glass tanks, 100 × 80 mm | Pyrex | 3250 | other sizes can be used, in which case liquid content may need adjustment |
| flow hood | (various) | ||
| forcepts | (various) | ||
| yeast extract | (various) | ||
| tryptone | (various) | ||
| MgSO4 | (various) | ||
| shaking incubator | (various) | ||
| spectrophotometer | (various) | ||
| NaCl | (various) | ||
| shaker | (various) | ||
| scissors | (various) | optional, depends on downstream analyses | |
| fluorescence microscope | (various) | optional, depends on downstream analyses | |
| microscope slides and cover slips | (various) | optional, depends on downstream analyses | |
| nail polish | (various) | optional, depends on downstream analyses | |
| Bacterial RNA extraction kit | (various) | optional, depends on downstream analyses | |
| plant RNA extraction kit (RNeasy Plant Mini Kit) | Qiagen | 74903 or 74904 | optional, depends on downstream analyses |
| material and equipment for RT-qPCR | (various) | optional, depends on downstream analyses | |
| material and equipment for microarray analysis | (various) | optional, depends on downstream analyses | |
| liquid nitrogen | (various) | optional, depends on downstream analyses | |
| mortar and pestle | (various) | optional, depends on downstream analyses | |
| 0.2 µm pore filter | (various) | optional, depends on downstream analyses | |
| 50 mL conical tubes | (various) | optional, depends on downstream analyses | |
| freeze dryer | (various) | optional, depends on downstream analyses | |
| sealable test tubes | (various) | optional, depends on downstream analyses | |
| ethyl acetate | (various) | optional, depends on downstream analyses | |
| nitrogen gas | (various) | optional, depends on downstream analyses | |
| material and equipment for HPLC | (various) | optional, depends on downstream analyses | |
| material and equipment for ESI-TOF-MS | (various) | optional, depends on downstream analyses |
References
- Lambers, H., Mougel, C., Jaillard, B., Hinsinger, P. Plant-microbe-soil interactions in the rhizosphere: An evolutionary perspective. Plant Soil. 321 (1), 83-115 (2009).
- Philippot, L., Raaijmakers, J. M., Lemanceau, P., vander Putten, W. H. Going back to the roots: The microbial ecology of the rhizosphere. Nat. Rev. Microbiol. 11 (11), 789-799 (2013).
- Somers, E., Vanderleyden, J., Srinivasan, M. Rhizosphere bacterial signalling: A Love Parade beneath our feet. Crit. Rev. Microbiol. 30 (4), 205-240 (2004).
- Barah, P., Winge, P., Kusnierczyk, A., Tran, D. H., Bones, A. M. Molecular signatures in Arabidopsis thaliana in response to insect attack and bacterial infection. PLoS ONE. 8 (3), (2013).
- Zhang, J., Zhou, J. -. M. Plant immunity triggered by microbial molecular signatures. Mol. Plant. 3 (5), 783-793 (2010).
- Paterson, E., Gebbing, T., Abel, C., Sim, A., Telfer, G. Rhizodeposition shapes rhizosphere microbial community structure in organic soil. New Phytol. 173 (3), 600-610 (2006).
- Hartmann, A., Schmid, M., van Tuinen, D., Berg, G. Plant-driven selection of microbes. Plant Soil. 321 (1-2), 235-257 (2008).
- Micallef, S. A., Shiaris, M. P., Colon-Carmona, A. Influence of Arabidopsis thaliana accessions on rhizobacterial communities and natural variation in root exudates. J. Exp. Bot. 60 (6), 1729-1742 (2009).
- Burr, T., Otten, L. Crown gall of grape: Biology and disease management. Annu. Rev. Phytopathol. 37, 53-80 (2001).
- Clough, S. J., Bent, A. F. Floral dip: A simplified method for Agrobacterium-mediated transformation of Arabidopsis thaliana. Plant J. 16 (6), 735-743 (1998).
- Binns, A. N., Costantino, P., Spaink, H. P., Kondorosi, A., Hooykaas, P. J. J. The Agrobacterium oncogenes. The Rhizobiaceae. , (1998).
- Gheysen, G., Angenon, G., Van Montagu, M., Lindsey, K. Agrobacterium-mediated plant transformation: a scientifically intriguing story with significant applications. Transgenic Plant Research. , (1998).
- Valvekens, D., Von Montagu, M. V., Van Lijsebettens, M. Agrobacterium tumefaciens-mediated transformation of Arabidopsis thaliana root explants by using kanamycin selection. Proc. Natl. Acad. Sci. USA. 85 (15), 5536-5540 (1988).
- Krysan, P. J., Young, J. C., Sussman, M. R. T-DNA as an insertional mutagen in Arabidopsis. Plant Cell. 11 (12), 2283 (1999).
- Chilton, M. D., et al. Stable incorporation of plasmid DNA into higher plant cells: The molecular basis of crown gall tumorigenesis. Cell. 11 (2), 263-271 (1977).
- Gelvin, S. B. Agrobacterium in the genomics age. Plant Physiol. 150 (4), 1665-1676 (2009).
- Subramoni, S., Nathoo, N., Klimov, E., Yuan, Z. -. C. Agrobacterium tumefaciens responses to plant-derived signaling molecules. Front. Plant Sci. 5, 322 (2014).
- Yuan, Z., Liu, P., Saenkham, P., Kerr, K., Nester, E. W. Transcriptome profiling and functional analysis of Agrobacterium tumefaciens reveals a general conserved response to acidic conditions (pH 5.5) and a complex acid-mediated signaling involved in Agrobacterium-plant interactions. J. Bacteriol. 190 (2), 494-507 (2008).
- Stachel, S. E., Messens, E., Van Montagu, M., Zambryski, P. Identification of the signal molecules produced by wounded plant cells that activate T-DNA transfer in Agrobacterium tumefaciens. Nature. 318 (6047), 624-629 (1985).
- Memelink, J., de Pater, B. S., Hoge, J. H. C., Schilperoort, R. A. T-DNA hormone biosynthetic genes: Phytohormones and gene expression in plants. Dev. Genet. 8 (5-6), 321-337 (1987).
- Yuan, Z. -. C., et al. The plant signal salicylic acid shuts down expression of the vir regulon and activates quormone-quenching genes in Agrobacterium. Proc. Natl. Acad. Sci. USA. 104 (28), 11790-11795 (2007).
- Yuan, Z. -. C., Haudecoeur, E., Faure, D., Kerr, K. F., Nester, E. W. Comparative transcriptome analysis of Agrobacterium tumefaciens in response to plant signal salicylic acid, indole-3-acetic acid and γ-amino butyric acid reveals signalling cross-talk and Agrobacterium-plant co-evolution. Cell. Microbiol. 10 (11), 2339-2354 (2008).
- Li, P. L., Farrand, S. K. The replicator of the nopaline-type Ti plasmid pTiC58 is a member of the repABC family and is influenced by the TraR-dependent quorum-sensing regulatory system. J. Bacteriol. 182 (1), 179-188 (2000).
- Atkinson, M. M., Huang, J., Knopp, J. A. Hypersensitivity of suspension-cultured tobacco cells to pathogenic bacteria. Phytopathology. 75 (11), 1270-1274 (1985).
- Veena, ., Jiang, H., Doerge, R. W., Gelvin, S. B. Transfer of T-DNA and Vir proteins to plant cells by Agrobacterium tumefaciens induces expression of host genes involved in mediating transformation and suppresses host defense gene expression. Plant J. 35 (2), 219-236 (2003).
- León, J., Rojo, E., Sanchez-Serrano, J. J. Wound signalling in plants. J. Exp. Bot. 52 (354), 1-9 (2001).
- Ditt, R. F., Kerr, K. F., de Figueiredo, P., Delrow, J., Comai, L., Nester, E. W. The Arabidopsis thaliana transcriptome in response to Agrobacterium tumefaciens. Mol. Plant Microbe In. 19 (6), 665-681 (2006).
- Mandimba, G., Heulin, T., Bally, R., Guckert, A., Balandreau, J. Chemotaxis of free-living nitrogen-fixing bacteria towards maize mucilage. Plant Soil. 90 (1-3), 129-139 (1986).
- Rudrappa, T., Czymmek, K. J., Pare, P. W., Bais, H. P. Root-secreted malic acid recruits beneficial soil bacteria. Plant Physiol. 148 (3), 1547-1556 (2008).
- Lee, C. W., et al. Agrobacterium tumefaciens promotes tumor induction by modulating pathogen defense in Arabidopsis thaliana. Plant Cell. 21 (9), 2948-2962 (2009).
- Reymond, P., Weber, H., Damond, M., Farmer, E. E. Differential gene expression in response to mechanical wounding and insect feeding in Arabidopsis. Plant Cell. 12 (5), 707-720 (2000).
- Matthysse, A. G. Attachment of Agrobacterium to plant surfaces. Front. Plant Sci. 5, 252 (2014).
- Alatorre-Cobos, F., et al. An improved, low-cost, hydroponic system for growing Arabidopsis and other plant species under aseptic conditions. BMC Plant Biol. 14 (1), 69 (2014).
- Conn, S. J., et al. Protocol: Optimising hydroponic growth systems for nutritional and physiological analysis of Arabidopsis thaliana and other plants. Plant Methods. 9 (1), 4 (2013).
- Nathoo, N. . Identification of putative plant defense genes using a novel hydroponic co-cultivation technique for studying plant-pathogen interaction. , (2015).
- Shaner, N. C., Campbell, R. E., Steinbach, P. A., Giepmans, B. N., Palmer, A. E., Tsien, R. Y. Improved monomeric red, orange and yellow fluorescent proteins derived from Discosoma sp. red fluorescent protein. Nat. Biotechnol. 22 (12), 1567-1572 (2004).
- Wise, A. A., Liu, H., Binns, A. N. Nucleic acid extraction from Agrobacterium strains. Methods Mol. Bio. 343, 67-76 (2006).
- Nolan, T., Hands, R. E., Bustin, S. A. Quantification of mRNA using real-time RT-PCR. Nat. Protoc. 1 (3), 1559-1582 (2006).
- Salter, M. G., Conlon, H. E. Extraction of plant RNA. Methods Mol. Bio. 362, 309-314 (2007).
- Bao, Y., Wang, S., Yang, X., Li, T., Xia, Y., Meng, X. Metabolomic study of the intervention effects of Shuihonghuazi Formula, a Traditional Chinese Medicinal formulae, on hepatocellular carcinoma (HCC) rats using performance HPLC/ESI-TOF-MS. J. Ethnopharmacol. 198, 468-478 (2017).
- Korves, T. M., Bergelson, J. A developmental response to pathogen infection in Arabidopsis. Plant Physiol. 133 (1), 339-347 (2003).
- Lyons, R., Rusu, A., Stiller, J., Powell, J., Manners, J. M., Kazan, K. Investigating the association between flowering time and defense in the Arabidopsis thaliana-Fusarium oxysporum interaction. PLoS ONE. 10 (6), e0127699 (2015).
- Badri, D. V., Weir, T. L., vander Lelie, D., Vivanco, J. M. Rhizosphere chemical dialogues: Plant-microbe interactions. Curr. Opin. Biotechnol. 20 (6), 642-650 (2009).
- Baerson, S. R., et al. Detoxification and transcriptome response in Arabidopsis seedlings exposed to the allelochemical benzoxazolin-2(3H)-one. J. Biol. Chem. 280 (23), 21867-21881 (2005).

