संयंत्र / सूक्ष्मजीव आणविक संबंधों और सिगनलिंग के एक साथ और व्यवस्थित विश्लेषण के लिए एक हाइड्रोपोनिक सह-खेती प्रणाली
Summary
वर्णित हाइड्रोपोनिक cocultivation प्रणाली धातु जाल स्क्रीन के साथ बरकरार पौधों का समर्थन करता है और बैक्टीरिया के साथ उन्हें cocultivates। संयंत्र के ऊतक, बैक्टीरिया और स्रावित अणुओं को फिर से नीचे की ओर के विश्लेषण के लिए अलग-अलग काटा जा सकता है, साथ ही साथ दोनों पौधों के मेजबानों के आणविक प्रतिक्रियाओं की जांच की जा सकती है और जांच की जाने वाली सूक्ष्मजीवों या सूक्ष्मजीवों के बारे में बातचीत की जा सकती है।
Abstract
जटिल पौधे-सूक्ष्म सिग्नल प्रक्रियाओं को चित्रित करने के लिए प्राकृतिक पौधे-सूक्ष्म जीवों की बातचीत का अनुकरण करना एक प्रायोगिक डिजाइन है। अरबिडोप्सिस थालियाना – एग्रोबैक्टीरियम ट्यूमेफिएन्स जीवाणु रोगजनन और पौधों की बातचीत का अध्ययन करने के लिए एक उत्कृष्ट मॉडल प्रणाली प्रदान करता है। पौधे का पिछला अध्ययन – एग्रोबैक्टीरियम परस्पर क्रियाएं बड़े पैमाने पर संयंत्र सेल निलंबन संस्कृतियों, पौधों के कृत्रिम घावों या कृत्रिम रूप से माइक्रोबियल वायरलेंस कारक या कृत्रिम रसायनों द्वारा पौधे की सुरक्षा पर भरोसा करती हैं। हालांकि, पौधों और सूक्ष्म जीवों को स्थानिक और लौकिक शिष्टाचारों में पहचान और जवाब देने में पौधों में प्राकृतिक सिगनलिंग से ये विधि अलग-अलग हैं। यह काम हाइड्रोपोनिक कोकोल्टीविएशन सिस्टम को प्रस्तुत करता है जहां बरकरार पौधों को धातु जाल स्क्रीनों द्वारा समर्थित किया जाता है और एग्रोबैक्टीरियम के साथ उर्वरित होता है। इस cocultivation प्रणाली में, कोई सिंथेटिक phytohormone या रासायनिक कि micr लाती हैओपिअल वायरलेंस या प्लांट डिफेन्स पूरक है। हाइड्रोपोनिक cocultivation प्रणाली बारीकी से प्राकृतिक पौधों-सूक्ष्म जीव बातचीत और planta में संकेत समस्थिति जैसा दिखता है। संयंत्र जड़ों को एग्रोबैक्टेरियम युक्त माध्यम से अलग किया जा सकता है, और दोनों संयंत्र मेजबानों के संकेत और प्रतिक्रियाएं और इंटरैक्टिंग रोगाणुओं की एक साथ और व्यवस्थित रूप से जांच की जा सकती है। किसी भी समय के अंतराल / अंतराल पर, पौधों के ऊतकों या बैक्टीरिया को विभिन्न "ओमिक्स" विश्लेषणों के लिए अलग से काटा जा सकता है, इस प्रणाली की शक्ति और प्रभावकारिता का प्रदर्शन। हाइड्रोपोनिक कोकोलिटिवेशन सिस्टम को आसानी से अध्ययन करने के लिए अनुकूलित किया जा सकता है: 1) विविध पौधे-सूक्ष्म जीवों के सिस्टम के पारस्परिक संकेत, 2) एक प्लांट होस्ट और कई माइक्रोबियल प्रजातियों ( यानी माइक्रोबियल कॉन्सर्टिया या माइक्रोबोमास) के बीच सिग्नलिंग, 3) पोषक तत्वों और रसायनों को किस प्रकार फंसाया जाता है पौधे-सूक्ष्म सिग्नलिंग में, और 4) कैसे रोगाणुओं संयंत्र मेजबान के साथ बातचीत करते हैं और जैविक ओ के लिए पौधे की सहिष्णुता में योगदान करते हैंआर एबियोटिक तनाव
Introduction
संयंत्र से जुड़े रोगाणुओं में जैव-रासायनिक साइक्लिंग, बायोरेमेडिएशन, जलवायु परिवर्तन, पौधों की वृद्धि और स्वास्थ्य का शमन, जैविक और अबायटीक तनावों के लिए पौधे सहिष्णुता में महत्वपूर्ण भूमिका निभाएं। सूक्ष्मजीव पौधों की कोशिका दीवार संपर्क के माध्यम से और परोक्ष रूप से रासायनिक स्राव के माध्यम से और 1 , 2 , 3 के संकेत के माध्यम से पौधों के साथ बातचीत करते हैं। जीवाणुओं द्वारा संक्रमण का विरोध करने के लिए पौधों ने प्रत्यक्ष और अप्रत्यक्ष तंत्र विकसित किए हैं। डायरेक्ट डिफेंस में स्ट्रक्चरल डिफेंस और रक्षा प्रोटीन की अभिव्यक्ति शामिल है, जबकि अप्रत्यक्ष सुरक्षा में द्वितीयक पौधे के मेटाबोलाइट उत्पादन और रोगज़नुओं 4 , 5 पर हमला करने वाले जीवों के आकर्षण शामिल हैं। प्लांट-व्युत्पन्न रूट एक्स्टेट्स, स्राक्र्रिज, म्यूसीलाज, म्यूसिगेल, और लाइसेट्स, रेजोस्फीयर की भौतिक-रासायनिक गुणों को आकर्षित करने या पीछे हटानारोगाणुओं की मेजबानी 6 मूल स्राव की रासायनिक संरचना प्रजाति-विशिष्ट है, जिससे एक चयनात्मक फिल्टर के रूप में कार्य किया जाता है जो ऐसे सूक्ष्मजीवों को ऐसे यौगिकों को पहचानने में सक्षम बनाता है जो कि rhizosphere 6 में पनपने में सक्षम है। इस प्रकार, संगत माइक्रोबियल प्रजातियों को अपने संगठनों को सक्रिय करने और बढ़ाने के लिए प्रोत्साहित किया जा सकता है, या तो संयंत्र होस्ट 1 के लाभ या हानि के लिए।
पौध-सूक्ष्म जीवों की बातचीत को समझना, पौध उत्पादकता और पारिस्थितिक तंत्र के कामकाज को बढ़ाने के लिए महत्वपूर्ण है, चूंकि अधिकांश माइक्रोबियल और रासायनिक एक्सपोजर रूट स्ट्रक्चर और मिट्टी-एयर इंटरफेस 2 , 6 , 7 , 8 में होता है । हालांकि, भूगर्भीय पौधे-सूक्ष्म जीवों की बातचीत और पारस्परिक प्रतिक्रियाओं की परीक्षा इसकी चतुराई से एक चुनौती रही है जटिल और गतिशील प्रकृति और प्राकृतिक रूट संरचना और पौधों के आकारिकी के साथ उपयुक्त प्रायोगिक मॉडल की कमी, कसकर नियंत्रित होने वाली वृद्धि स्थितियों के तहत। सबसे ज्यादा अध्ययन किए गए फाइटॉपैथोजेन के रूप में, एग्रोबैक्टेरियम , चेरी, सेब, नाशपाती, अंगूर और 9 गुलाब सहित कृषि और बागवानी महत्व के साथ पौधों की एक विस्तृत श्रृंखला को संक्रमित करता है। एग्रोबेक्टेरियम संयंत्र-रोगज़नक़ व्यवहार को समझने के लिए एक महत्वपूर्ण मॉडल जीव है और संयंत्र परिवर्तन और संयंत्र इंजीनियरिंग 10 , 11 , 12 , 13 , 14 में एक शक्तिशाली उपकरण है।
आणविक पौधे- एग्रोबैक्टेरियम बातचीत का कई दशकों से अच्छी तरह से अध्ययन किया गया है, और एग्रोबैक्टीरियम रोगजनकता की वर्तमान समझ 9 व्यापक है ,एफ "> 11 , 15 , 16. एग्रोबैक्टेरीयम रोगजनकता काफी हद तक पौधों से प्राप्त संकेतों को समझने की अपनी विकसित क्षमताओं को जिम्मेदार ठहराता है, जिसके परिणामस्वरूप इसके खतरे कार्यक्रम और सेल-टू-सेल संचार, तथाकथित कोरम सेंसिंग 17 का ठीक स्वरुपरण हो जाता है। एग्रोबैक्टेरियम वायरलेंस प्रोग्राम को कई सिग्नल द्वारा विनियमित किया जाता है जो कि रेजोस्फीयर में उपलब्ध होता है और इसमें 2-घटक प्रणालियों के दो सेट, च्वीजी / आई सिस्टम और वीरिया / जी सिस्टम शामिल होते हैं। रेजोस्फ़ेयर में एसिडिक सिस्टम्स , सीएचजी / आई , वीएए / जी के प्रतिलेखन को सक्रिय करते हैं। , और कई अन्य virE0, virE1, virH1, virH2, और प्रकार छठी स्राव प्रणाली (T6SS) 18। के जीन सहित एग्रोबैक्टीरियम pathogenicity, में शामिल जीनों संयंत्र व्युत्पन्न acetosyringone (4'-हाइड्रोक्सी-3 ', 5 सहित फेनिलक यौगिकों, '-डिमैथॉक्सीएसीटोपोनोन), वी को सक्रिय करेंफास्फारिलीकरण संकेत तंत्र 19 के माध्यम से ईरा / जी 2-घटक प्रणाली। वीरा / जी तो पूरे वीर regulon सक्रिय करता है, इसके ट्यूमर उत्प्रेरण (ती) प्लाज्मिड से हस्तांतरण और एक ~ 20 केबी बैक्टीरियल डीएनए टुकड़ा बुलाया हस्तांतरण डीएनए (टी डीएनए) के एकीकरण संयंत्र नाभिक 16 में हो जाती है। टी डीएनए संयंत्र के संश्लेषण के लिए जिम्मेदार जीनों वहन करती हार्मोन इण्डोल-3- एसिटिक एसिड (आईएए) (iaaM और iaaH) और साइटोकिनिन (IPT), और एक बार पौधों की कोशिकाओं में व्यक्त किया, इन phytohormones की बड़ी मात्रा में उत्पादन कर रहे हैं। यह असामान्य ऊतक प्रसार और पौधे के ट्यूमर के विकास का परिणाम है, जिसे ताज पल रोग कहा जाता है, जो पौधों 9 , 11 , 20 के लिए एक पुरानी और पुनरुत्थान समस्या है। एएएए एग्रोबैक्टेरियम विषाक्तता को दबाने के लिए या एग्रोबैक्टीरियु को कम करने के लिए साल्लिसिलिक एसिड और गामा-एमिनो बोटोइरिक एसिड के साथ सामूहिक रूप से कार्य करता है मीटर कोरम संवेदन (QS) 17, 21, 22। इस दमन का मुकाबला करने के लिए, टी-डीएनए ऑप्लेन बायोसिंथेथेसिस के लिए जीन भी करता है, जो एग्रोबैक्टेरीयम कोरोमम को एग्रोबैक्टेरीयम पैथोजेनिकता को बढ़ावा देने के लिए सक्रिय करता है और यह 22 , 23 रोगज़नक़ों के लिए पोषक स्रोत के रूप में भी कार्य करता है।
एग्रोबैक्टीरियम -प्लान्ट इंटरैक्शन की पूरी गहरी समझ और परिणामस्वरूप प्लांट होस्ट में टी-डीएनए हस्तांतरण के बावजूद, बातचीत के प्रारंभिक चरण में जटिल सिग्नलिंग इवेंट कम अच्छी तरह से समझ गए हैं। यह एग्रोबैक्टीरियम- प्लांट सिग्नलिंग की जांच के लिए पारंपरिक तरीकों की सीमाओं के कारण आंशिक रूप से है। प्लांट सेल निलंबन संस्कृतियों और कृत्रिम साइट-विशिष्ट घावों को आम तौर पर आणविक पौधे-सूक्ष्म जीवों की बातचीत का अध्ययन करने के लिए 24 ,26 , 27. हालांकि, सेल निलंबन में विशिष्ट पौधों के आकारिकी की कमी होती है, विशेष रूप से, पौधे निलंबन कोशिकाओं में जड़ संरचना और रूट एक्सयूड्स नहीं होते हैं, जो कि माइक्रोबियल केमोटाक्सिस और वायरलेंस 28 , 29 को सक्रिय करने के लिए बहुत महत्वपूर्ण हैं। और रूट संरचना कृत्रिम रूप से घायल हुए पौधों द्वारा संबोधित किया गया है, जो साइट-विशिष्ट संक्रमण की सुविधा देता है, जिसके परिणामस्वरूप प्रत्यक्ष रूप से संक्रमित पौधे के ऊतक 30 , 31 में प्रेरित पौधे रक्षा संबंधी जीनों का पता लगाना पड़ता है। हालांकि, कृत्रिम घाव प्रकृति में रोगज़नक़ों के संक्रमण से काफी अलग है विशेष रूप से घायल होने से जशोनीक एसिड (जेए) संचय होता है, जो प्राकृतिक रूप से प्राकृतिक पौधों के संकेत और बचाव के साथ हस्तक्षेप करता है 26. इसके अलावा, सिंथेटिक रसायनों को आम तौर पर कृत्रिम रूप से पौधों के मेजबान प्रतिक्रियाओं को प्रेरित करने के लिए प्रयोग किया जाता हैया रोगजन विषाक्तता हालांकि planta में सांद्रता को प्रतिबिंबित करता इस तरह के रासायनिक यौगिकों के पूरकता संभव है, इस तरह के पूरकता जड़ रिसाव के प्रसार धीरे-धीरे आसपास के rhizosphere है, जो एक कीमोटैक्टिक ढाल रोगाणुओं 28, 32 द्वारा महसूस उत्पन्न में के लिए खाते में नहीं है। पौधे-सूक्ष्म जीवों की बातचीत का अध्ययन करने के लिए पारंपरिक दृष्टिकोण की सीमाओं को देखते हुए, प्राप्त आंकड़ों की सटीकता और गहराई को बाधित और प्रतिबंधात्मक बनाया जा सकता है, और परंपरागत तरीकों से उत्पन्न ज्ञान सीधे पौधे में अनुवाद नहीं हो सकता है। पौधे के कई पहलू- एग्रोबेक्टेरियम सिगनलिंग अभी तक पूरी तरह से समझा नहीं गया है, विशेष रूप से अंतःक्रियाओं के प्रारंभिक चरण में, जब बीमारी के लक्षण अभी तक विकसित नहीं हुए हैं।
पारंपरिक तरीकों की सीमाओं में संशोधन करने के लिए, यह काम एक सस्ती, कसकर नियंत्रणीय, और लचीला हाइड्रोपोनिक सी प्रस्तुत करता है।ऑक्विलिवेशन सिस्टम जो शोधकर्ताओं को आणविक पौधे-सूक्ष्म जीवों की बातचीत के प्रारंभिक चरण में जटिल संकेत और प्रतिक्रिया के रास्ते में गहरी अंतर्दृष्टि प्राप्त करने की अनुमति देता है। पौधों के पौधों, जड़ exudates, विकास की स्थिति, और पौधों 33 , 34 पर धातु विषाक्तता के प्रभाव का अध्ययन करने के लिए हीड्रोपोनिक्स का व्यापक रूप से उपयोग किया गया है। छोटे स्थानिक आवश्यकताओं, विभिन्न पौधे के ऊतकों की पहुंच, पोषक तत्व / पर्यावरणीय परिस्थितियों के नियंत्रण और कीट / बीमारी नियंत्रण के साथ हीड्रोपोनिक मॉडल के कई फायदे हैं। हाइड्रोपोनिक प्रणालियों, एगर / फिटोआगर चढ़ाना तकनीक की तुलना में पौधे वृद्धि के लिए सीमित हैं, जो आमतौर पर 2-3 सप्ताह के बाद वृद्धि को सीमित करती हैं। महत्वपूर्ण रूप से, पूरे संयंत्र संरचनाओं के रखरखाव से माइक्रोबियल केमोटाक्सिस और वायरलेंस इंश्योशन 8 , 2 9 के लिए आवश्यक प्राकृतिक रूट स्राक्रिशन की सुविधा मिलती है। प्रणाली descriयहां पर बिस्तर 33 , 34 के मुकाबले सरल और कम श्रमिक है यह कम भागों का उपयोग करता है और मानक कैंची के अलावा किसी अन्य उपकरण की आवश्यकता नहीं होती है। यह धातु के जाल (जैसा नायलॉन 33 का विरोध करता है) का उपयोग करता है, पौधों की वृद्धि के लिए एक मजबूत समर्थन और माइक्रोबियल विकास का समर्थन करने के लिए मिलाते हुए बाँझ शर्तों के तहत वातन की एक सरल विधि के रूप में। इसके अलावा, सिस्टम पौधों की वृद्धि का समर्थन करने के लिए विभिन्न आकारों के धातु के जाल का उपयोग कर सकता है, जो अपनी जड़ों की चौड़ाई को सीमित किए बिना विविध पौधों की प्रजातियों को समायोजित करता है।
यहां प्रस्तुत हाइड्रोपोनिक नगरीय तंत्र में, पौधों को एक बाँझ हीड्रोपोनिक प्रणाली में खेती की जाती है जहां पौधों की जड़ों में इनोक्लेटेड बैक्टीरिया के विकास के लिए कार्बनिक यौगिकों को छिपाना होता है। इस cocultivation प्रणाली में, कोई कृत्रिम रसायनों, जैसे संयंत्र हार्मोन, रक्षा elicitor, या virulence-inducing रसायनों, पूरक हैं, जो प्राकृतिक सेल को दर्शाता हैपौध-सूक्ष्म अंतर के दौरान साइनऑनिंग होमोस्टेसिस इस हाइड्रोपोनिक cocultivation प्रणाली के साथ, यह संभव था एक साथ एग्रोबैक्टीरियम द्वारा संक्रमण पर Arabidopsis थालिअना कर्नल -0 जड़ ऊतकों में जीन की अभिव्यक्ति है, साथ ही Arabidopsis साथ cocultivation पर एग्रोबैक्टीरियम जीन की सक्रियता निर्धारित करने के लिए। यह आगे दिखाया गया कि यह तंत्र एग्रोबैक्टीरियम ( चित्रा 1 ) के साथ सिकुड़ना (संक्रमण) पर पौधे जड़ों के साथ-साथ पौधे की जड़ों के लिए एग्रोबैक्टीरियम लगाव का अध्ययन करने के लिए उपयुक्त है।
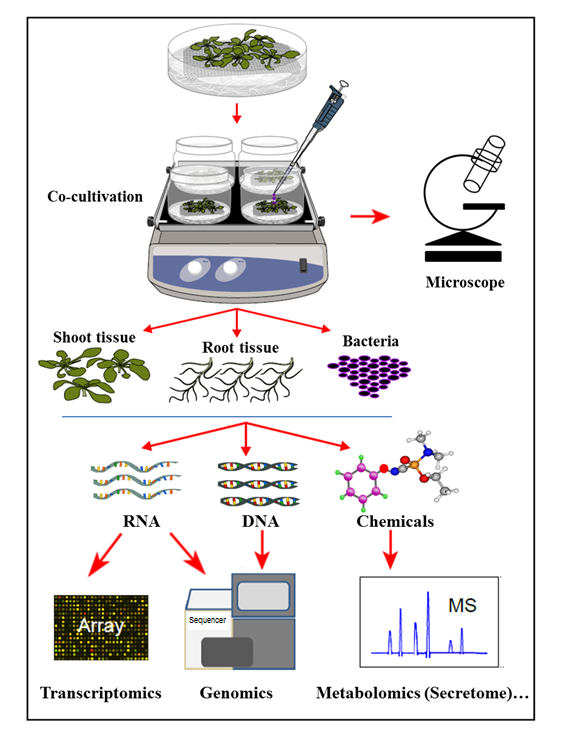
चित्रा 1: नमूना विश्लेषण के साथ, हाइड्रोपोनिक कोकिल्टी सिस्टम का अवलोकन। पौधों मेष के शीर्ष पर बढ़ी जाती हैं (जाल के ऊपर गोली मारती है), जलीय वायुमंडल में डूबे जड़ों के साथ, जो तब जीवाणुओं के साथ inoculated हैया कोकल्चर संयंत्र के ऊतकों और बैक्टीरिया को एक साथ निष्कर्षों और विश्लेषण के लिए अलग किया जाता है। यह आंकड़ा संदर्भ 35 से संशोधित किया गया है।
Protocol
Representative Results
Discussion
जड़ स्राव की क्रमिक प्रकृति को देखते हुए, प्लाया में उत्पन्न होने वाली जहरीले -उत्प्रेरण रसायनों की एकाग्रता और गतिशील पौधे-सूक्ष्म जीवों के संबंधों पर उनके प्रभावों को स्थानिक और लौकिक ग्रेड?…
Disclosures
The authors have nothing to disclose.
Acknowledgements
हम ब्रायन वस्लोव्स्की और सिकंदर डब्ल्यू ईस्टमैन को उनकी मदद और उपयोगी चर्चा के लिए धन्यवाद देना चाहेंगे। हम भी डीआरएस को धन्यवाद देना चाहेंगे। यूजीन डब्ल्यू। नेस्टर, लिंगुई झांग, हेटाओ शेन, यूहई कुई, और ग्रेग थॉर्न उनकी मदद के लिए, उपयोगी बातचीत, और पांडुलिपि का महत्वपूर्ण पढ़ना। इस शोध को कृषि और कृषि-खाद्य कनाडा, ग्रोइंग फॉरवर्ड-एग्रीफ्लक्स (आरबीपीआई नंबर 2555) और फॉरवर्ड II प्रोजेक्ट संख्या 1670 की बढ़ोतरी के लिए वित्त पोषित किया गया था, जो उनके कर्तव्यों के एक हिस्से के रूप में लेखकों द्वारा आयोजित किया गया था। इस अध्ययन को आंशिक तौर पर प्राकृतिक विज्ञान और इंजीनियरिंग रिसर्च काउंसिल ऑफ कनाडा (एनएसईआरसी) डिस्कवरी ग्रांट आरजीपीआईएन-2015-06052 द्वारा ज़ेडसी युआन को प्रदान किया गया था।
Materials
| plant seeds (Arabidopsis thaliana Col-0) | Arabidopsis Biological Resource Centre | CS7000 | https://abrc.osu.edu/order-stocks |
| bacteria (Agrobacterium tumefaciens C58) | University of Washington | N/A | |
| labeled bacteria | in-house | optional, depends on downstream analyses | |
| vortex | (various) | ||
| microcentrifuge tubes | (various) | ||
| microcentrifuge | (various) | ||
| 5% sodium hypochlorite | (various) | ||
| double distilled water | (various) | ||
| autoclave | (various) | ||
| micropipette | (various) | ||
| 70 % ethanol | (various) | ||
| Murashige and Skoog (MS) basal salts | Sigma-Aldrich | M5524 | |
| sucrose | (various) | ||
| MES | (various) | ||
| B5 vitamin mix | Sigma-Aldrich | G1019 | |
| phytoagar | (various) | ||
| deep Petri dishes | (various) | ||
| stainless steel mesh | Ferrier Wire Goods Company Ltd | N/A | grade: 304; mesh count: 40 × 40; wire DIA: 0.01 |
| micropore tape, 1 inch | 3M | 1530-1 | |
| diurnal growth chamber | (various) | ||
| cylindrical glass tanks, 100 × 80 mm | Pyrex | 3250 | other sizes can be used, in which case liquid content may need adjustment |
| flow hood | (various) | ||
| forcepts | (various) | ||
| yeast extract | (various) | ||
| tryptone | (various) | ||
| MgSO4 | (various) | ||
| shaking incubator | (various) | ||
| spectrophotometer | (various) | ||
| NaCl | (various) | ||
| shaker | (various) | ||
| scissors | (various) | optional, depends on downstream analyses | |
| fluorescence microscope | (various) | optional, depends on downstream analyses | |
| microscope slides and cover slips | (various) | optional, depends on downstream analyses | |
| nail polish | (various) | optional, depends on downstream analyses | |
| Bacterial RNA extraction kit | (various) | optional, depends on downstream analyses | |
| plant RNA extraction kit (RNeasy Plant Mini Kit) | Qiagen | 74903 or 74904 | optional, depends on downstream analyses |
| material and equipment for RT-qPCR | (various) | optional, depends on downstream analyses | |
| material and equipment for microarray analysis | (various) | optional, depends on downstream analyses | |
| liquid nitrogen | (various) | optional, depends on downstream analyses | |
| mortar and pestle | (various) | optional, depends on downstream analyses | |
| 0.2 µm pore filter | (various) | optional, depends on downstream analyses | |
| 50 mL conical tubes | (various) | optional, depends on downstream analyses | |
| freeze dryer | (various) | optional, depends on downstream analyses | |
| sealable test tubes | (various) | optional, depends on downstream analyses | |
| ethyl acetate | (various) | optional, depends on downstream analyses | |
| nitrogen gas | (various) | optional, depends on downstream analyses | |
| material and equipment for HPLC | (various) | optional, depends on downstream analyses | |
| material and equipment for ESI-TOF-MS | (various) | optional, depends on downstream analyses |
References
- Lambers, H., Mougel, C., Jaillard, B., Hinsinger, P. Plant-microbe-soil interactions in the rhizosphere: An evolutionary perspective. Plant Soil. 321 (1), 83-115 (2009).
- Philippot, L., Raaijmakers, J. M., Lemanceau, P., vander Putten, W. H. Going back to the roots: The microbial ecology of the rhizosphere. Nat. Rev. Microbiol. 11 (11), 789-799 (2013).
- Somers, E., Vanderleyden, J., Srinivasan, M. Rhizosphere bacterial signalling: A Love Parade beneath our feet. Crit. Rev. Microbiol. 30 (4), 205-240 (2004).
- Barah, P., Winge, P., Kusnierczyk, A., Tran, D. H., Bones, A. M. Molecular signatures in Arabidopsis thaliana in response to insect attack and bacterial infection. PLoS ONE. 8 (3), (2013).
- Zhang, J., Zhou, J. -. M. Plant immunity triggered by microbial molecular signatures. Mol. Plant. 3 (5), 783-793 (2010).
- Paterson, E., Gebbing, T., Abel, C., Sim, A., Telfer, G. Rhizodeposition shapes rhizosphere microbial community structure in organic soil. New Phytol. 173 (3), 600-610 (2006).
- Hartmann, A., Schmid, M., van Tuinen, D., Berg, G. Plant-driven selection of microbes. Plant Soil. 321 (1-2), 235-257 (2008).
- Micallef, S. A., Shiaris, M. P., Colon-Carmona, A. Influence of Arabidopsis thaliana accessions on rhizobacterial communities and natural variation in root exudates. J. Exp. Bot. 60 (6), 1729-1742 (2009).
- Burr, T., Otten, L. Crown gall of grape: Biology and disease management. Annu. Rev. Phytopathol. 37, 53-80 (2001).
- Clough, S. J., Bent, A. F. Floral dip: A simplified method for Agrobacterium-mediated transformation of Arabidopsis thaliana. Plant J. 16 (6), 735-743 (1998).
- Binns, A. N., Costantino, P., Spaink, H. P., Kondorosi, A., Hooykaas, P. J. J. The Agrobacterium oncogenes. The Rhizobiaceae. , (1998).
- Gheysen, G., Angenon, G., Van Montagu, M., Lindsey, K. Agrobacterium-mediated plant transformation: a scientifically intriguing story with significant applications. Transgenic Plant Research. , (1998).
- Valvekens, D., Von Montagu, M. V., Van Lijsebettens, M. Agrobacterium tumefaciens-mediated transformation of Arabidopsis thaliana root explants by using kanamycin selection. Proc. Natl. Acad. Sci. USA. 85 (15), 5536-5540 (1988).
- Krysan, P. J., Young, J. C., Sussman, M. R. T-DNA as an insertional mutagen in Arabidopsis. Plant Cell. 11 (12), 2283 (1999).
- Chilton, M. D., et al. Stable incorporation of plasmid DNA into higher plant cells: The molecular basis of crown gall tumorigenesis. Cell. 11 (2), 263-271 (1977).
- Gelvin, S. B. Agrobacterium in the genomics age. Plant Physiol. 150 (4), 1665-1676 (2009).
- Subramoni, S., Nathoo, N., Klimov, E., Yuan, Z. -. C. Agrobacterium tumefaciens responses to plant-derived signaling molecules. Front. Plant Sci. 5, 322 (2014).
- Yuan, Z., Liu, P., Saenkham, P., Kerr, K., Nester, E. W. Transcriptome profiling and functional analysis of Agrobacterium tumefaciens reveals a general conserved response to acidic conditions (pH 5.5) and a complex acid-mediated signaling involved in Agrobacterium-plant interactions. J. Bacteriol. 190 (2), 494-507 (2008).
- Stachel, S. E., Messens, E., Van Montagu, M., Zambryski, P. Identification of the signal molecules produced by wounded plant cells that activate T-DNA transfer in Agrobacterium tumefaciens. Nature. 318 (6047), 624-629 (1985).
- Memelink, J., de Pater, B. S., Hoge, J. H. C., Schilperoort, R. A. T-DNA hormone biosynthetic genes: Phytohormones and gene expression in plants. Dev. Genet. 8 (5-6), 321-337 (1987).
- Yuan, Z. -. C., et al. The plant signal salicylic acid shuts down expression of the vir regulon and activates quormone-quenching genes in Agrobacterium. Proc. Natl. Acad. Sci. USA. 104 (28), 11790-11795 (2007).
- Yuan, Z. -. C., Haudecoeur, E., Faure, D., Kerr, K. F., Nester, E. W. Comparative transcriptome analysis of Agrobacterium tumefaciens in response to plant signal salicylic acid, indole-3-acetic acid and γ-amino butyric acid reveals signalling cross-talk and Agrobacterium-plant co-evolution. Cell. Microbiol. 10 (11), 2339-2354 (2008).
- Li, P. L., Farrand, S. K. The replicator of the nopaline-type Ti plasmid pTiC58 is a member of the repABC family and is influenced by the TraR-dependent quorum-sensing regulatory system. J. Bacteriol. 182 (1), 179-188 (2000).
- Atkinson, M. M., Huang, J., Knopp, J. A. Hypersensitivity of suspension-cultured tobacco cells to pathogenic bacteria. Phytopathology. 75 (11), 1270-1274 (1985).
- Veena, ., Jiang, H., Doerge, R. W., Gelvin, S. B. Transfer of T-DNA and Vir proteins to plant cells by Agrobacterium tumefaciens induces expression of host genes involved in mediating transformation and suppresses host defense gene expression. Plant J. 35 (2), 219-236 (2003).
- León, J., Rojo, E., Sanchez-Serrano, J. J. Wound signalling in plants. J. Exp. Bot. 52 (354), 1-9 (2001).
- Ditt, R. F., Kerr, K. F., de Figueiredo, P., Delrow, J., Comai, L., Nester, E. W. The Arabidopsis thaliana transcriptome in response to Agrobacterium tumefaciens. Mol. Plant Microbe In. 19 (6), 665-681 (2006).
- Mandimba, G., Heulin, T., Bally, R., Guckert, A., Balandreau, J. Chemotaxis of free-living nitrogen-fixing bacteria towards maize mucilage. Plant Soil. 90 (1-3), 129-139 (1986).
- Rudrappa, T., Czymmek, K. J., Pare, P. W., Bais, H. P. Root-secreted malic acid recruits beneficial soil bacteria. Plant Physiol. 148 (3), 1547-1556 (2008).
- Lee, C. W., et al. Agrobacterium tumefaciens promotes tumor induction by modulating pathogen defense in Arabidopsis thaliana. Plant Cell. 21 (9), 2948-2962 (2009).
- Reymond, P., Weber, H., Damond, M., Farmer, E. E. Differential gene expression in response to mechanical wounding and insect feeding in Arabidopsis. Plant Cell. 12 (5), 707-720 (2000).
- Matthysse, A. G. Attachment of Agrobacterium to plant surfaces. Front. Plant Sci. 5, 252 (2014).
- Alatorre-Cobos, F., et al. An improved, low-cost, hydroponic system for growing Arabidopsis and other plant species under aseptic conditions. BMC Plant Biol. 14 (1), 69 (2014).
- Conn, S. J., et al. Protocol: Optimising hydroponic growth systems for nutritional and physiological analysis of Arabidopsis thaliana and other plants. Plant Methods. 9 (1), 4 (2013).
- Nathoo, N. . Identification of putative plant defense genes using a novel hydroponic co-cultivation technique for studying plant-pathogen interaction. , (2015).
- Shaner, N. C., Campbell, R. E., Steinbach, P. A., Giepmans, B. N., Palmer, A. E., Tsien, R. Y. Improved monomeric red, orange and yellow fluorescent proteins derived from Discosoma sp. red fluorescent protein. Nat. Biotechnol. 22 (12), 1567-1572 (2004).
- Wise, A. A., Liu, H., Binns, A. N. Nucleic acid extraction from Agrobacterium strains. Methods Mol. Bio. 343, 67-76 (2006).
- Nolan, T., Hands, R. E., Bustin, S. A. Quantification of mRNA using real-time RT-PCR. Nat. Protoc. 1 (3), 1559-1582 (2006).
- Salter, M. G., Conlon, H. E. Extraction of plant RNA. Methods Mol. Bio. 362, 309-314 (2007).
- Bao, Y., Wang, S., Yang, X., Li, T., Xia, Y., Meng, X. Metabolomic study of the intervention effects of Shuihonghuazi Formula, a Traditional Chinese Medicinal formulae, on hepatocellular carcinoma (HCC) rats using performance HPLC/ESI-TOF-MS. J. Ethnopharmacol. 198, 468-478 (2017).
- Korves, T. M., Bergelson, J. A developmental response to pathogen infection in Arabidopsis. Plant Physiol. 133 (1), 339-347 (2003).
- Lyons, R., Rusu, A., Stiller, J., Powell, J., Manners, J. M., Kazan, K. Investigating the association between flowering time and defense in the Arabidopsis thaliana-Fusarium oxysporum interaction. PLoS ONE. 10 (6), e0127699 (2015).
- Badri, D. V., Weir, T. L., vander Lelie, D., Vivanco, J. M. Rhizosphere chemical dialogues: Plant-microbe interactions. Curr. Opin. Biotechnol. 20 (6), 642-650 (2009).
- Baerson, S. R., et al. Detoxification and transcriptome response in Arabidopsis seedlings exposed to the allelochemical benzoxazolin-2(3H)-one. J. Biol. Chem. 280 (23), 21867-21881 (2005).

