새로운 천연 제품을 발견하는 도구로 질량 분광법 유도 게놈 채굴
Summary
질량 분광법 유도 게놈 마이닝 프로토콜이 확립되고 여기에 설명되어 있습니다. 그것은 게놈 순서 정보 및 LC-MS/MS 분석을 기반으로 하고 복잡한 미생물 및 식물 추출물에서 분자의 식별을 용이하게 하는 것을 목표로 합니다.
Abstract
천연 제품으로 덮인 화학 공간은 엄청나고 널리 인식되지 않습니다. 따라서, 자연에서 그 기능및 잠재적인 인간 이익(예를 들면, 약물 발견 신청을 위한)의 광범위한 평가를 수행하는 편리한 방법론이 요구된다. 이 프로토콜은 게놈 채굴(GM)과 분자 네트워킹(MN)의 조합을 설명하며, 전체 게놈 시퀀싱에서 유전자 클러스터 인코딩 주석과 조잡한 대사 추출물의 화학 구조 시그니처와 일치하는 두 가지 현대적 접근법을 설명합니다. 이것은 새로운 자연 개체의 발견을 향한 첫 번째 단계입니다. 이러한 개념은 함께 적용될 때 MS 유도 게놈 마이닝으로 정의됩니다. 이 방법에서, 주요 성분은 이전에 지정 (MN을 사용하여), 구조적으로 관련된 새로운 후보는 게놈 서열 주석 (GM을 사용하여)와 연관된다. GM과 MN을 결합하는 것은 이미 알려진 화합물에서 유사체를 식별하기 위해 새로운 분자 백본을 대상으로하거나 신진 대사 프로파일을 수확하는 수익성있는 전략입니다.
Introduction
이차 물질 대사의 조사는 수시로 활성 분획에 속하는 성분의 정제, 식별 및 특성에 선행된 특정 생물학 활동에 대한 조추출물을 검열하는 것으로 구성됩니다. 이 과정은 여러 화학 물질의 격리를 촉진, 효율적인 것으로 입증되었습니다. 그러나 요즘에는 재발견비율이 높기 때문에 실현 불가능한 것으로 보입니다. 제약 산업이 전문 대사 산물의 역할과 기능에 대한 지식없이 혁명을 일으켰기 때문에 자연을 정확하게 나타내지 않은 실험실 조건에서 식별이 수행되었습니다1. 오늘날, 자연 신호 영향의 더 나은 이해가있다, 분비, 그리고 탐지 낮은 농도에서 대부분의 대상의 존재. 또한, 프로세스의 규제는 학계 와 제약 산업이 지식을 활용하는 데 도움이 될 것입니다. 그것은 또한 침묵 하는 생 합성 유전자 클러스터와 관련 된 대사 산물의 직접 적인 격리를 포함 하는 연구 혜택을 것입니다 (BGCs)2.
이러한 맥락에서, 게놈 염기서열 분석의 발전은 미생물 대사 산물 선별에 대한 관심을 새롭게 하고 있다. 이는 발견되지 않은 생합성 클러스터의 게놈 정보를 분석하면 실험실 조건하에서 관찰또는 생성되지 않은 새로운 화합물을 코딩하는 유전자가 밝혀지므로. 많은 미생물 전체 게놈 프로젝트 또는 초안은 오늘 유효하고, 수는 게놈광업을통해 새로운 생리 활성 분자를 밝히기위한 거대한 전망을 제공, 매년 증가하고있다3,4.
생합성 유전자 클러스터의 아틀라스는 공동 게놈 연구소 (JGI IMG-ABC)의 통합 미생물 게놈 플랫폼의 구성 요소로 자동으로 채굴 된 유전자 클러스터의 현재 가장 큰 컬렉션입니다2. 가장 최근에는 생합성 유전자 클러스터(MIBiG) 표준화 이니셔티브에 대한 최소 정보로 BGC의 수동 재분석이 촉진되어 고도로 선별된 기준 데이터 집합5를제공합니다. 요즘, 많은 도구는 유전 데이터의 계산 마이닝 및 알려진된 보조 대사 산물에 그들의 연결을 사용 하 여 사용할 수 있습니다. 다른 전략은 또한 새로운 생리 활성 천연 제품에 액세스하기 위해 개발되었습니다 (즉, 이종 발현, 대상 유전자 삭제, 체외 재구성, 게놈 서열, 동위원소 유도 스크리닝 [genomisotopic 접근], 지역 및 글로벌 규제 기관의 조작, 저항 표적 기반 광업, 문화 독립적 인 광업, 그리고, 최근에는 MS-유도 / 코드 접근,9, 법 2,8 ,,88,, 8 , 8 ,8, 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 , 8 10,11,1212,13,14,15).
단수 전략으로 게놈 채굴은 분자의 단일 또는 작은 그룹에 추가하는 노력이 필요합니다; 따라서, 프로세스의 간격은 새로운 화합물이 격리 및 구조 해명에 대한 우선 순위를 지정하는 남아있다. 원칙적으로, 이러한 접근법은 실험당 하나의 생합성 경로만을 표적으로 하여 발견 속도가 느려집니다. 이러한 의미에서, 분자 네트워킹 접근법과 함께 GM을 사용하는 것은 천연물 연구에 대한 중요한 진보를 나타낸다14,,15.
액체 크로마토그래피 질량 분석법(LC-MS)의 다기능성, 정확도 및 고감도는 화합물 식별을 위한 좋은 방법입니다. 현재, 여러 플랫폼은 비 표적 대사체학16,,17,,18,19,,20에대한 알고리즘및소프트웨어 제품군을 투자했다. 이러한 프로그램의 핵심에는 피처 감지(피크 피킹)21 및 피크 정렬이 포함되어 있어 샘플 배치에서 동일한 피처를 일치시키고 패턴을 검색할 수 있습니다. MS 패턴 기반 알고리즘22,,23은 특징적인 단편화 패턴을 비교하고 구조적 특징을 공유하는 분자 패밀리를 생성하는 MS2 유사성을 일치시다. 이러한 특징은 강조되고 군집화될 수 있으며, MS2,,24,,25에의한 복잡한 생물학적 추출물로부터 알려진 및 알려지지 않은 분자를 빠르게 발견할 수 있는 능력을 부여한다. 따라서, 탠덤 MS는 다량의 데이터에 동시에 포함된 여러 화학유형의 구조적 정보를 동시에 얻을 수 있는 다목적 방법이다.
글로벌 천연물 사회 분자 네트워킹(GNPS)26 알고리즘은 정규화된 단편 이온 강도를 사용하여 코신 함수를 사용하여 유사성을 비교하는 다차원 벡터를 구성합니다. 서로 다른 상위 이온 간의 관계는 각 조각화가 노드(원)로 시각화되고 각 노드의 유사성이 모서리(선)로 정의되는 다이어그램 표현으로 플롯됩니다. 단일 소스에서 분자의 글로벌 시각화는 분자 네트워크로 정의됩니다. 구조적으로 서로 다른 분자는 고유하게 단편화하는 분자가 그들의 자신의 특정 클러스터 또는 별자리를 형성하는 반면, 관련 분자는 함께 클러스터됩니다. 군집 화학 형은 생합성 기원에 유사한 구조적 특징의 가상 연결을 할 수 있습니다.
BGCs와 그들의 소분자 제품 사이 생물 정보학 링크를 만들 때 화학형-유전자형 및 유전자형-화학형 접근을 둘 다 결합하는 것은 강력합니다27. 따라서 MS 유도 게놈 채굴은 신속한 방법과 낮은 물질 소비 전략이며, 다양한 대사 및 환경 조건 하에서 하나 이상의 균주의 WGS에 의해 밝혀진 모이온 및 생합성 경로를 연결하는 데 도움이 됩니다.
이 프로토콜의 워크플로우(도1)는AntiSMASH,28,29,30과같은 생합성 유전자 클러스터 분석 플랫폼에 WGS 데이터를 공급하는 것으로 구성됩니다., 그것은 게놈에 의해 인코딩된 화합물 및 종류 화합물의 다양성을 추정하는 것을 돕습니다. 관심 있는 화학적 개체를 코딩하는 생합성 유전자 클러스터를 표적으로 하는 전략이 채택되어야 하며, BGC를 함유하는 야생형 균주 및/또는 이종 균주에서 배양 추출물을 분석하여 GNPS26,,31을이용하여 유사성에 기초하여 클러스터된 이온을 생성할 수 있다. 따라서, 표적 BGC와 연관되고 데이터베이스에서 사용할 수 없는 새로운 분자를 식별할 수 있습니다(주로 알려지지 않은 유사체, 때로는 낮은 적규기에서 생성). 사용자가 이러한 플랫폼에 기여할 수 있고 생물 정보학 및 MS/MS 데이터의 가용성이 급속히 증가하고 있으며, 분자와 복잡한 추출물의 효율적인 연결을 안내하는 효과적인 계산 도구 및 알고리즘의 지속적인 개발 및 업그레이드가 가능해지고 있다는 점을 고려하는 것이 관련이 있습니다.
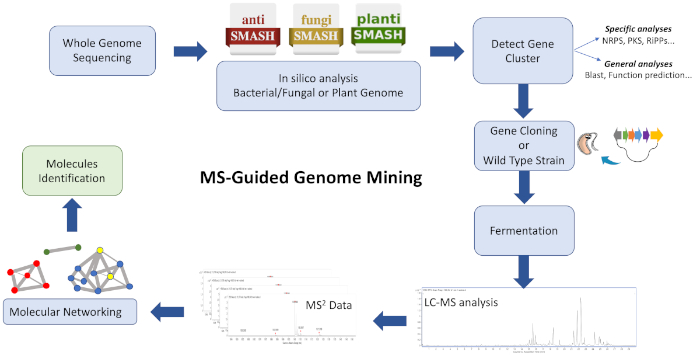
그림 1: 전체 워크플로에 대한 개요입니다. 도시된 것은 새로운 대사산물을 식별하기 위해 기재된 MS 유도 게놈 채굴 접근법에 관여하는 생물정보학, 복제 및 분자 네트워킹 단계의 예시이다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
이 프로토콜은 게놈 채굴과 분자 네트워킹을 천연 제품 발견 파이프라인의 시작점으로 결합하는 신속하고 효율적인 워크플로우를 설명합니다. 많은 응용 프로그램이 하나의 네트워크에서 MS 검출 가능한 분자의 조성 및 관련성을 시각화할 수 있지만, 구조적으로 유사한 클러스터된 분자를 시각화하기 위해 여러 가지가 여기에 채택되었습니다. 이 전략을 사용 하 여, 연쇄상 구균 sp. CBMAI 2042의 대사 추출물에서 관찰 된 새로운 사이클로데펩티드 제품 성공적으로 확인. 게놈 채굴에 의해 유도되는, valinomycins를 위한 전체 생합성 유전자 클러스터 인코딩은 생산자 균주 연쇄상 구균 Coelicolor M1146으로 인식되고 복제됩니다. 마지막으로, MS 패턴 기지를 둔 분자 네트워킹에 따라, MS에 의해 검출된 분자는 그들의 생물발생32를책임지는 BGCs와 상관됩니다.
Protocol
Representative Results
Discussion
이 프로토콜의 가장 큰 장점은 새로운 분자, 특히 구조적 유사체2의구조를 해명하기 위해 MS 데이터와 함께 대사 프로파일및 브리지 게놈 정보를 신속하게 복제하는 능력입니다. 게놈 정보에 기초하여, 다른 천연물 화학형은 폴리케타이드 (PK), 비리보소몰 펩티드 (NRP), 글리코실화 천연물 (GNP)뿐만 아니라 비밀BGC. Metabolomic 스크리닝은 실험실 조건 하에서 특정 균주에 의해 생성된 …
Disclosures
The authors have nothing to disclose.
Acknowledgements
이 연구에 대한 재정 지원은 상파울루 연구 재단에 의해 제공되었다 – FAPESP (2019/10564-5, 2014/12727-5 및 2014/50249-8~ L.G.O; 2013/12598-8 및 2015/01013-4~ R.S.; 및 2019/08853-9- C.F.F.A. B.S.P, C.F.F.A., 및 L.G.O.는 국가 과학 기술 개발위원회 – CNPq (205729/2018-5, 162191/2015-4, 및 313492/2017-4)로부터 펠로우십을 받았습니다. L.G.O.는 또한 과학의 여성을 위한 프로그램(2008, 브라질 판)에서 제공하는 보조금 지원에 감사드립니다. 모든 저자는 CAPES (고등 교육 인력의 개선을위한 조정)가 브라질에서 졸업 후 프로그램을 지원하기위한 인정합니다.
Materials
| Acetonitrile | Tedia | AA1120-048 | HPLC grade |
| Agar | Oxoid | LP0011 | NA |
| Apramycin | Sigma Aldrich | A2024 | NA |
| Carbenicillin | Sigma Aldrich | C9231 | NA |
| Centrifuge | Eppendorf | NA | 5804 |
| Chloramphenicol | Sigma Aldrich | C3175 | NA |
| Column C18 | Agilent Technologies | NA | ZORBAX RRHD Extend-C18, 80Å, 2.1 x 50 mm, 1.8 µm, 1200 bar pressure limit P/N 757700-902 |
| Kanamycin | Sigma Aldrich | K1377 | NA |
| Manitol P.A.- A.C.S. | Synth | NA | NA |
| Microcentrifuge | Eppendorf | NA | 5418 |
| Nalidixic acid | Sigma Aldrich | N4382 | NA |
| Phusion Flash High-Fidelity PCR Master Mix | ThermoFisher Scientific | F548S | NA |
| Q-TOF mass spectrometer | Agilent technologies | NA | 6550 iFunnel Q-TOF LC/MS |
| Sacarose P.A.- A.C.S. | Synth | NA | NA |
| Shaker/Incubator | Marconi | MA420 | NA |
| Sodium Chloride | Synth | NA | P. A. – ACS |
| Soy extract | NA | NA | NA |
| Sucrose | Synth | NA | P. A. – ACS |
| Thermal Cycles | Eppendorf | NA | Mastercycler Nexus Gradient |
| Thiostrepton | Sigma Aldrich | T8902 | NA |
| Tryptone | Oxoid | LP0042 | NA |
| Tryptone Soy Broth | Oxoid | CM0129 | NA |
| UPLC | Agilent Technologies | NA | 1290 Infinity LC System |
| Yeast extract | Oxoid | LP0021 | NA |
References
- Davies, J. Specialized microbial metabolites: functions and origins. The Journal of Antibiotics. 66 (7), 361-364 (2013).
- Ziemert, N., Alanjary, M., Weber, T. The evolution of genome mining in microbes – a review. Natural Product Reports. 33 (8), 988-1005 (2016).
- Zerikly, M., Challis, G. L. Strategies for the Discovery of New Natural Products by Genome Mining. ChemBioChem. 10 (4), 625-633 (2009).
- Gomez-Escribano, J. P., Bibb, M. J. Heterologous expression of natural product biosynthetic gene clusters in Streptomyces coelicolor: from genome mining to manipulation of biosynthetic pathways. Journal of Industrial Microbiology & Biotechnology. 41 (2), 425-431 (2014).
- Medema, M. H., et al. Minimum Information about a Biosynthetic Gene cluster. Nature Chemical Biology. 11 (9), 625-631 (2015).
- Lautru, S., Deeth, R. J., Bailey, L. M., Challis, G. L. Discovery of a new peptide natural product by Streptomyces coelicolor genome mining. Nature Chemical Biology. 1 (5), 265-269 (2005).
- Chiang, Y. -. M., et al. Molecular Genetic Mining of the Aspergillus Secondary Metabolome: Discovery of the Emericellamide Biosynthetic Pathway. Chemistry & Biology. 15 (6), 527-532 (2008).
- Huang, T., et al. Identification and Characterization of the Pyridomycin Biosynthetic Gene Cluster of Streptomyces pyridomyceticus NRRL B-2517. Journal of Biological Chemistry. 286 (23), 20648-20657 (2011).
- Udwary, D. W., et al. Genome sequencing reveals complex secondary metabolome in the marine actinomycete Salinispora tropica. Proceedings of the National Academy of Sciences. 104 (25), 10376-10381 (2007).
- Gross, H., et al. The Genomisotopic Approach: A Systematic Method to Isolate Products of Orphan Biosynthetic Gene Clusters. Chemistry & Biology. 14 (1), 53-63 (2007).
- Spohn, M., Wohlleben, W., Stegmann, E. Elucidation of the zinc-dependent regulation in Amycolatopsis japonicum enabled the identification of the ethylenediamine-disuccinate ([S,S ]-EDDS) genes. Environmental Microbiology. 18 (4), 1249-1263 (2016).
- Thaker, M. N., Waglechner, N., Wright, G. D. Antibiotic resistance-mediated isolation of scaffold-specific natural product producers. Nature Protocols. 9 (6), 1469-1479 (2014).
- Katz, M., Hover, B. M., Brady, S. F. Culture-independent discovery of natural products from soil metagenomes. Journal of Industrial Microbiology & Biotechnology. 43, 129-141 (2016).
- Quinn, R. A., et al. Molecular Networking as a Drug Discovery, Drug Metabolism, and Precision Medicine Strategy. Trends in Pharmacological Sciences. 38 (2), 143-154 (2017).
- Yang, J. Y., et al. Molecular Networking as a Dereplication Strategy. Journal of Natural Products. 76 (9), 1686-1699 (2013).
- Lommen, A. MetAlign: Interface-Driven, Versatile Metabolomics Tool for Hyphenated Full-Scan Mass Spectrometry Data Preprocessing. Analytical Chemistry. 81 (8), 3079-3086 (2009).
- Katajamaa, M., Miettinen, J., Oresic, M. MZmine: toolbox for processing and visualization of mass spectrometry based molecular profile data. Bioinformatics. 22 (5), 634-636 (2006).
- Pluskal, T., Castillo, S., Villar-Briones, A., Orešič, M. MZmine 2: Modular framework for processing, visualizing, and analyzing mass spectrometry-based molecular profile data. BMC Bioinformatics. 11 (1), 395 (2010).
- Tautenhahn, R., Patti, G. J., Rinehart, D., Siuzdak, G. XCMS Online: A Web-Based Platform to Process Untargeted Metabolomic Data. Analytical Chemistry. 84 (11), 5035-5039 (2012).
- Kuhl, C., Tautenhahn, R., Böttcher, C., Larson, T. R., Neumann, S. CAMERA: An Integrated Strategy for Compound Spectra Extraction and Annotation of Liquid Chromatography/Mass Spectrometry Data Sets. Analytical Chemistry. 84 (1), 283-289 (2012).
- Katajamaa, M., Orešič, M. Data processing for mass spectrometry-based metabolomics. Journal of Chromatography A. 1158, 318-328 (2007).
- Liu, W. -. T., et al. Interpretation of Tandem Mass Spectra Obtained from Cyclic Nonribosomal Peptides. Analytical Chemistry. 81 (11), 4200-4209 (2009).
- Ng, J., et al. Dereplication and de novo sequencing of nonribosomal peptides. Nature Methods. 6 (8), 596-599 (2009).
- Liaw, C., et al. Vitroprocines, new antibiotics against Acinetobacter baumannii, discovered from marine Vibrio sp. QWI-06 using mass-spectrometry-based metabolomics approach. Scientific Reports. 5 (1), 1-11 (2015).
- Kang, K. B., et al. Targeted Isolation of Neuroprotective Dicoumaroyl Neolignans and Lignans from Sageretia theezans Using in Silico Molecular Network Annotation Propagation-Based Dereplication. Journal of Natural Products. 81 (8), 1819-1828 (2018).
- Wang, M., et al. Sharing and community curation of mass spectrometry data with Global Natural Products Social Molecular Networking. Nature Biotechnology. 34 (8), 828-837 (2016).
- Doroghazi, J. R., et al. A roadmap for natural product discovery based on large-scale genomics and metabolomics. Nature Chemical Biology. 10 (11), 963-968 (2014).
- Medema, M. H., et al. antiSMASH: rapid identification, annotation and analysis of secondary metabolite biosynthesis gene clusters in bacterial and fungal genome sequences. Nucleic Acids Research. 39, 339-346 (2011).
- Weber, T., et al. antiSMASH 3.0-a comprehensive resource for the genome mining of biosynthetic gene clusters. Nucleic Acids Research. 43, 237-243 (2015).
- Blin, K., et al. antiSMASH 5.0: updates to the secondary metabolite genome mining pipeline. Nucleic Acids Research. 47, 81-87 (2019).
- Watrous, J., et al. Mass spectral molecular networking of living microbial colonies. Proceedings of the National Academy of Sciences. 109 (26), 1743-1752 (2012).
- Paulo, B. S., Sigrist, R., Angolini, C. F. F., De Oliveira, L. G. New Cyclodepsipeptide Derivatives Revealed by Genome Mining and Molecular Networking. ChemistrySelect. 4 (27), 7785-7790 (2019).
- Gonzaga de Oliveira, L., Sigrist, R., Sachetto Paulo, B., Samborskyy, M. Whole-Genome Sequence of the Endophytic Streptomyces sp. Strain CBMAI 2042, Isolated from Citrus sinensis. Microbiology Resource Announcements. 8 (2), 1-2 (2019).
- Aziz, R. K., et al. The RAST Server: Rapid Annotations using Subsystems Technology. BMC Genomics. 9 (1), 75 (2008).
- Nah, H. -. J., Pyeon, H. -. R., Kang, S. -. H., Choi, S. -. S., Kim, E. -. S. Cloning and Heterologous Expression of a Large-sized Natural Product Biosynthetic Gene Cluster in Streptomyces Species. Frontiers in Microbiology. 8, 1-10 (2017).
- Zhang, J. J., Tang, X., Moore, B. S. Genetic platforms for heterologous expression of microbial natural products. Natural Product Reports. 36 (9), 1313-1332 (2019).
- Alduina, R., et al. Artificial chromosome libraries of Streptomyces coelicolor A3(2) and Planobispora rosea. FEMS Microbiology Letters. 218 (1), 181-186 (2003).
- Jones, A. C., et al. Phage P1-Derived Artificial Chromosomes Facilitate Heterologous Expression of the FK506 Gene Cluster. PLoS One. 8 (7), 69319 (2013).
- Gomez-Escribano, J. P., Bibb, M. J. Engineering Streptomyces coelicolor for heterologous expression of secondary metabolite gene clusters. Microbial Biotechnology. 4 (2), 207-215 (2011).
- Cannell, R. J. P. . Natural Products Isolation. , (1998).
- Kersten, R. D., et al. A mass spectrometry-guided genome mining approach for natural product peptidogenomics. Nature Chemical Biology. 7 (11), 794-802 (2011).
- Kersten, R. D., et al. Glycogenomics as a mass spectrometry-guided genome-mining method for microbial glycosylated molecules. Proceedings of the National Academy of Sciences. 110 (47), 4407-4416 (2013).
- Liu, W., et al. MS/MS-based networking and peptidogenomics guided genome mining revealed the stenothricin gene cluster in Streptomyces roseosporus. The Journal of Antibiotics. 67 (1), 99-104 (2014).
- Duncan, K. R., et al. Molecular Networking and Pattern-Based Genome Mining Improves Discovery of Biosynthetic Gene Clusters and their Products from Salinispora Species. Chemistry & Biology. 22 (4), 460-471 (2015).
- Cao, L., et al. MetaMiner: A Scalable Peptidogenomics Approach for Discovery of Ribosomal Peptide Natural Products with Blind Modifications from Microbial Communities. Cell Systems. , (2019).
- Chen, L. -. Y., Cui, H. -. T., Su, C., Bai, F. -. W., Zhao, X. -. Q. Analysis of the complete genome sequence of a marine-derived strain Streptomyces sp. S063 CGMCC 14582 reveals its biosynthetic potential to produce novel anti-complement agents and peptides. PeerJ. 7 (1), 6122 (2019).
- Kim Tiam, S., et al. Insights into the Diversity of Secondary Metabolites of Planktothrix Using a Biphasic Approach Combining Global Genomics and Metabolomics. Toxins. 11 (9), 498 (2019).
- Özakin, S., Ince, E. Genome and metabolome mining of marine obligate Salinispora strains to discover new natural products. Turkish Journal of Biology. 43 (1), 28-36 (2019).
- Trivella, D. B. B., de Felicio, R. The Tripod for Bacterial Natural Product Discovery: Genome Mining, Silent Pathway Induction, and Mass Spectrometry-Based Molecular Networking. mSystems. 3 (2), 00160 (2018).
- Maansson, M., et al. An Integrated Metabolomic and Genomic Mining Workflow To Uncover the Biosynthetic Potential of Bacteria. mSystems. 1 (3), 1-14 (2016).
- Blin, K., Kim, H. U., Medema, M. H., Weber, T. Recent development of antiSMASH and other computational approaches to mine secondary metabolite biosynthetic gene clusters. Briefings in Bioinformatics. 20 (4), 1103-1113 (2019).
- Fisch, K. M. Biosynthesis of natural products by microbial iterative hybrid PKS-NRPS. RSC Advances. 3 (40), 18228-18247 (2013).
- Tatsuno, S., Arakawa, K., Kinashi, H. Analysis of Modular-iterative Mixed Biosynthesis of Lankacidin by Heterologous Expression and Gene Fusion. The Journal of Antibiotics. 60 (11), 700-708 (2007).
- Helfrich, E. J. N., Piel, J. Biosynthesis of polyketides by trans-AT polyketide synthases. Natural Product Reports. 33 (2), 231-316 (2016).

