Massespektrometri-guidet genom minedrift som et redskab til at afdække nye naturlige produkter
Summary
En massespektrometri-guidet genom minedrift protokol er etableret og beskrevet her. Den er baseret på oplysninger om genomsekvenser og LC-MS/MS-analyse og har til formål at lette identifikationen af molekyler fra komplekse mikrobielle ekstrakter og planteekstrakter.
Abstract
Det kemiske rum, der er omfattet af naturlige produkter, er enormt og almindeligt ukendt. Derfor er praktiske metoder til at udføre vidtrækkende evaluering af deres funktioner i naturen og potentielle menneskelige fordele (f.eks for narkotika opdagelse applikationer) ønskes. Denne protokol beskriver kombinationen af genomminedrift (GM) og molekylære netværk (MN), to nutidige tilgange, der matcher genklyngekodede anmærkninger i hele genomsekvensering med kemiske struktursignaturer fra rå metaboliske ekstrakter. Dette er det første skridt i retning af opdagelsen af nye naturlige enheder. Disse begreber, når de anvendes sammen, er defineret her som MS-guidet genom minedrift. I denne metode er de vigtigste komponenter tidligere udpeget (ved hjælp af MN), og strukturelt relaterede nye kandidater er forbundet med genom sekvens anmærkninger (ved hjælp af GM). Kombinere GM og MN er en rentabel strategi for at målrette nye molekyle backbones eller høste metaboliske profiler med henblik på at identificere analoger fra allerede kendte forbindelser.
Introduction
Undersøgelser af sekundær metabolisme består ofte af screening af rå ekstrakter til specifikke biologiske aktiviteter efterfulgt af rensning, identifikation og karakterisering af bestanddele, der tilhører aktive fraktioner. Denne proces har vist sig at være effektiv og har fremmet isolationen af flere kemiske enheder. Men i dag er dette ses som umuligt, primært på grund af de høje satser for genopdagelse. Som den farmaceutiske industri revolutionerede uden kendskab til roller og funktioner specialiserede metabolitter, deres identifikation blev udført under laboratorieforhold, der ikke præcist repræsenterer naturen1. I dag er der en bedre forståelse af naturlige signalering påvirkninger, sekresion, og tilstedeværelsen af de fleste mål på målbart lave koncentrationer. Derudover vil regulering af processen hjælpe det akademiske samfund og medicinalindustrien til at drage fordel af denne viden. Det vil også gavne forskning, der indebærer direkte isolering af metabolitter i forbindelse med tavse biosyntetiske genklynger (BGC)2.
I den forbindelse har fremskridt inden for genomisk sekventering fornyet interesse for screening af mikroorganismers metabolitter. Dette skyldes, at analysere genomiske oplysninger om udækkede biosyntetiske klynger kan afsløre gener, der koder nye forbindelser, der ikke observeres eller produceres under laboratorieforhold. Mange mikrobielle hele genom projekter eller udkast er tilgængelige i dag, og antallet vokser hvert år, hvilket giver massive udsigter til at afdække nye bioaktive molekyler gennem genom minedrift3,4.
Atlasset over biosyntetiske genklynger er den nuværende største samling af automatisk udvindede genklynger som en del af den integrerede mikrobielle genomplatform under Joint Genome Institute (JGI IMG-ABC)2. Senest har minimuminformationen for biosyntetiske genklynger (MIBiG) Standardiseringsinitiativ fremmet manuel reannotation af bgcs, hvilket giver et meget kurateret referencedatasæt5. I dag, masser af værktøjer er tilgængelige for at muliggøre beregningsmæssige minedrift af genetiske data og deres forbindelse til kendte sekundære metabolitter. Der er også udviklet forskellige strategier for at få adgang til nye bioaktive naturprodukter (dvs. heterologt udtryk, målgensletning, in vitro-rekonstitution, genomisk sekvens, isotopstyret screening [genomisotoptilgang], manipulation af lokale og globale tilsynsmyndigheder, resistensmålbaseret minedrift, kulturuafhængig minedrift og senest MS-styrede/kodetilgange22,6,7,8,9, 10,11,12,13,14,15).
Genomminedrift som en enestående strategi kræver en indsats for at anmærke en enkelt eller lille gruppe molekyler; dermed er der stadig huller i processen, hvor nye forbindelser prioriteres til isolation og strukturbelysning. I princippet er disse tilgange kun rettet mod én biosyntetisk vej pr. eksperiment, hvilket resulterer i en langsom opdagelseshastighed. I denne forstand er brugen af GM sammen med en molekylær netværkstilgang et vigtigt fremskridt for forskning i naturlige produkter14,15.
Alsidigheden, nøjagtigheden og den høje følsomhed af flydende kromatografi-massespektrometri (LC-MS) gør det til en god metode til sammentæding identifikation. I øjeblikket har flere platforme investeret algoritmer og software suiter for ikke-målrettede metabolomics16,17,18,19,20. Kernen i disse programmer omfatter funktionsregistrering (peak picking)21 og peak alignment, som giver mulighed for match af identiske funktioner på tværs af en batch af prøver og søge efter mønstre. MS mønster-baserede algoritmer22,23 sammenligne karakteristiske fragmentering mønstre og matche MS2 ligheder genererer molekylære familier deler strukturelle funktioner. Disse funktioner kan derefter fremhæves og grupperes, hvilket giver mulighed for hurtigt at opdage kendte og ukendte molekyler fra en kompleks biologisk ekstrakt ved tandem MS2,24,25. Tandem MS er derfor en alsidig metode til at få strukturelle oplysninger om flere kemotyper, der er indeholdt i en stor mængde data samtidigt.
Global Natural Products Social Molecular Networking (BNPS)26 algoritme bruger den normaliserede fragment ioner intensitet til at konstruere flerdimensionale vektorer, hvor ligheder sammenlignes ved hjælp af en cosinus funktion. Forholdet mellem forskellige overordnede ioner afbildes i en diagramrepræsentation, hvor hver fragmentering visualiseres som en node (cirkler), og hver nodes sammenktening defineres af en kant (linjer). Den globale visualisering af molekyler fra en enkelt kilde defineres som et molekylært netværk. Strukturelt divergerende molekyler, der fragmenterer entydigt vil danne deres egen specifikke klynge eller konstellation, mens beslægtede molekyler klynge sammen. Klyngedannelse kemotyper tillader den hypotetiske tilslutning af lignende strukturelle træk til deres biosyntetiske oprindelse.
Kombinationen af både kemotype-til-genotype og genotype-til-kemotype tilgange er kraftfuld, når man skaber bioinformatik forbindelser mellem BGCs og deres små molekyle produkter27. Derfor ms-guidede genom minedrift er en hurtig metode og lav materiale-forbrugende strategi, og det hjælper bro forældre ioner og biosyntetiske veje afsløret af WGS af en eller flere stammer under forskellige metaboliske og miljømæssige forhold.
Arbejdsprocessen i denne protokol (figur 1) består i at fodre WGS-data i en biosyntetisk genklyngeanmærkningsplatform som antiSMASH28,29,30. Det hjælper med at vurdere de mange forskellige forbindelser og klasse af forbindelser kodet af genomet. Der skal vedtages en strategi for at målrette indsatsen mod en biosyntetisk genklynge , der koder en kemisk enhed af interesse , og dyrkningsekstrakter fra en vild typestamme og/eller heterolog stamme , der indeholder BGC , kan analyseres for at generere klyngebaserede ioner baseret på ligheder ved hjælp af GNPS26,31. Det er derfor muligt at identificere nye molekyler, der forbinder med den målrettede BGC, og som ikke er tilgængelige i databasen (hovedsagelig ukendte analoger, undertiden fremstillet i lave titers). Det er relevant at antage, at brugerne kan bidrage til disse platforme, og at tilgængeligheden af bioinformatik og MS/MS-data stiger hurtigt, hvilket fører til en konstant udvikling og opgradering af effektive beregningsværktøjer og algoritmer til at styre effektive forbindelser af komplekse ekstrakter med molekyler.
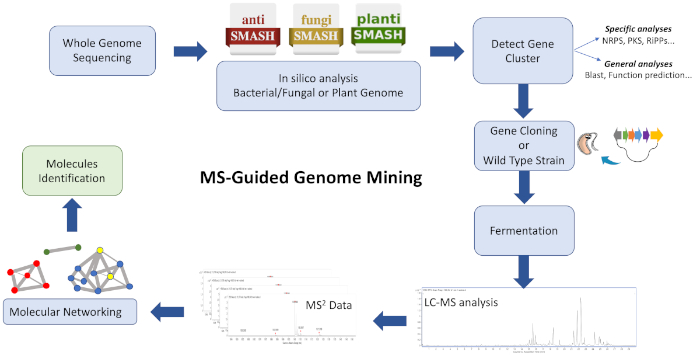
Figur 1: Oversigt over hele arbejdsprocessen. Vist er en illustration af bioinformatik, kloning, og molekylære netværkstrin involveret i den beskrevne MS-guidede genom minedrift tilgang til at identificere nye metabolitter. Klik her for at se en større version af dette tal.
Denne protokol beskriver en hurtig og effektiv arbejdsgang til at kombinere genomminedrift og molekylære netværk som udgangspunkt for den naturlige produktopdagelsespipeline. Selv om mange applikationer er i stand til at visualisere sammensætningen og relatedness af MS-påviselige molekyler i et netværk, flere er vedtaget her for at visualisere strukturelt lignende grupperede molekyler. Ved hjælp af denne strategi, nye cyclodepsipeptid produkter observeret i metaboliske ekstrakter af Streptomyces sp. CBMAI 2042 er med succes identificeret. Styret af genomminedrift, hele biosyntetiske gen klynge kodning for valinomyciner er anerkendt og klonet ind i producenten stamme Streptomyces coelicolor M1146. Endelig, efter en MS mønster-baserede molekylære netværk, molekyler opdaget af MS er korreleret med BGCs ansvarlig for deres biogenesis32.
Protocol
Representative Results
Discussion
Den største fordel ved denne protokol er dens evne til hurtigt at dereplikere metaboliske profiler og bro genomiske oplysninger med MS data med henblik på at belyse strukturerne i nye molekyler, især strukturelle analoger2. På grundlag af genomiske oplysninger kan forskellige naturlige produkters kemotyper undersøges, såsom polyketid (PK), nonribosomale peptider (NRP) og glycosylated natural products (BNP) samt kryptiske BGCs. Metabolomisk screening giver beviser for aktiverede BGC-profiler …
Disclosures
The authors have nothing to disclose.
Acknowledgements
Den finansielle støtte til denne undersøgelse blev ydet af São Paulo Research Foundation – FAPESP (2019/10564-5, 2014/12727-5 og 2014/50249-8 til L.G.O; 2013/12598-8 og 2015/01013-4 til R.S.; og 2019/08853-9 til C.F.F.A). B.S.P, C.F.F.A., og L.G.O. modtog stipendier fra Det Nationale Råd for Videnskabelig og Teknologisk Udvikling – CNPq (205729/2018-5, 162191/2015-4 og 313492/2017-4). L.G.O. er også taknemmelig for tilskuddet fra programmet For Women in Science (2008, Brasilianske Edition). Alle forfattere anerkender CAPES (Koordinering for forbedring af de videregående uddannelser personale) for at støtte post-graduering programmer i Brasilien.
Materials
| Acetonitrile | Tedia | AA1120-048 | HPLC grade |
| Agar | Oxoid | LP0011 | NA |
| Apramycin | Sigma Aldrich | A2024 | NA |
| Carbenicillin | Sigma Aldrich | C9231 | NA |
| Centrifuge | Eppendorf | NA | 5804 |
| Chloramphenicol | Sigma Aldrich | C3175 | NA |
| Column C18 | Agilent Technologies | NA | ZORBAX RRHD Extend-C18, 80Å, 2.1 x 50 mm, 1.8 µm, 1200 bar pressure limit P/N 757700-902 |
| Kanamycin | Sigma Aldrich | K1377 | NA |
| Manitol P.A.- A.C.S. | Synth | NA | NA |
| Microcentrifuge | Eppendorf | NA | 5418 |
| Nalidixic acid | Sigma Aldrich | N4382 | NA |
| Phusion Flash High-Fidelity PCR Master Mix | ThermoFisher Scientific | F548S | NA |
| Q-TOF mass spectrometer | Agilent technologies | NA | 6550 iFunnel Q-TOF LC/MS |
| Sacarose P.A.- A.C.S. | Synth | NA | NA |
| Shaker/Incubator | Marconi | MA420 | NA |
| Sodium Chloride | Synth | NA | P. A. – ACS |
| Soy extract | NA | NA | NA |
| Sucrose | Synth | NA | P. A. – ACS |
| Thermal Cycles | Eppendorf | NA | Mastercycler Nexus Gradient |
| Thiostrepton | Sigma Aldrich | T8902 | NA |
| Tryptone | Oxoid | LP0042 | NA |
| Tryptone Soy Broth | Oxoid | CM0129 | NA |
| UPLC | Agilent Technologies | NA | 1290 Infinity LC System |
| Yeast extract | Oxoid | LP0021 | NA |
References
- Davies, J. Specialized microbial metabolites: functions and origins. The Journal of Antibiotics. 66 (7), 361-364 (2013).
- Ziemert, N., Alanjary, M., Weber, T. The evolution of genome mining in microbes – a review. Natural Product Reports. 33 (8), 988-1005 (2016).
- Zerikly, M., Challis, G. L. Strategies for the Discovery of New Natural Products by Genome Mining. ChemBioChem. 10 (4), 625-633 (2009).
- Gomez-Escribano, J. P., Bibb, M. J. Heterologous expression of natural product biosynthetic gene clusters in Streptomyces coelicolor: from genome mining to manipulation of biosynthetic pathways. Journal of Industrial Microbiology & Biotechnology. 41 (2), 425-431 (2014).
- Medema, M. H., et al. Minimum Information about a Biosynthetic Gene cluster. Nature Chemical Biology. 11 (9), 625-631 (2015).
- Lautru, S., Deeth, R. J., Bailey, L. M., Challis, G. L. Discovery of a new peptide natural product by Streptomyces coelicolor genome mining. Nature Chemical Biology. 1 (5), 265-269 (2005).
- Chiang, Y. -. M., et al. Molecular Genetic Mining of the Aspergillus Secondary Metabolome: Discovery of the Emericellamide Biosynthetic Pathway. Chemistry & Biology. 15 (6), 527-532 (2008).
- Huang, T., et al. Identification and Characterization of the Pyridomycin Biosynthetic Gene Cluster of Streptomyces pyridomyceticus NRRL B-2517. Journal of Biological Chemistry. 286 (23), 20648-20657 (2011).
- Udwary, D. W., et al. Genome sequencing reveals complex secondary metabolome in the marine actinomycete Salinispora tropica. Proceedings of the National Academy of Sciences. 104 (25), 10376-10381 (2007).
- Gross, H., et al. The Genomisotopic Approach: A Systematic Method to Isolate Products of Orphan Biosynthetic Gene Clusters. Chemistry & Biology. 14 (1), 53-63 (2007).
- Spohn, M., Wohlleben, W., Stegmann, E. Elucidation of the zinc-dependent regulation in Amycolatopsis japonicum enabled the identification of the ethylenediamine-disuccinate ([S,S ]-EDDS) genes. Environmental Microbiology. 18 (4), 1249-1263 (2016).
- Thaker, M. N., Waglechner, N., Wright, G. D. Antibiotic resistance-mediated isolation of scaffold-specific natural product producers. Nature Protocols. 9 (6), 1469-1479 (2014).
- Katz, M., Hover, B. M., Brady, S. F. Culture-independent discovery of natural products from soil metagenomes. Journal of Industrial Microbiology & Biotechnology. 43, 129-141 (2016).
- Quinn, R. A., et al. Molecular Networking as a Drug Discovery, Drug Metabolism, and Precision Medicine Strategy. Trends in Pharmacological Sciences. 38 (2), 143-154 (2017).
- Yang, J. Y., et al. Molecular Networking as a Dereplication Strategy. Journal of Natural Products. 76 (9), 1686-1699 (2013).
- Lommen, A. MetAlign: Interface-Driven, Versatile Metabolomics Tool for Hyphenated Full-Scan Mass Spectrometry Data Preprocessing. Analytical Chemistry. 81 (8), 3079-3086 (2009).
- Katajamaa, M., Miettinen, J., Oresic, M. MZmine: toolbox for processing and visualization of mass spectrometry based molecular profile data. Bioinformatics. 22 (5), 634-636 (2006).
- Pluskal, T., Castillo, S., Villar-Briones, A., Orešič, M. MZmine 2: Modular framework for processing, visualizing, and analyzing mass spectrometry-based molecular profile data. BMC Bioinformatics. 11 (1), 395 (2010).
- Tautenhahn, R., Patti, G. J., Rinehart, D., Siuzdak, G. XCMS Online: A Web-Based Platform to Process Untargeted Metabolomic Data. Analytical Chemistry. 84 (11), 5035-5039 (2012).
- Kuhl, C., Tautenhahn, R., Böttcher, C., Larson, T. R., Neumann, S. CAMERA: An Integrated Strategy for Compound Spectra Extraction and Annotation of Liquid Chromatography/Mass Spectrometry Data Sets. Analytical Chemistry. 84 (1), 283-289 (2012).
- Katajamaa, M., Orešič, M. Data processing for mass spectrometry-based metabolomics. Journal of Chromatography A. 1158, 318-328 (2007).
- Liu, W. -. T., et al. Interpretation of Tandem Mass Spectra Obtained from Cyclic Nonribosomal Peptides. Analytical Chemistry. 81 (11), 4200-4209 (2009).
- Ng, J., et al. Dereplication and de novo sequencing of nonribosomal peptides. Nature Methods. 6 (8), 596-599 (2009).
- Liaw, C., et al. Vitroprocines, new antibiotics against Acinetobacter baumannii, discovered from marine Vibrio sp. QWI-06 using mass-spectrometry-based metabolomics approach. Scientific Reports. 5 (1), 1-11 (2015).
- Kang, K. B., et al. Targeted Isolation of Neuroprotective Dicoumaroyl Neolignans and Lignans from Sageretia theezans Using in Silico Molecular Network Annotation Propagation-Based Dereplication. Journal of Natural Products. 81 (8), 1819-1828 (2018).
- Wang, M., et al. Sharing and community curation of mass spectrometry data with Global Natural Products Social Molecular Networking. Nature Biotechnology. 34 (8), 828-837 (2016).
- Doroghazi, J. R., et al. A roadmap for natural product discovery based on large-scale genomics and metabolomics. Nature Chemical Biology. 10 (11), 963-968 (2014).
- Medema, M. H., et al. antiSMASH: rapid identification, annotation and analysis of secondary metabolite biosynthesis gene clusters in bacterial and fungal genome sequences. Nucleic Acids Research. 39, 339-346 (2011).
- Weber, T., et al. antiSMASH 3.0-a comprehensive resource for the genome mining of biosynthetic gene clusters. Nucleic Acids Research. 43, 237-243 (2015).
- Blin, K., et al. antiSMASH 5.0: updates to the secondary metabolite genome mining pipeline. Nucleic Acids Research. 47, 81-87 (2019).
- Watrous, J., et al. Mass spectral molecular networking of living microbial colonies. Proceedings of the National Academy of Sciences. 109 (26), 1743-1752 (2012).
- Paulo, B. S., Sigrist, R., Angolini, C. F. F., De Oliveira, L. G. New Cyclodepsipeptide Derivatives Revealed by Genome Mining and Molecular Networking. ChemistrySelect. 4 (27), 7785-7790 (2019).
- Gonzaga de Oliveira, L., Sigrist, R., Sachetto Paulo, B., Samborskyy, M. Whole-Genome Sequence of the Endophytic Streptomyces sp. Strain CBMAI 2042, Isolated from Citrus sinensis. Microbiology Resource Announcements. 8 (2), 1-2 (2019).
- Aziz, R. K., et al. The RAST Server: Rapid Annotations using Subsystems Technology. BMC Genomics. 9 (1), 75 (2008).
- Nah, H. -. J., Pyeon, H. -. R., Kang, S. -. H., Choi, S. -. S., Kim, E. -. S. Cloning and Heterologous Expression of a Large-sized Natural Product Biosynthetic Gene Cluster in Streptomyces Species. Frontiers in Microbiology. 8, 1-10 (2017).
- Zhang, J. J., Tang, X., Moore, B. S. Genetic platforms for heterologous expression of microbial natural products. Natural Product Reports. 36 (9), 1313-1332 (2019).
- Alduina, R., et al. Artificial chromosome libraries of Streptomyces coelicolor A3(2) and Planobispora rosea. FEMS Microbiology Letters. 218 (1), 181-186 (2003).
- Jones, A. C., et al. Phage P1-Derived Artificial Chromosomes Facilitate Heterologous Expression of the FK506 Gene Cluster. PLoS One. 8 (7), 69319 (2013).
- Gomez-Escribano, J. P., Bibb, M. J. Engineering Streptomyces coelicolor for heterologous expression of secondary metabolite gene clusters. Microbial Biotechnology. 4 (2), 207-215 (2011).
- Cannell, R. J. P. . Natural Products Isolation. , (1998).
- Kersten, R. D., et al. A mass spectrometry-guided genome mining approach for natural product peptidogenomics. Nature Chemical Biology. 7 (11), 794-802 (2011).
- Kersten, R. D., et al. Glycogenomics as a mass spectrometry-guided genome-mining method for microbial glycosylated molecules. Proceedings of the National Academy of Sciences. 110 (47), 4407-4416 (2013).
- Liu, W., et al. MS/MS-based networking and peptidogenomics guided genome mining revealed the stenothricin gene cluster in Streptomyces roseosporus. The Journal of Antibiotics. 67 (1), 99-104 (2014).
- Duncan, K. R., et al. Molecular Networking and Pattern-Based Genome Mining Improves Discovery of Biosynthetic Gene Clusters and their Products from Salinispora Species. Chemistry & Biology. 22 (4), 460-471 (2015).
- Cao, L., et al. MetaMiner: A Scalable Peptidogenomics Approach for Discovery of Ribosomal Peptide Natural Products with Blind Modifications from Microbial Communities. Cell Systems. , (2019).
- Chen, L. -. Y., Cui, H. -. T., Su, C., Bai, F. -. W., Zhao, X. -. Q. Analysis of the complete genome sequence of a marine-derived strain Streptomyces sp. S063 CGMCC 14582 reveals its biosynthetic potential to produce novel anti-complement agents and peptides. PeerJ. 7 (1), 6122 (2019).
- Kim Tiam, S., et al. Insights into the Diversity of Secondary Metabolites of Planktothrix Using a Biphasic Approach Combining Global Genomics and Metabolomics. Toxins. 11 (9), 498 (2019).
- Özakin, S., Ince, E. Genome and metabolome mining of marine obligate Salinispora strains to discover new natural products. Turkish Journal of Biology. 43 (1), 28-36 (2019).
- Trivella, D. B. B., de Felicio, R. The Tripod for Bacterial Natural Product Discovery: Genome Mining, Silent Pathway Induction, and Mass Spectrometry-Based Molecular Networking. mSystems. 3 (2), 00160 (2018).
- Maansson, M., et al. An Integrated Metabolomic and Genomic Mining Workflow To Uncover the Biosynthetic Potential of Bacteria. mSystems. 1 (3), 1-14 (2016).
- Blin, K., Kim, H. U., Medema, M. H., Weber, T. Recent development of antiSMASH and other computational approaches to mine secondary metabolite biosynthetic gene clusters. Briefings in Bioinformatics. 20 (4), 1103-1113 (2019).
- Fisch, K. M. Biosynthesis of natural products by microbial iterative hybrid PKS-NRPS. RSC Advances. 3 (40), 18228-18247 (2013).
- Tatsuno, S., Arakawa, K., Kinashi, H. Analysis of Modular-iterative Mixed Biosynthesis of Lankacidin by Heterologous Expression and Gene Fusion. The Journal of Antibiotics. 60 (11), 700-708 (2007).
- Helfrich, E. J. N., Piel, J. Biosynthesis of polyketides by trans-AT polyketide synthases. Natural Product Reports. 33 (2), 231-316 (2016).

