基于CRISPR-Cas系统和抗CRISPR蛋白的基因数字电路
Summary
CRISPR-Cas系统和抗CRISPR蛋白被整合到 酿酒酵母的布尔门方案中。新的小逻辑电路表现出良好的性能,加深了对基于dCas9/dCas12a的转录因子和抗CRISPR蛋白性质的理解。
Abstract
合成基因布尔门和数字电路具有广泛的应用,从医疗诊断到环境保护。CRISPR-Cas系统及其天然抑制剂——抗CRISPR蛋白(Acrs)的发现为设计和实施 体内 基因数字电路提供了一种新工具。在这里,我们描述了一种协议,该协议遵循“设计-构建-测试-学习”生物工程循环的思想,并利用dCas9 / dCas12a及其相应的Acrs来建立小型转录网络,其中一些行为类似于 酿酒酵母中的布尔门。这些结果指出了dCas9/dCas12a作为转录因子的性质。特别是,为了实现基因表达的最大激活,dSpCas9需要与收集VP64激活域(AD)多个拷贝的工程支架RNA相互作用。相比之下,dCas12a应在C末端与强VP64-p65-RTA (VPR) AD融合。此外,两种Cas蛋白的活性不会通过增加细胞中sgRNA / crRNA的量来增强。本文还解释了如何基于CRISPR-dCas-Acr相互作用构建布尔门。人雌激素受体的 AcrIIA4 融合激素结合结构域是对 β-雌二醇有反应的 NOT 门的核心,而由诱导性 GAL1 启动子合成的 AcrVA 允许以半乳糖作为输入来模拟 YES 和 NOT 门。在后一个电路中,AcrVA5与dLbCas12a一起表现出最佳的逻辑行为。
Introduction
2011年,研究人员提出了一种计算方法,并开发了相应的软件,用于数字合成基因电路的自动设计1。用户必须指定输入数量(三个或四个)并填写电路真值表;这提供了使用电子技术推导电路结构的所有必要信息。真值表通过 Karnaugh 映射方法2 转换为两个布尔公式。每个布尔公式都由描述电路输入(部分)之间的逻辑运算(和或乘法)及其否定(文字)的子句组成。子句依次相加(OR)或乘法(AND)以计算电路输出。每个电路都可以根据其两个相应的公式中的任何一个来实现:一个以POS(总和乘积)形式编写,另一个以SOP(产品总和)表示形式编写。前者由包含文字逻辑和的子句(即布尔门)的乘法组成。相比之下,后者是文字相乘的子句的总和。
电路可以在面包板上通过将不同的门物理连接在一起来实现。电流允许门之间交换信号,从而计算输出。
在生物学中,情况更为复杂。布尔门可以实现为转录单元(TU;即真核细胞内的序列“启动子编码区域终止子”),其中转录或翻译(或两者)受到调节。因此,至少有两种分子建立了生物线路:转录因子蛋白和非编码、反义RNA。
基因数字电路被组织成两层或三层门,即:1)输入层,由YES(缓冲器)和NOT门组成,并将输入化学物质转换为布线分子;2) 内层,它由与相应布尔公式中的子句一样多的 TU 组成。如果电路按照SOP公式设计,则内层中的每个子句都将在所谓的分布式输出架构中产生电路输出(例如荧光)。如果使用和积(POS)公式,则需要3)最后一层,它将包含一个从内层收集布线分子的乘法门。
总体而言,在合成生物学中,可以为同一电路设计许多不同的方案。它们在TU和连接分子的数量和种类上有所不同。为了选择在酵母细胞中实现的最简单解决方案,每个电路设计都与复杂性评分S相关联,定义为
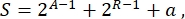
其中 A 代表激活剂的数量, R 代表阻遏剂的数量, a 是反义RNA分子的数量。如果电路中没有激活剂或抑制因子,则它们对S的贡献为零。因此,当需要大量正交转录因子时,在实验室中实现电路方案(高S)更加困难。这意味着新的活化器和抑制器应 重新 设计,以实现数字电路内部的完整布线。原则上,可以使用锌指蛋白3 和TAL效应子4 作为模板来组装新型DNA结合蛋白。但是,此选项似乎过于艰巨和耗时;因此,人们应该主要依靠小RNA和翻译调控来完成复杂的基因回路。
最初,这种方法是为了在细菌中制造数字电路而开发的。事实上,在真核细胞中,比起反义RNA,更适合谈论microRNA(miRNA)或小干扰RNA(siRNA)5。然而,RNAi途径不存在于 酿酒酵母中。因此,应该选择完全转录网络。假设一个电路需要五个激活器和五个抑制器;其复杂性分数为 S = 32。通过将10个转录因子替换为融合到激活域(AD)的单个dCas96 (核酸酶缺陷型Cas9),可以降低电路复杂性。如7所示,当在TATA盒和TSS(转录起始位点)之间结合启动子时,dCas9-AD在酵母中充当阻遏剂,当在TATA盒上游结合时,作为激活剂起作用。因此,可以用单个dCas9-AD融合蛋白和10个sgRNA(单个向导RNA)替换10个转录因子,总复杂性得分为S = 11。合成十种sgRNA既快速又容易,而如前所述,组装10种蛋白质需要更长、更复杂的工作。
或者,可以使用两种正交的dCas蛋白(例如dCas9和dCas12a):一种融合到AD,另一种裸露或与抑制结构域组合。复杂度分数只会增加一个单位 (S = 12)。因此,CRISPR-dCas系统是 酿酒酵母中构建非常复杂的基因数字电路的关键。
本文深入表征了酵母中基于dCas9和dCas12a的阻遏剂和激活剂的效率。结果表明,它们不需要大量的sgRNA来优化其活性,因此优先避免使用游离体质粒。此外,当使用募集VP64 AD拷贝的支架RNA(scRNA)时,基于dCas9的激活剂更有效。相比之下,dCas12a在直接融合到强VPR AD时效果很好。此外,根据激活剂的配置,合成活化启动子需要可变数量的靶位点(例如,使用 dCas12a-VPR 时为 3 个,dCas9-VP64 为 6 个,dCas9 和 scRNA 时只有一个)。作为阻遏因子,dCas12a在结合编码区而不是启动子时显得更敏锐。
然而,作为一个缺点,CRISPR-dCas9 / dCas12a不直接与化学物质相互作用。因此,它们在输入图层中可能没有用处。出于这个原因,已经研究了含有抗CRISPR蛋白(Acrs)的替代布尔门设计。Acrs作用于(d)Cas蛋白并抑制其工作8。因此,它们是调节CRISPR-(d)Cas系统活性的一种手段。本文深入分析了酿酒酵母II型Acrs与(d)Cas9以及V型Acrs和(d)Cas12a之间的相互作用。由于Acrs比Cas蛋白小得多,因此通过将人雌激素受体9-HBD(hER)的激素结合结构域融合到AcrIIA4,构建了对雌激素β-雌二醇有反应的NOT门。此外,在用半乳糖诱导时实现了少数表达dCas12a(-AD)的YES和NOT门和AcrVAs。目前,这些门仅用作概念证明。然而,它们也代表了深入重新思考算法的第一步,以执行酵母细胞中合成基因数字电路的计算自动设计。
Protocol
Representative Results
Discussion
该协议显示了合成基因数字电路可能的完整工作流程,遵循“设计-构建-测试-学习”(DBTL)生物工程周期,涉及干实验室和湿实验室实验。在这里,我们专注于CRISPR-Cas系统,主要是dSpCas9,denAsCas12a,dLbCas12a和相应的抗CRISPR蛋白,通过在 酿酒酵母 中设计和构建小转录网络。其中一些模仿布尔门,布尔门是数字电路的基本组件。这里描述的所有回路都允许我们描述酿 酒酵母中CRISPR相?…
Disclosures
The authors have nothing to disclose.
Acknowledgements
我们要感谢TJU合成生物学实验室-SPST的所有学生的帮助,以及李智和张向阳在FACS实验中的帮助。
Materials
| 0.1 mL PCR 8-strip tubes | NEST | 403112 | |
| 0.2 mL PCR tubes | Axygen | PCR-02-C | |
| 1.5 mL Microtubes | Axygen | MCT-150-C | |
| 15 mL Centrifuge tubes | BIOFIL | CFT011150 | |
| 2 mL Microtubes | Axygen | MCT-200-C | |
| 50 mL Centrifuge tubes | BIOFIL | CFT011500 | |
| Agarose-molecular biology grade | Invitrogen | 75510-019 | |
| Ampicillin sodium salt | Solarbio | 69-52-3 | |
| Applied biosystems veriti 96-well thermal cycler | Thermo Fisher Scientific | 4375786 | |
| AxyPrep DNA gel extraction kit | Axygen | AP-GX-250 | |
| BD FACSuite CS&T research beads | BD | 650621 | Fluorescent beads |
| BD FACSVerse flow cytometer | BD | – | |
| Centrifuge | Eppendorf | 5424 | |
| Centrifuge Sorvall ST 16R | Thermo Fisher Scientific | 75004380 | |
| E. coli competent cells (Strain DH5α) | Life Technologies | 18263-012 | |
| ECL select Western Blotting detection reagent | GE Healthcare | RPN2235 | |
| Electrophoresis apparatus | Beijing JUNYI Electrophoresis Co., Ltd | JY300C | |
| Flat 8-strip caps | NEST | 406012 | |
| Gene synthesis company | Azenta Life Sciences | https://web.azenta.com/zh-cn/azenta-life-sciences | |
| Goat anti-Mouse IgG (H+L) cross-adsorbed secondary antibody Alexa Fluor 568 | Invitrogen | A-11004 | |
| HiFiScript cDNA synthesis kit | CWBIO | CW2569M | Kit used in step 6.2.2.1 |
| Lysate solution (Zymolyase) | zymoresearch | E1004-A | |
| Nikon Eclipse 80i fluorescence microscope | Nikon | – | Fluorescence microscope |
| Pipet tips—10 μL | Axygen | T-300-R-S | |
| Pipet tips—1000 μL | Axygen | T-1000-B-R-S | |
| Pipet tips—200 μL | Axygen | T-200-Y-R-S | |
| pRSII403 | Addgene | 35436 | |
| pRSII404 | Addgene | 35438 | |
| pRSII405 | Addgene | 35440 | |
| pRSII406 | Addgene | 35442 | |
| pRSII424 | Addgene | 35466 | |
| pTPGI_dSpCas9_VP64 | Addgene | 49013 | |
| Q5 High-fidelity DNApolymerase | New England Biolabs | M0491 | |
| Restriction enzyme-Acc65I | New England Biolabs | R0599 | |
| Restriction enzyme-BamHI | New England Biolabs | R0136 | |
| Restriction enzyme-SacI-HF | New England Biolabs | R3156 | |
| Restriction enzyme-XhoI | New England Biolabs | R0146 | |
| Roche LightCycler 96 | Roche | – | Real-Time PCR Instrument |
| S. cerevisiae CEN.PK2-1C | – | – | The parent strain. The genotype is: MATa; his3D1; leu2-3_112; ura3-52; trp1-289; MAL2-8c; SUC2 |
| Stem-Loop Kit | SparkJade | AG0502 | Kit used in step 6.2.1.3 |
| T100 Thermal Cycler | BIO-RAD | 186-1096 | |
| T4 DNA ligase | New England Biolabs | M0202 | |
| T5 Exonuclease | New England Biolabs | M0363 | |
| Taq DNA ligase | New England Biolabs | M0208 | |
| Taq DNA polymerase | New England Biolabs | M0495 | |
| TB Green Premix Ex Taq II (Tli RNaseH Plus)(2x) (SYBR Green I dye) | Takara | RR820Q | |
| YeaStar RNA kit | Zymo Research | R1002 | |
| β-estradiol | Sigma-Aldrich | E8875 |
References
- Marchisio, M. A., Stelling, J. Automatic design of digital synthetic gene circuits. PLOS Computational Biology. 7 (2), 1001083 (2011).
- Karnaugh, M. The map method for synthesis of combinational logic circuits. Transactions of the American Institute of Electrical Engineers. 72 (9), 593-599 (1953).
- Mandell, J. G., Barbas, C. F. Zinc finger tools: custom DNA-binding domains for transcription factors and nucleases. Nucleic Acids Research. 34, 516-523 (2006).
- Bogdanove, A. J., Voytas, D. F. TAL effectors: customizable proteins for DNA targeting. Science. 333 (6051), 1843-1846 (2011).
- Drinnenberg, I. A., et al. RNAi in budding yeast. Science. 326 (5952), 544-550 (2009).
- Gander, M. W., Vrana, J. D., Voje, W. E., Carothers, J. M., Klavins, E. Digital logic circuits in yeast with CRISPR-dCas9 NOR gates. Nature Communications. 8, 15459 (2017).
- Farzadfard, F., Perli, S. D., Lu, T. K. Tunable and multifunctional eukaryotic transcription factors based on CRISPR/Cas. ACS Synthetic Biology. 2 (10), 604-613 (2013).
- Nakamura, M., et al. Anti-CRISPR-mediated control of gene editing and synthetic circuits in eukaryotic cells. Nature Communications. 10 (1), 194 (2019).
- Louvion, J. F., Havaux-Copf, B., Picard, D. Fusion of GAL4-VP16 to a steroid-binding domain provides a tool for gratuitous induction of galactose-responsive genes in yeast. Gene. 131 (1), 129-134 (1993).
- DiCarlo, J. E., et al. Genome engineering in Saccharomyces cerevisiae using CRISPR-Cas systems. Nucleic Acids Research. 41 (7), 4336-4343 (2013).
- Gao, Y., Zhao, Y. Self-processing of ribozyme-flanked RNAs into guide RNAs in vitro and in vivo for CRISPR-mediated genome editing. Journal of Integrative Plant Biology. 56 (4), 343-349 (2014).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Zetsche, B., et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell. 163 (3), 759-771 (2015).
- Fonfara, I., Richter, H., Bratovic, M., Le Rhun, A., Charpentier, E. The CRISPR-associated DNA-cleaving enzyme Cpf1 also processes precursor CRISPR RNA. Nature. 532 (7600), 517-521 (2016).
- Yu, L., Marchisio, M. A. Saccharomyces cerevisiae synthetic transcriptional networks harnessing dCas12a and Type V-A anti-CRISPR proteins. ACS Synthetic Biology. 10 (4), 870-883 (2021).
- Zhang, Y., Marchisio, M. A. Interaction of bare dSpCas9, scaffold gRNA, and type II anti-CRISPR proteins highly favors the control of gene expression in the yeast S. cerevisiae. ACS Synthetic Biology. 11 (1), 176-190 (2022).
- Sheff, M. A., Thorn, K. S. Optimized cassettes for fluorescent protein tagging in Saccharomyces cerevisiae. Yeast. 21 (8), 661-670 (2004).
- Naito, Y., Hino, K., Bono, H., Ui-Tei, K. CRISPRdirect: software for designing CRISPR/Cas guide RNA with reduced off-target sites. Bioinformatics. 31 (7), 1120-1123 (2015).
- Chee, M. K., Haase, S. B. New and redesigned pRS plasmid shuttle vectors for genetic manipulation of Saccharomyces cerevisiae. G3: Genes|Genomes|Genetics. 2 (5), 515-526 (2012).
- Gibson, D. G. Synthesis of DNA fragments in yeast by one-step assembly of overlapping oligonucleotides. Nucleic Acids Research. 37 (20), 6984-6990 (2009).
- Froger, A., Hall, J. E. Transformation of plasmid DNA into E. coli using the heat shock method. Journal of Visualized Experiments. (6), e253 (2007).
- Green, M. R., Sambrook, J. . Molecular Cloning. Fourth edition. , (2012).
- Sanger, F. Determination of nucleotide sequences in DNA. Science. 214 (4526), 1205-1210 (1981).
- Gietz, R. D., Woods, R. A. Transformation of yeast by lithium acetate/single-stranded carrier DNA/polyethylene glycol method. Methods in Enzymology. 350, 87-96 (2002).
- Zalatan, J. G., et al. Engineering complex synthetic transcriptional programs with CRISPR RNA scaffolds. Cell. 160 (1-2), 339-350 (2015).
- Song, W., Li, J., Liang, Q., Marchisio, M. A. Can terminators be used as insulators into yeast synthetic gene circuits. Journal of Biological Engineering. 10, 19 (2016).
- Rauch, B. J., et al. Inhibition of CRISPR-Cas9 with bacteriophage proteins. Cell. 168 (1-2), 150-158 (2017).
- Hynes, A. P., et al. An anti-CRISPR from a virulent streptococcal phage inhibits Streptococcus pyogenes Cas9. Nature Microbiology. 2 (10), 1374-1380 (2017).
- Watters, K. E., Fellmann, C., Bai, H. B., Ren, S. M., Doudna, J. A. Systematic discovery of natural CRISPR-Cas12a inhibitors. Science. 362 (6411), 236-239 (2018).
- Chen, C., et al. Real-time quantification of microRNAs by stem-loop RT-PCR. Nucleic Acids Research. 33 (20), 179 (2005).
- Pfaffl, M. W. A new mathematical model for relative quantification in real-time RT-PCR. Nucleic Acids Research. 29 (9), 45 (2001).
- Hahne, F., et al. flowCore: a Bioconductor package for high throughput flow cytometry. BMC Bioinformatics. 10 (1), 106 (2009).
- Li, J., Xu, Z., Chupalov, A., Marchisio, M. A. Anti-CRISPR-based biosensors in the yeast S. cerevisiae. Journal of Biological Engineering. 12, 11 (2018).
- Dong, L., et al. An anti-CRISPR protein disables type V Cas12a by acetylation. Nature Structural & Molecular Biology. 26 (4), 308-314 (2019).

