CRISPR-Casシステムと抗CRISPRタンパク質に基づく遺伝子デジタル回路
Summary
CRISPR-Cas系および抗CRISPRタンパク質は、 出芽酵母のブールゲートのスキームに統合されました。新しい小型論理回路は良好な性能を示し、dCas9/dCas12aベースの転写因子と抗CRISPRタンパク質の特性の両方の理解を深めました。
Abstract
合成遺伝子ブールゲートとデジタル回路は、医療診断から環境ケアまで、幅広い用途があります。CRISPR-Casシステムとその天然阻害剤である抗CRISPRタンパク質(Acrs)の発見は、 in vivo 遺伝子デジタル回路を設計および実装するための新しいツールを提供します。ここでは、「設計-構築-テスト-学習」生物工学サイクルの考え方に従い、dCas9 / dCas12aとそれに対応するAcrを利用して、 サッカロミセス・セレビシエでブールゲートのように振る舞う小さな転写ネットワークを確立するプロトコルについて説明します。これらの結果は、転写因子としてのdCas9/dCas12aの性質を指摘しています。特に、遺伝子発現の最大活性化を達成するために、dSpCas9はVP64活性化ドメイン(AD)の複数のコピーを収集する操作された足場RNAと相互作用する必要があります。対照的に、dCas12aはC末端で強力なVP64-p65-Rta(VPR)ADと融合する必要があります。さらに、両方のCasタンパク質の活性は、細胞内のsgRNA/crRNAの量を増加させることによって増強されない。この記事では、CRISPR-dCas-Acr の相互作用に基づいてブール型ゲートを構築する方法についても説明します。ヒトエストロゲン受容体のAcrIIA4融合ホルモン結合ドメインは、β-エストラジオールに応答するNOTゲートのコアですが、誘導性 GAL1 プロモーターによって合成されたAcrVAは、ガラクトースを入力としてYESゲートとNOTゲートの両方を模倣することができます。後者の回路では、AcrVA5とdLbCas12aが最良のロジック動作を示しました。
Introduction
2011年、研究者は計算方法を提案し、デジタル合成遺伝子回路の自動設計に対応するソフトウェアを開発しました1。ユーザーは入力数(3つまたは4つ)を指定し、回路真理値表に記入する必要がありました。これにより、電子機器の技術を使用して回路構造を導き出すために必要なすべての情報が提供されました。真理値表は、Karnaughマップ法2を介して2つのブール式に変換されました。各ブール式は、回路入力(の一部)とその否定(リテラル)間の論理演算(合計または乗算)を記述する句で構成されています。句は、順番に合計(OR)または乗算(AND)されて回路出力を計算します。すべての回路は、POS(合計の積)形式で記述された式とSOP(製品の合計)表現で記述された2つの対応する式のいずれかに従って実現できます。前者は、リテラルの論理和を含む句(つまり、ブールゲート)の乗算で構成されます。対照的に、後者は、リテラルが乗算される句の合計です。
電気回路は、ブレッドボード上で、異なるゲートを物理的に配線することによって実現できます。電流はゲート間の信号交換を可能にし、出力の計算につながります。
生物学では、状況はより複雑です。ブールゲートは、転写または翻訳(またはその両方)が調節される転写単位(TU、すなわち、真核細胞内の配列「プロモーター-コード領域-ターミネーター」)として実現することができる。したがって、少なくとも2種類の分子、すなわち転写因子タンパク質および非コードアンチセンスRNA1の生物学的配線を確立する。
遺伝子デジタル回路は、2層または3層のゲート、すなわち1)YES(バッファー)およびNOTゲートで構成された入力層で構成され、入力化学物質を配線分子に変換する。2)対応するブール式に句があるのと同じ数のTUで構成される内部層。回路がSOP式に従って設計されている場合、内部層のすべての句は、いわゆる分散出力アーキテクチャで回路出力(蛍光など)を生成します。和の積(POS)式が使用される場合、3)最終層が必要であり、これには内部層から配線分子を収集する単一の乗法ゲートが含まれます。
全体として、合成生物学では、同じ回路に対して多くの異なるスキームを設計することができます。それらはTUと配線分子の両方の数と種類が異なります。酵母細胞に実装する最も簡単なソリューションを選択するために、各回路設計は、次のように定義される複雑さスコアSに関連付けられています。
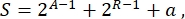
ここで、 A は活性化剤の数を表し、 R はリプレッサーの数を表し、 a はアンチセンスRNA分子の量です。アクティベータまたはリプレッサーのいずれかが回路にない場合、Sへの寄与はゼロです。そのため、ラボでの回路スキーム(高S)の実現は、多数の直交転写因子を必要とする場合により困難である。これは、デジタル回路内の完全な配線を実現するために、新しいアクティベータとリプレッサーを de novo で設計する必要があることを意味します。原理的には、ジンクフィンガータンパク質3 およびTALエフェクター4 を鋳型として使用することにより、新規DNA結合タンパク質を組み立てることができる。ただし、このオプションは非常に困難で時間がかかるように見えます。したがって、複雑な遺伝子回路を完成させるために、主に小さなRNAと翻訳調節に頼る必要があります。
もともと、この方法は細菌のデジタル回路を作製するために開発されました。実際、真核細胞では、アンチセンスRNAの代わりに、マイクロRNA(miRNA)または低分子干渉RNA(siRNA)について話す方が適しています5。しかしながら、RNAi経路は酵母 S.セレビシエには存在しない。したがって、完全に転写ネットワークを選択する必要があります。回路に5つのアクティベータと5つのリプレッサーが必要であるとします。その複雑さのスコアはS = 32になります。10個の転写因子を活性化ドメイン(AD)に融合した単一のdCas96 (ヌクレアーゼ欠損Cas9)に置き換えることで、回路の複雑さを軽減することができます。7に示すように、dCas9-ADは、TATAボックスとTSS(転写開始部位)の間でプロモーターを結合させる際の酵母のリプレッサーとして、またTATAボックスのかなり上流に結合するときの活性化剤として働きます。したがって、10個の転写因子を単一のdCas9-AD融合タンパク質と10個のsgRNA(シングルガイドRNA)に置き換えることができ、合計複雑さスコアはS = 11になります。10個のsgRNAを合成するのは迅速かつ簡単ですが、前述のように、10個のタンパク質の組み立てにははるかに長く複雑な作業が必要になります。
あるいは、2つの直交するdCasタンパク質(例えば、dCas9およびdCas12a)を使用することができ、1つはADに融合し、もう1つは裸または抑制ドメインと組み合わせて使用する。複雑さのスコアは 1 単位だけ増加します (S = 12)。したがって、CRISPR-dCasシステムは、 出芽酵母における非常に複雑な遺伝子デジタル回路の構築の鍵となります。
この論文は、酵母におけるdCas9およびdCas12aベースのリプレッサーおよびアクチベーターの両方の効率を深く特徴付けています。結果は、それらがそれらの活性を最適化するために大量のsgRNAを必要としないことを示したので、エピソームプラスミドは優先的に避けられる。さらに、dCas9ベースのアクチベーターは、VP64 ADのコピーをリクルートするスキャフォールドRNA(scRNA)を使用する場合にはるかに効果的です。対照的に、dCas12aは、強力なVPR ADに直接融合するとうまく機能します。さらに、合成活性化プロモーターは、活性化因子の構成に応じて、可変数の標的部位を必要とします(例えば、dCas12a-VPRを使用する場合は3つ、dCas9-VP64を使用する場合は6つ、dCas9とscRNAを使用する場合は1つだけ)。リプレッサーとして、dCas12aはプロモーターよりもコード領域に結合するとより鋭敏に見えます。
ただし、欠点として、CRISPR-dCas9 / dCas12aは化学物質と直接相互作用しません。したがって、入力層では役に立たない可能性があります。このため、抗CRISPRタンパク質(ACRS)を含む代替ブールゲート設計が検討されています。ACRは(d)Casタンパク質に作用し、その働きを阻害します8.したがって、それらはCRISPR-(d)Casシステムの活性を調節する手段です。この論文では、出芽酵母のII型ACRSと(d)Cas9、およびV型Acrと(d)Cas12aの相互作用を徹底的に分析しています。AcrsはCasタンパク質よりもはるかに小さいため、ヒトエストロゲン受容体9-HBD(hER)-のホルモン結合ドメインをAcrIIA4に融合させることによって、エストロゲンβ-エストラジオールに応答するNOTゲートを構築しました。さらに、dCas12a(-AD)を構成的に発現する少数のYESゲートとNOTゲート、およびガラクトースによる誘導時にAcrVAが実現されました。現在、これらのゲートは概念実証としてのみ機能します。しかし、それらはまた、酵母細胞における合成遺伝子デジタル回路の計算自動設計を実行するためのアルゴリズムの深い再考に向けた第一歩を表しています。
Protocol
Representative Results
Discussion
このプロトコルは、「設計-構築-テスト-学習」(DBTL)生物工学サイクルに従い、ドライラボ実験とウェットラボ実験の両方に関する合成遺伝子デジタル回路の完全なワークフローの可能性を示しました。ここでは、出 芽酵母 の低分子転写ネットワークを設計・構築することにより、主にdSpCas9、denAsCas12a、dLbCas12a、および対応する抗CRISPRタンパク質であるCRISPR-Casシステムに焦点を当てま?…
Disclosures
The authors have nothing to disclose.
Acknowledgements
合成生物学ラボ(SPST、TJU)のすべての学生に、FACS実験に協力してくれたZhi LiとXiangyang Zhangに感謝したいと思います。
Materials
| 0.1 mL PCR 8-strip tubes | NEST | 403112 | |
| 0.2 mL PCR tubes | Axygen | PCR-02-C | |
| 1.5 mL Microtubes | Axygen | MCT-150-C | |
| 15 mL Centrifuge tubes | BIOFIL | CFT011150 | |
| 2 mL Microtubes | Axygen | MCT-200-C | |
| 50 mL Centrifuge tubes | BIOFIL | CFT011500 | |
| Agarose-molecular biology grade | Invitrogen | 75510-019 | |
| Ampicillin sodium salt | Solarbio | 69-52-3 | |
| Applied biosystems veriti 96-well thermal cycler | Thermo Fisher Scientific | 4375786 | |
| AxyPrep DNA gel extraction kit | Axygen | AP-GX-250 | |
| BD FACSuite CS&T research beads | BD | 650621 | Fluorescent beads |
| BD FACSVerse flow cytometer | BD | – | |
| Centrifuge | Eppendorf | 5424 | |
| Centrifuge Sorvall ST 16R | Thermo Fisher Scientific | 75004380 | |
| E. coli competent cells (Strain DH5α) | Life Technologies | 18263-012 | |
| ECL select Western Blotting detection reagent | GE Healthcare | RPN2235 | |
| Electrophoresis apparatus | Beijing JUNYI Electrophoresis Co., Ltd | JY300C | |
| Flat 8-strip caps | NEST | 406012 | |
| Gene synthesis company | Azenta Life Sciences | https://web.azenta.com/zh-cn/azenta-life-sciences | |
| Goat anti-Mouse IgG (H+L) cross-adsorbed secondary antibody Alexa Fluor 568 | Invitrogen | A-11004 | |
| HiFiScript cDNA synthesis kit | CWBIO | CW2569M | Kit used in step 6.2.2.1 |
| Lysate solution (Zymolyase) | zymoresearch | E1004-A | |
| Nikon Eclipse 80i fluorescence microscope | Nikon | – | Fluorescence microscope |
| Pipet tips—10 μL | Axygen | T-300-R-S | |
| Pipet tips—1000 μL | Axygen | T-1000-B-R-S | |
| Pipet tips—200 μL | Axygen | T-200-Y-R-S | |
| pRSII403 | Addgene | 35436 | |
| pRSII404 | Addgene | 35438 | |
| pRSII405 | Addgene | 35440 | |
| pRSII406 | Addgene | 35442 | |
| pRSII424 | Addgene | 35466 | |
| pTPGI_dSpCas9_VP64 | Addgene | 49013 | |
| Q5 High-fidelity DNApolymerase | New England Biolabs | M0491 | |
| Restriction enzyme-Acc65I | New England Biolabs | R0599 | |
| Restriction enzyme-BamHI | New England Biolabs | R0136 | |
| Restriction enzyme-SacI-HF | New England Biolabs | R3156 | |
| Restriction enzyme-XhoI | New England Biolabs | R0146 | |
| Roche LightCycler 96 | Roche | – | Real-Time PCR Instrument |
| S. cerevisiae CEN.PK2-1C | – | – | The parent strain. The genotype is: MATa; his3D1; leu2-3_112; ura3-52; trp1-289; MAL2-8c; SUC2 |
| Stem-Loop Kit | SparkJade | AG0502 | Kit used in step 6.2.1.3 |
| T100 Thermal Cycler | BIO-RAD | 186-1096 | |
| T4 DNA ligase | New England Biolabs | M0202 | |
| T5 Exonuclease | New England Biolabs | M0363 | |
| Taq DNA ligase | New England Biolabs | M0208 | |
| Taq DNA polymerase | New England Biolabs | M0495 | |
| TB Green Premix Ex Taq II (Tli RNaseH Plus)(2x) (SYBR Green I dye) | Takara | RR820Q | |
| YeaStar RNA kit | Zymo Research | R1002 | |
| β-estradiol | Sigma-Aldrich | E8875 |
References
- Marchisio, M. A., Stelling, J. Automatic design of digital synthetic gene circuits. PLOS Computational Biology. 7 (2), 1001083 (2011).
- Karnaugh, M. The map method for synthesis of combinational logic circuits. Transactions of the American Institute of Electrical Engineers. 72 (9), 593-599 (1953).
- Mandell, J. G., Barbas, C. F. Zinc finger tools: custom DNA-binding domains for transcription factors and nucleases. Nucleic Acids Research. 34, 516-523 (2006).
- Bogdanove, A. J., Voytas, D. F. TAL effectors: customizable proteins for DNA targeting. Science. 333 (6051), 1843-1846 (2011).
- Drinnenberg, I. A., et al. RNAi in budding yeast. Science. 326 (5952), 544-550 (2009).
- Gander, M. W., Vrana, J. D., Voje, W. E., Carothers, J. M., Klavins, E. Digital logic circuits in yeast with CRISPR-dCas9 NOR gates. Nature Communications. 8, 15459 (2017).
- Farzadfard, F., Perli, S. D., Lu, T. K. Tunable and multifunctional eukaryotic transcription factors based on CRISPR/Cas. ACS Synthetic Biology. 2 (10), 604-613 (2013).
- Nakamura, M., et al. Anti-CRISPR-mediated control of gene editing and synthetic circuits in eukaryotic cells. Nature Communications. 10 (1), 194 (2019).
- Louvion, J. F., Havaux-Copf, B., Picard, D. Fusion of GAL4-VP16 to a steroid-binding domain provides a tool for gratuitous induction of galactose-responsive genes in yeast. Gene. 131 (1), 129-134 (1993).
- DiCarlo, J. E., et al. Genome engineering in Saccharomyces cerevisiae using CRISPR-Cas systems. Nucleic Acids Research. 41 (7), 4336-4343 (2013).
- Gao, Y., Zhao, Y. Self-processing of ribozyme-flanked RNAs into guide RNAs in vitro and in vivo for CRISPR-mediated genome editing. Journal of Integrative Plant Biology. 56 (4), 343-349 (2014).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Zetsche, B., et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell. 163 (3), 759-771 (2015).
- Fonfara, I., Richter, H., Bratovic, M., Le Rhun, A., Charpentier, E. The CRISPR-associated DNA-cleaving enzyme Cpf1 also processes precursor CRISPR RNA. Nature. 532 (7600), 517-521 (2016).
- Yu, L., Marchisio, M. A. Saccharomyces cerevisiae synthetic transcriptional networks harnessing dCas12a and Type V-A anti-CRISPR proteins. ACS Synthetic Biology. 10 (4), 870-883 (2021).
- Zhang, Y., Marchisio, M. A. Interaction of bare dSpCas9, scaffold gRNA, and type II anti-CRISPR proteins highly favors the control of gene expression in the yeast S. cerevisiae. ACS Synthetic Biology. 11 (1), 176-190 (2022).
- Sheff, M. A., Thorn, K. S. Optimized cassettes for fluorescent protein tagging in Saccharomyces cerevisiae. Yeast. 21 (8), 661-670 (2004).
- Naito, Y., Hino, K., Bono, H., Ui-Tei, K. CRISPRdirect: software for designing CRISPR/Cas guide RNA with reduced off-target sites. Bioinformatics. 31 (7), 1120-1123 (2015).
- Chee, M. K., Haase, S. B. New and redesigned pRS plasmid shuttle vectors for genetic manipulation of Saccharomyces cerevisiae. G3: Genes|Genomes|Genetics. 2 (5), 515-526 (2012).
- Gibson, D. G. Synthesis of DNA fragments in yeast by one-step assembly of overlapping oligonucleotides. Nucleic Acids Research. 37 (20), 6984-6990 (2009).
- Froger, A., Hall, J. E. Transformation of plasmid DNA into E. coli using the heat shock method. Journal of Visualized Experiments. (6), e253 (2007).
- Green, M. R., Sambrook, J. . Molecular Cloning. Fourth edition. , (2012).
- Sanger, F. Determination of nucleotide sequences in DNA. Science. 214 (4526), 1205-1210 (1981).
- Gietz, R. D., Woods, R. A. Transformation of yeast by lithium acetate/single-stranded carrier DNA/polyethylene glycol method. Methods in Enzymology. 350, 87-96 (2002).
- Zalatan, J. G., et al. Engineering complex synthetic transcriptional programs with CRISPR RNA scaffolds. Cell. 160 (1-2), 339-350 (2015).
- Song, W., Li, J., Liang, Q., Marchisio, M. A. Can terminators be used as insulators into yeast synthetic gene circuits. Journal of Biological Engineering. 10, 19 (2016).
- Rauch, B. J., et al. Inhibition of CRISPR-Cas9 with bacteriophage proteins. Cell. 168 (1-2), 150-158 (2017).
- Hynes, A. P., et al. An anti-CRISPR from a virulent streptococcal phage inhibits Streptococcus pyogenes Cas9. Nature Microbiology. 2 (10), 1374-1380 (2017).
- Watters, K. E., Fellmann, C., Bai, H. B., Ren, S. M., Doudna, J. A. Systematic discovery of natural CRISPR-Cas12a inhibitors. Science. 362 (6411), 236-239 (2018).
- Chen, C., et al. Real-time quantification of microRNAs by stem-loop RT-PCR. Nucleic Acids Research. 33 (20), 179 (2005).
- Pfaffl, M. W. A new mathematical model for relative quantification in real-time RT-PCR. Nucleic Acids Research. 29 (9), 45 (2001).
- Hahne, F., et al. flowCore: a Bioconductor package for high throughput flow cytometry. BMC Bioinformatics. 10 (1), 106 (2009).
- Li, J., Xu, Z., Chupalov, A., Marchisio, M. A. Anti-CRISPR-based biosensors in the yeast S. cerevisiae. Journal of Biological Engineering. 12, 11 (2018).
- Dong, L., et al. An anti-CRISPR protein disables type V Cas12a by acetylation. Nature Structural & Molecular Biology. 26 (4), 308-314 (2019).

