Circuits numériques géniques basés sur des systèmes CRISPR-CAS et des protéines anti-CRISPR
Summary
Les systèmes CRISPR-Cas et les protéines anti-CRISPR ont été intégrés dans le schéma des portes booléennes chez Saccharomyces cerevisiae. Les nouveaux petits circuits logiques ont montré de bonnes performances et approfondi la compréhension des facteurs de transcription basés sur dCas9 / dCas12a et des propriétés des protéines anti-CRISPR.
Abstract
Les portes booléennes géniques synthétiques et les circuits numériques ont un large éventail d’applications, du diagnostic médical à la protection de l’environnement. La découverte des systèmes CRISPR-Cas et de leurs inhibiteurs naturels, les protéines anti-CRISPR (Acrs), fournit un nouvel outil pour concevoir et mettre en œuvre des circuits numériques géniques in vivo . Ici, nous décrivons un protocole qui suit l’idée du cycle de génie biologique « Design-Build-Test-Learn » et utilise dCas9 / dCas12a avec leurs Acrs correspondants pour établir de petits réseaux transcriptionnels, dont certains se comportent comme des portes booléennes, chez Saccharomyces cerevisiae. Ces résultats soulignent les propriétés de dCas9/dCas12a comme facteurs de transcription. En particulier, pour obtenir une activation maximale de l’expression génique, dSpCas9 doit interagir avec un ARN d’échafaudage modifié qui collecte plusieurs copies du domaine d’activation (AD) VP64. En revanche, dCas12a doit être fusionné, à l’extrémité C, avec le puissant VP64-p65-Rta (VPR) AD. De plus, l’activité des deux protéines Cas n’est pas améliorée par l’augmentation de la quantité d’ARNg / ARNcr dans la cellule. Cet article explique également comment construire des portes booléennes basées sur l’interaction CRISPR-dCas-Acr. Le domaine de liaison hormonale fusionné AcrIIA4 du récepteur humain des œstrogènes est le cœur d’une porte NOT sensible au β-estradiol, tandis que les AcrVAs synthétisés par le promoteur GAL1 inductible permettent d’imiter les portes YES et NOT avec le galactose comme entrée. Dans ces derniers circuits, AcrVA5, avec dLbCas12a, a montré le meilleur comportement logique.
Introduction
En 2011, des chercheurs ont proposé une méthode de calcul et développé un logiciel correspondant pour la conception automatique de circuits génétiques synthétiques numériques1. Un utilisateur devait spécifier le nombre d’entrées (trois ou quatre) et remplir la table de vérité du circuit; Cela a fourni toutes les informations nécessaires pour dériver la structure du circuit en utilisant des techniques de l’électronique. La table de vérité a été traduite en deux formules booléennes via la méthode2 de la carte de Karnaugh. Chaque formule booléenne est constituée de clauses qui décrivent des opérations logiques (somme ou multiplication) entre (partie de) les entrées du circuit et leurs négations (les littéraux). Les clauses, à leur tour, sont soit additionnées (OR) ou multipliées (ET) pour calculer la sortie du circuit. Chaque circuit peut être réalisé selon l’une de ses deux formules correspondantes : l’une écrite sous forme POS (produit des sommes) et l’autre en SOP (somme des produits). Le premier consiste en une multiplication de clauses (c’est-à-dire des portes booléennes) qui contiennent une somme logique des littéraux. Ce dernier, en revanche, est une somme de clauses où les littéraux sont multipliés.
Les circuits électriques peuvent être réalisés, sur une planche à pain, en câblant physiquement différentes portes ensemble. Le courant électrique permet l’échange de signaux entre les portes, ce qui conduit au calcul de la sortie.
En biologie, la situation est plus complexe. Une porte booléenne peut être réalisée comme une unité de transcription (TU; c’est-à-dire la séquence « promoteur-codant région-terminateur » à l’intérieur des cellules eucaryotes), où la transcription ou la traduction (ou les deux) sont régulées. Ainsi, au moins deux sortes de molécules établissent un câblage biologique : les protéines du facteur de transcription et les ARN antisens non codants1.
Un circuit numérique génétique est organisé en deux ou trois couches de portes, à savoir: 1) la couche d’entrée, qui est faite de portes YES (tampon) et NOT et convertit les produits chimiques d’entrée en molécules de câblage; 2) la couche interne, qui se compose d’autant de TU qu’il y a de clauses dans la formule booléenne correspondante. Si le circuit est conçu selon la formule SOP, chaque clause de la couche interne produira la sortie du circuit (par exemple, la fluorescence) dans une architecture dite de sortie distribuée. Si la formule du produit de somme (POS) est utilisée, alors une couche finale 3) est nécessaire, qui contiendra une seule porte multiplicative collectant les molécules de câblage de la couche interne.
Dans l’ensemble, en biologie synthétique, de nombreux schémas différents peuvent être conçus pour le même circuit. Ils diffèrent par le nombre et le type de TU et de molécules de câblage. Afin de choisir la solution la plus simple à mettre en œuvre dans les cellules de levure, chaque conception de circuit est associée à un score de complexité S, défini par
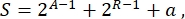
où A représente le nombre d’activateurs, R représente le nombre de répresseurs et a est la quantité de molécules d’ARN antisens. Si les activateurs ou les répresseurs sont absents du circuit, leur contribution à S est nulle. Par conséquent, il est plus difficile de réaliser un schéma de circuit en laboratoire (S élevé) lorsqu’il nécessite un nombre élevé de facteurs de transcription orthogonaux. Cela signifie que de nouveaux activateurs et répresseurs doivent être conçus de novo afin de réaliser le câblage complet à l’intérieur des circuits numériques. En principe, de nouvelles protéines de liaison à l’ADN peuvent être assemblées en utilisant les protéines Zinc Finger3 et les effecteurs TAL4 comme modèles. Cependant, cette option semble trop ardue et prend trop de temps; par conséquent, il faut s’appuyer principalement sur les petits ARN et la régulation de la traduction pour finaliser des circuits génétiques complexes.
À l’origine, cette méthode a été développée pour fabriquer des circuits numériques dans des bactéries. En effet, dans les cellules eucaryotes, au lieu d’ARN antisens, il est plus approprié de parler de microARN (miARN) ou de petits ARN interférents (siRNAs)5. Cependant, la voie ARNi n’est pas présente dans la levure S. cerevisiae. Par conséquent, il faut opter pour des réseaux entièrement transcriptionnels. Supposons qu’un circuit ait besoin de cinq activateurs et de cinq répresseurs; son score de complexité serait S = 32. La complexité du circuit peut être réduite en remplaçant les 10 facteurs de transcription par un seul dCas96 (Cas9 déficient en nucléase) fusionné à un domaine d’activation (AD). Comme le montrele point 7, dCas9-AD fonctionne comme un répresseur dans la levure lors de la liaison d’un promoteur entre la boîte TATA et le TSS (site de début de transcription) et comme activateur lors de la liaison bien en amont de la boîte TATA. Ainsi, on peut remplacer 10 facteurs de transcription par une seule protéine de fusion dCas9-AD et 10 sgRNA (ARN guides uniques) pour un score de complexité totale de S = 11. Il est rapide et facile de synthétiser dix ARNg, alors que, comme indiqué précédemment, l’assemblage de 10 protéines nécessiterait un travail beaucoup plus long et plus compliqué.
Alternativement, on peut utiliser deux protéines orthogonales dCas (par exemple, dCas9 et dCas12a): l’une pour fusionner avec une AD, et l’autre nue ou en combinaison avec un domaine de répression. Le score de complexité n’augmenterait que d’une unité (S = 12). Par conséquent, les systèmes CRISPR-dCas sont la clé de la construction de circuits numériques de gènes très complexes chez S. cerevisiae.
Cet article caractérise en profondeur l’efficacité des répresseurs et des activateurs à base de dCas9 et dCas12a chez la levure. Les résultats montrent qu’ils ne nécessitent pas une grande quantité d’ARNg pour optimiser leur activité, de sorte que les plasmides épisomiques sont préférentiellement évités. De plus, les activateurs basés sur dCas9 sont beaucoup plus efficaces lorsqu’ils utilisent un ARN d’échafaudage (scRNA) qui recrute des copies du VP64 AD. En revanche, dCas12a fonctionne bien lorsqu’il est fusionné directement avec le puissant VPR AD. De plus, un promoteur activé synthétique exige un nombre variable de sites cibles, en fonction de la configuration de l’activateur (par exemple, trois lors de l’utilisation de dCas12a-VPR, six pour dCas9-VP64 et un seul avec dCas9 et un scRNA). En tant que répresseur, dCas12a apparaît plus incisif lors de la liaison de la région de codage plutôt que du promoteur.
Cependant, CRISPR-dCas9 / dCas12a n’interagissent pas directement avec les produits chimiques. Par conséquent, ils peuvent ne pas être utiles dans la couche d’entrée. Pour cette raison, d’autres conceptions de portes booléennes contenant des protéines anti-CRISPR (Acrs) ont été étudiées. Les ACRS agissent sur les protéines (d)Cas et inhibent leur fonctionnement8. Par conséquent, ils sont un moyen de moduler l’activité des systèmes CRISPR-(d)Cas. Cet article analyse en profondeur les interactions entre les Acrs de type II et (d)Cas9, ainsi que les Acrs de type V et (d)Cas12a chez S. cerevisiae. Étant donné que les Acr sont beaucoup plus petits que les protéines Cas, une porte NOT sensible à l’œstrogène β-estradiol a été construite en fusionnant le domaine de liaison hormonale du récepteur humaindes œstrogènes 9-HBD(hER)-à AcrIIA4. En outre, une poignée de portes YES et NOT qui exprimaient dCas12a (-AD) de manière constitutive et AcrVAs lors de l’induction avec du galactose ont été réalisées. À l’heure actuelle, ces portes ne servent que de preuve de concept. Cependant, ils représentent également la première étape vers une refonte profonde de l’algorithme pour réaliser la conception automatique computationnelle de circuits numériques de gènes synthétiques dans les cellules de levure.
Protocol
Representative Results
Discussion
Le protocole a montré un flux de travail complet possible pour les circuits numériques de gènes synthétiques, suivant le cycle de génie biologique « Design-Build-Test-Learn » (DBTL) et concernant à la fois des expériences de laboratoire sec et de laboratoire humide. Ici, nous nous sommes concentrés sur le système CRISPR-Cas, principalement dSpCas9, denAsCas12a, dLbCas12a et les protéines anti-CRISPR correspondantes, en concevant et en construisant dans S. cerevisiae de petits réseaux transcription…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Nous tenons à remercier tous les étudiants du laboratoire de biologie synthétique SPST, TJU pour leur aide générale, ainsi que Zhi Li et Xiangyang Zhang pour leur aide dans les expériences FACS.
Materials
| 0.1 mL PCR 8-strip tubes | NEST | 403112 | |
| 0.2 mL PCR tubes | Axygen | PCR-02-C | |
| 1.5 mL Microtubes | Axygen | MCT-150-C | |
| 15 mL Centrifuge tubes | BIOFIL | CFT011150 | |
| 2 mL Microtubes | Axygen | MCT-200-C | |
| 50 mL Centrifuge tubes | BIOFIL | CFT011500 | |
| Agarose-molecular biology grade | Invitrogen | 75510-019 | |
| Ampicillin sodium salt | Solarbio | 69-52-3 | |
| Applied biosystems veriti 96-well thermal cycler | Thermo Fisher Scientific | 4375786 | |
| AxyPrep DNA gel extraction kit | Axygen | AP-GX-250 | |
| BD FACSuite CS&T research beads | BD | 650621 | Fluorescent beads |
| BD FACSVerse flow cytometer | BD | – | |
| Centrifuge | Eppendorf | 5424 | |
| Centrifuge Sorvall ST 16R | Thermo Fisher Scientific | 75004380 | |
| E. coli competent cells (Strain DH5α) | Life Technologies | 18263-012 | |
| ECL select Western Blotting detection reagent | GE Healthcare | RPN2235 | |
| Electrophoresis apparatus | Beijing JUNYI Electrophoresis Co., Ltd | JY300C | |
| Flat 8-strip caps | NEST | 406012 | |
| Gene synthesis company | Azenta Life Sciences | https://web.azenta.com/zh-cn/azenta-life-sciences | |
| Goat anti-Mouse IgG (H+L) cross-adsorbed secondary antibody Alexa Fluor 568 | Invitrogen | A-11004 | |
| HiFiScript cDNA synthesis kit | CWBIO | CW2569M | Kit used in step 6.2.2.1 |
| Lysate solution (Zymolyase) | zymoresearch | E1004-A | |
| Nikon Eclipse 80i fluorescence microscope | Nikon | – | Fluorescence microscope |
| Pipet tips—10 μL | Axygen | T-300-R-S | |
| Pipet tips—1000 μL | Axygen | T-1000-B-R-S | |
| Pipet tips—200 μL | Axygen | T-200-Y-R-S | |
| pRSII403 | Addgene | 35436 | |
| pRSII404 | Addgene | 35438 | |
| pRSII405 | Addgene | 35440 | |
| pRSII406 | Addgene | 35442 | |
| pRSII424 | Addgene | 35466 | |
| pTPGI_dSpCas9_VP64 | Addgene | 49013 | |
| Q5 High-fidelity DNApolymerase | New England Biolabs | M0491 | |
| Restriction enzyme-Acc65I | New England Biolabs | R0599 | |
| Restriction enzyme-BamHI | New England Biolabs | R0136 | |
| Restriction enzyme-SacI-HF | New England Biolabs | R3156 | |
| Restriction enzyme-XhoI | New England Biolabs | R0146 | |
| Roche LightCycler 96 | Roche | – | Real-Time PCR Instrument |
| S. cerevisiae CEN.PK2-1C | – | – | The parent strain. The genotype is: MATa; his3D1; leu2-3_112; ura3-52; trp1-289; MAL2-8c; SUC2 |
| Stem-Loop Kit | SparkJade | AG0502 | Kit used in step 6.2.1.3 |
| T100 Thermal Cycler | BIO-RAD | 186-1096 | |
| T4 DNA ligase | New England Biolabs | M0202 | |
| T5 Exonuclease | New England Biolabs | M0363 | |
| Taq DNA ligase | New England Biolabs | M0208 | |
| Taq DNA polymerase | New England Biolabs | M0495 | |
| TB Green Premix Ex Taq II (Tli RNaseH Plus)(2x) (SYBR Green I dye) | Takara | RR820Q | |
| YeaStar RNA kit | Zymo Research | R1002 | |
| β-estradiol | Sigma-Aldrich | E8875 |
References
- Marchisio, M. A., Stelling, J. Automatic design of digital synthetic gene circuits. PLOS Computational Biology. 7 (2), 1001083 (2011).
- Karnaugh, M. The map method for synthesis of combinational logic circuits. Transactions of the American Institute of Electrical Engineers. 72 (9), 593-599 (1953).
- Mandell, J. G., Barbas, C. F. Zinc finger tools: custom DNA-binding domains for transcription factors and nucleases. Nucleic Acids Research. 34, 516-523 (2006).
- Bogdanove, A. J., Voytas, D. F. TAL effectors: customizable proteins for DNA targeting. Science. 333 (6051), 1843-1846 (2011).
- Drinnenberg, I. A., et al. RNAi in budding yeast. Science. 326 (5952), 544-550 (2009).
- Gander, M. W., Vrana, J. D., Voje, W. E., Carothers, J. M., Klavins, E. Digital logic circuits in yeast with CRISPR-dCas9 NOR gates. Nature Communications. 8, 15459 (2017).
- Farzadfard, F., Perli, S. D., Lu, T. K. Tunable and multifunctional eukaryotic transcription factors based on CRISPR/Cas. ACS Synthetic Biology. 2 (10), 604-613 (2013).
- Nakamura, M., et al. Anti-CRISPR-mediated control of gene editing and synthetic circuits in eukaryotic cells. Nature Communications. 10 (1), 194 (2019).
- Louvion, J. F., Havaux-Copf, B., Picard, D. Fusion of GAL4-VP16 to a steroid-binding domain provides a tool for gratuitous induction of galactose-responsive genes in yeast. Gene. 131 (1), 129-134 (1993).
- DiCarlo, J. E., et al. Genome engineering in Saccharomyces cerevisiae using CRISPR-Cas systems. Nucleic Acids Research. 41 (7), 4336-4343 (2013).
- Gao, Y., Zhao, Y. Self-processing of ribozyme-flanked RNAs into guide RNAs in vitro and in vivo for CRISPR-mediated genome editing. Journal of Integrative Plant Biology. 56 (4), 343-349 (2014).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Zetsche, B., et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell. 163 (3), 759-771 (2015).
- Fonfara, I., Richter, H., Bratovic, M., Le Rhun, A., Charpentier, E. The CRISPR-associated DNA-cleaving enzyme Cpf1 also processes precursor CRISPR RNA. Nature. 532 (7600), 517-521 (2016).
- Yu, L., Marchisio, M. A. Saccharomyces cerevisiae synthetic transcriptional networks harnessing dCas12a and Type V-A anti-CRISPR proteins. ACS Synthetic Biology. 10 (4), 870-883 (2021).
- Zhang, Y., Marchisio, M. A. Interaction of bare dSpCas9, scaffold gRNA, and type II anti-CRISPR proteins highly favors the control of gene expression in the yeast S. cerevisiae. ACS Synthetic Biology. 11 (1), 176-190 (2022).
- Sheff, M. A., Thorn, K. S. Optimized cassettes for fluorescent protein tagging in Saccharomyces cerevisiae. Yeast. 21 (8), 661-670 (2004).
- Naito, Y., Hino, K., Bono, H., Ui-Tei, K. CRISPRdirect: software for designing CRISPR/Cas guide RNA with reduced off-target sites. Bioinformatics. 31 (7), 1120-1123 (2015).
- Chee, M. K., Haase, S. B. New and redesigned pRS plasmid shuttle vectors for genetic manipulation of Saccharomyces cerevisiae. G3: Genes|Genomes|Genetics. 2 (5), 515-526 (2012).
- Gibson, D. G. Synthesis of DNA fragments in yeast by one-step assembly of overlapping oligonucleotides. Nucleic Acids Research. 37 (20), 6984-6990 (2009).
- Froger, A., Hall, J. E. Transformation of plasmid DNA into E. coli using the heat shock method. Journal of Visualized Experiments. (6), e253 (2007).
- Green, M. R., Sambrook, J. . Molecular Cloning. Fourth edition. , (2012).
- Sanger, F. Determination of nucleotide sequences in DNA. Science. 214 (4526), 1205-1210 (1981).
- Gietz, R. D., Woods, R. A. Transformation of yeast by lithium acetate/single-stranded carrier DNA/polyethylene glycol method. Methods in Enzymology. 350, 87-96 (2002).
- Zalatan, J. G., et al. Engineering complex synthetic transcriptional programs with CRISPR RNA scaffolds. Cell. 160 (1-2), 339-350 (2015).
- Song, W., Li, J., Liang, Q., Marchisio, M. A. Can terminators be used as insulators into yeast synthetic gene circuits. Journal of Biological Engineering. 10, 19 (2016).
- Rauch, B. J., et al. Inhibition of CRISPR-Cas9 with bacteriophage proteins. Cell. 168 (1-2), 150-158 (2017).
- Hynes, A. P., et al. An anti-CRISPR from a virulent streptococcal phage inhibits Streptococcus pyogenes Cas9. Nature Microbiology. 2 (10), 1374-1380 (2017).
- Watters, K. E., Fellmann, C., Bai, H. B., Ren, S. M., Doudna, J. A. Systematic discovery of natural CRISPR-Cas12a inhibitors. Science. 362 (6411), 236-239 (2018).
- Chen, C., et al. Real-time quantification of microRNAs by stem-loop RT-PCR. Nucleic Acids Research. 33 (20), 179 (2005).
- Pfaffl, M. W. A new mathematical model for relative quantification in real-time RT-PCR. Nucleic Acids Research. 29 (9), 45 (2001).
- Hahne, F., et al. flowCore: a Bioconductor package for high throughput flow cytometry. BMC Bioinformatics. 10 (1), 106 (2009).
- Li, J., Xu, Z., Chupalov, A., Marchisio, M. A. Anti-CRISPR-based biosensors in the yeast S. cerevisiae. Journal of Biological Engineering. 12, 11 (2018).
- Dong, L., et al. An anti-CRISPR protein disables type V Cas12a by acetylation. Nature Structural & Molecular Biology. 26 (4), 308-314 (2019).

