Circuiti digitali genici basati su sistemi CRISPR-Cas e proteine anti-CRISPR
Summary
I sistemi CRISPR-Cas e le proteine anti-CRISPR sono stati integrati nello schema delle porte booleane in Saccharomyces cerevisiae. I nuovi piccoli circuiti logici hanno mostrato buone prestazioni e approfondito la comprensione sia dei fattori di trascrizione basati su dCas9/dCas12a che delle proprietà delle proteine anti-CRISPR.
Abstract
I gate booleani e i circuiti digitali dei geni sintetici hanno una vasta gamma di applicazioni, dalla diagnostica medica alla cura dell’ambiente. La scoperta dei sistemi CRISPR-Cas e dei loro inibitori naturali, le proteine anti-CRISPR (Acrs), fornisce un nuovo strumento per progettare e implementare circuiti digitali genici in vivo . Qui, descriviamo un protocollo che segue l’idea del ciclo di ingegneria biologica “Design-Build-Test-Learn” e fa uso di dCas9 / dCas12a insieme ai loro corrispondenti Acr per stabilire piccole reti trascrizionali, alcune delle quali si comportano come porte booleane, in Saccharomyces cerevisiae. Questi risultati evidenziano le proprietà di dCas9/dCas12a come fattori di trascrizione. In particolare, per ottenere la massima attivazione dell’espressione genica, dSpCas9 deve interagire con un RNA scaffold ingegnerizzato che raccoglie più copie del dominio di attivazione VP64 (AD). Al contrario, dCas12a deve essere fuso, al terminale C, con il forte VP64-p65-Rta (VPR) AD. Inoltre, l’attività di entrambe le proteine Cas non è potenziata aumentando la quantità di sgRNA/crRNA nella cellula. Questo articolo spiega anche come costruire porte booleane basate sull’interazione CRISPR-dCas-Acr. Il dominio di legame ormonale fuso AcrIIA4 del recettore degli estrogeni umani è il nucleo di un gate NOT sensibile al β-estradiolo, mentre gli AcrVA sintetizzati dal promotore inducibile GAL1 consentono di imitare sia le porte SÌ che NOT con galattosio come input. In questi ultimi circuiti, AcrVA5, insieme a dLbCas12a, ha mostrato il miglior comportamento logico.
Introduction
Nel 2011, i ricercatori hanno proposto un metodo computazionale e sviluppato un software corrispondente per la progettazione automatica di circuiti genetici sintetici digitali1. Un utente doveva specificare il numero di ingressi (tre o quattro) e compilare la tabella di verità del circuito; Ciò ha fornito tutte le informazioni necessarie per derivare la struttura del circuito utilizzando tecniche dall’elettronica. La tavola di verità è stata tradotta in due formule booleane tramite il metodo 2 della mappa di Karnaugh. Ogni formula booleana è composta da clausole che descrivono le operazioni logiche (somma o moltiplicazione) tra (parte di) gli ingressi del circuito e le loro negazioni (i letterali). Le clausole, a loro volta, vengono sommate (OR) o moltiplicate (AND) per calcolare l’uscita del circuito. Ogni circuito può essere realizzato secondo una qualsiasi delle sue due formule corrispondenti: una scritta in forma POS (prodotto di somme) e l’altra in rappresentazione SOP (somma di prodotti). Il primo consiste in una moltiplicazione di clausole (cioè porte booleane) che contengono una somma logica dei letterali. Quest’ultimo, al contrario, è una somma di clausole in cui i valori letterali sono moltiplicati.
I circuiti elettrici possono essere realizzati, su una breadboard, collegando fisicamente diversi cancelli insieme. La corrente elettrica consente lo scambio di segnali tra i gate, che porta al calcolo dell’uscita.
In biologia, la situazione è più complessa. Un gate booleano può essere realizzato come unità di trascrizione (TU; cioè la sequenza “promotore-codificante regione-terminatore” all’interno di cellule eucariotiche), dove la trascrizione o la traduzione (o entrambe) sono regolate. Quindi, almeno due tipi di molecole stabiliscono un cablaggio biologico: le proteine del fattore di trascrizione e gli RNA antisenso non codificanti1.
Un circuito digitale genico è organizzato in due o tre strati di porte, vale a dire: 1) lo strato di ingresso, che è costituito da SÌ (buffer) e NON porte e converte le sostanze chimiche in ingresso in molecole di cablaggio; 2) il livello interno, che consiste di tanti TU quante sono le clausole nella corrispondente formula booleana. Se il circuito è progettato secondo la formula SOP, ogni clausola nello strato interno produrrà l’uscita del circuito (ad esempio, fluorescenza) in una cosiddetta architettura di uscita distribuita. Se viene utilizzata la formula del prodotto della somma (POS), è necessario uno strato finale 3), che conterrà un singolo gate moltiplicativo che raccoglie le molecole di cablaggio dallo strato interno.
Nel complesso, in biologia sintetica, molti schemi diversi possono essere progettati per lo stesso circuito. Differiscono nel numero e nel tipo di TU e molecole di cablaggio. Al fine di scegliere la soluzione più semplice da implementare nelle cellule di lievito, ogni progetto di circuito è associato a un punteggio di complessità S, definito come
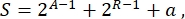
dove A rappresenta il numero di attivatori, R rappresenta il numero di repressori e A è la quantità di molecole di RNA antisenso. Se gli attivatori o i repressori sono assenti dal circuito, il loro contributo a S è zero. Pertanto, è più difficile realizzare uno schema circuitale in laboratorio (alto S) quando richiede un numero elevato di fattori di trascrizione ortogonali. Ciò significa che nuovi attivatori e repressori devono essere progettati de novo per realizzare il cablaggio completo all’interno dei circuiti digitali. In linea di principio, nuove proteine leganti il DNA possono essere assemblate utilizzando le proteine Zinc Finger3 e gli effettori TAL4 come modelli. Tuttavia, questa opzione appare troppo ardua e richiede tempo; pertanto, si dovrebbe fare affidamento principalmente su piccoli RNA e regolazione della traduzione per finalizzare circuiti genici complessi.
Originariamente, questo metodo è stato sviluppato per fabbricare circuiti digitali nei batteri. Infatti, nelle cellule eucariotiche, invece che di RNA antisenso, è più adatto parlare di microRNA (miRNA) o piccoli RNA interferenti (siRNA)5. Tuttavia, la via dell’RNAi non è presente nel lievito S. cerevisiae. Quindi, si dovrebbe optare per reti completamente trascrizionali. Supponiamo che un circuito abbia bisogno di cinque attivatori e cinque repressori; il suo punteggio di complessità sarebbe S = 32. La complessità del circuito può essere ridotta sostituendo i 10 fattori di trascrizione con un singolo dCas96 (Cas9 carente di nucleasi) fuso in un dominio di attivazione (AD). Come mostrato in7, dCas9-AD funziona come repressore nel lievito quando lega un promotore tra la scatola TATA e il TSS (sito di inizio della trascrizione) e come attivatore quando si lega bene a monte della scatola TATA. Pertanto, è possibile sostituire 10 fattori di trascrizione con una singola proteina di fusione dCas9-AD e 10 sgRNA (RNA guida singola) per un punteggio di complessità totale di S = 11. È facile e veloce sintetizzare dieci sgRNA, mentre, come precedentemente commentato, l’assemblaggio di 10 proteine richiederebbe un lavoro molto più lungo e complicato.
In alternativa, si potrebbero usare due proteine dCas ortogonali (ad esempio, dCas9 e dCas12a): una per fondersi con un AD, e l’altra nuda o in combinazione con un dominio di repressione. Il punteggio di complessità aumenterebbe di una sola unità (S = 12). Quindi, i sistemi CRISPR-dCas sono la chiave per la costruzione di circuiti digitali genici molto complessi in S. cerevisiae.
Questo documento caratterizza profondamente l’efficienza dei repressori e degli attivatori basati su dCas9 e dCas12a nel lievito. I risultati mostrano che non richiedono un’elevata quantità di sgRNA per ottimizzare la loro attività, quindi i plasmidi episomiali sono preferenzialmente evitati. Inoltre, gli attivatori basati su dCas9 sono molto più efficaci quando si utilizza uno scaffold RNA (scRNA) che recluta copie del VP64 AD. Al contrario, dCas12a funziona bene se fuso direttamente al potente VPR AD. Inoltre, un promotore sintetico attivato richiede un numero variabile di siti bersaglio, a seconda della configurazione dell’attivatore (ad esempio, tre quando si utilizza dCas12a-VPR, sei per dCas9-VP64 e solo uno con dCas9 e uno scRNA). Come repressore, dCas12a appare più incisivo quando si lega la regione codificante piuttosto che il promotore.
Come svantaggio, tuttavia, CRISPR-dCas9 / dCas12a non interagiscono direttamente con le sostanze chimiche. Pertanto, potrebbero non essere utili nel livello di input. Per questo motivo, sono stati studiati progetti alternativi di gate booleani contenenti proteine anti-CRISPR (Acrs). Gli Acr agiscono sulle proteine (d)Cas e ne inibiscono il funzionamento8. Quindi, sono un mezzo per modulare l’attività dei sistemi CRISPR-(d)Cas. Questo articolo analizza a fondo le interazioni tra il tipo II Acrs e (d)Cas9, così come il tipo V Acrs e (d)Cas12a in S. cerevisiae. Poiché gli Acr sono molto più piccoli delle proteine Cas, è stato costruito un gate NOT sensibile all’estrogeno β-estradiolo fondendo il dominio legante l’ormone del recettore degli estrogeni umani9-HBD (hER) ad AcrIIA4. Inoltre, sono state realizzate una manciata di porte SÌ e NON che esprimevano costitutivamente dCas12a(-AD) e AcrVA dopo induzione con galattosio. Al momento, queste porte servono solo come prova di concetto. Tuttavia, rappresentano anche il primo passo verso un profondo ripensamento dell’algoritmo per effettuare la progettazione automatica computazionale di circuiti digitali genici sintetici nelle cellule di lievito.
Protocol
Representative Results
Discussion
Il protocollo ha mostrato un possibile flusso di lavoro completo per circuiti digitali di geni sintetici, seguendo il ciclo di ingegneria biologica “Design-Build-Test-Learn” (DBTL) e riguardante sia esperimenti di laboratorio secco che di laboratorio umido. Qui, ci siamo concentrati sul sistema CRISPR-Cas, principalmente dSpCas9, denAsCas12a, dLbCas12a e le corrispondenti proteine anti-CRISPR, progettando e costruendo in S. cerevisiae piccole reti trascrizionali. Alcuni di loro imitavano le porte booleane, che s…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Vogliamo ringraziare tutti gli studenti del laboratorio di biologia sintetica – SPST, TJU – per il loro aiuto generale, insieme a Zhi Li e Xiangyang Zhang per la loro assistenza negli esperimenti FACS.
Materials
| 0.1 mL PCR 8-strip tubes | NEST | 403112 | |
| 0.2 mL PCR tubes | Axygen | PCR-02-C | |
| 1.5 mL Microtubes | Axygen | MCT-150-C | |
| 15 mL Centrifuge tubes | BIOFIL | CFT011150 | |
| 2 mL Microtubes | Axygen | MCT-200-C | |
| 50 mL Centrifuge tubes | BIOFIL | CFT011500 | |
| Agarose-molecular biology grade | Invitrogen | 75510-019 | |
| Ampicillin sodium salt | Solarbio | 69-52-3 | |
| Applied biosystems veriti 96-well thermal cycler | Thermo Fisher Scientific | 4375786 | |
| AxyPrep DNA gel extraction kit | Axygen | AP-GX-250 | |
| BD FACSuite CS&T research beads | BD | 650621 | Fluorescent beads |
| BD FACSVerse flow cytometer | BD | – | |
| Centrifuge | Eppendorf | 5424 | |
| Centrifuge Sorvall ST 16R | Thermo Fisher Scientific | 75004380 | |
| E. coli competent cells (Strain DH5α) | Life Technologies | 18263-012 | |
| ECL select Western Blotting detection reagent | GE Healthcare | RPN2235 | |
| Electrophoresis apparatus | Beijing JUNYI Electrophoresis Co., Ltd | JY300C | |
| Flat 8-strip caps | NEST | 406012 | |
| Gene synthesis company | Azenta Life Sciences | https://web.azenta.com/zh-cn/azenta-life-sciences | |
| Goat anti-Mouse IgG (H+L) cross-adsorbed secondary antibody Alexa Fluor 568 | Invitrogen | A-11004 | |
| HiFiScript cDNA synthesis kit | CWBIO | CW2569M | Kit used in step 6.2.2.1 |
| Lysate solution (Zymolyase) | zymoresearch | E1004-A | |
| Nikon Eclipse 80i fluorescence microscope | Nikon | – | Fluorescence microscope |
| Pipet tips—10 μL | Axygen | T-300-R-S | |
| Pipet tips—1000 μL | Axygen | T-1000-B-R-S | |
| Pipet tips—200 μL | Axygen | T-200-Y-R-S | |
| pRSII403 | Addgene | 35436 | |
| pRSII404 | Addgene | 35438 | |
| pRSII405 | Addgene | 35440 | |
| pRSII406 | Addgene | 35442 | |
| pRSII424 | Addgene | 35466 | |
| pTPGI_dSpCas9_VP64 | Addgene | 49013 | |
| Q5 High-fidelity DNApolymerase | New England Biolabs | M0491 | |
| Restriction enzyme-Acc65I | New England Biolabs | R0599 | |
| Restriction enzyme-BamHI | New England Biolabs | R0136 | |
| Restriction enzyme-SacI-HF | New England Biolabs | R3156 | |
| Restriction enzyme-XhoI | New England Biolabs | R0146 | |
| Roche LightCycler 96 | Roche | – | Real-Time PCR Instrument |
| S. cerevisiae CEN.PK2-1C | – | – | The parent strain. The genotype is: MATa; his3D1; leu2-3_112; ura3-52; trp1-289; MAL2-8c; SUC2 |
| Stem-Loop Kit | SparkJade | AG0502 | Kit used in step 6.2.1.3 |
| T100 Thermal Cycler | BIO-RAD | 186-1096 | |
| T4 DNA ligase | New England Biolabs | M0202 | |
| T5 Exonuclease | New England Biolabs | M0363 | |
| Taq DNA ligase | New England Biolabs | M0208 | |
| Taq DNA polymerase | New England Biolabs | M0495 | |
| TB Green Premix Ex Taq II (Tli RNaseH Plus)(2x) (SYBR Green I dye) | Takara | RR820Q | |
| YeaStar RNA kit | Zymo Research | R1002 | |
| β-estradiol | Sigma-Aldrich | E8875 |
References
- Marchisio, M. A., Stelling, J. Automatic design of digital synthetic gene circuits. PLOS Computational Biology. 7 (2), 1001083 (2011).
- Karnaugh, M. The map method for synthesis of combinational logic circuits. Transactions of the American Institute of Electrical Engineers. 72 (9), 593-599 (1953).
- Mandell, J. G., Barbas, C. F. Zinc finger tools: custom DNA-binding domains for transcription factors and nucleases. Nucleic Acids Research. 34, 516-523 (2006).
- Bogdanove, A. J., Voytas, D. F. TAL effectors: customizable proteins for DNA targeting. Science. 333 (6051), 1843-1846 (2011).
- Drinnenberg, I. A., et al. RNAi in budding yeast. Science. 326 (5952), 544-550 (2009).
- Gander, M. W., Vrana, J. D., Voje, W. E., Carothers, J. M., Klavins, E. Digital logic circuits in yeast with CRISPR-dCas9 NOR gates. Nature Communications. 8, 15459 (2017).
- Farzadfard, F., Perli, S. D., Lu, T. K. Tunable and multifunctional eukaryotic transcription factors based on CRISPR/Cas. ACS Synthetic Biology. 2 (10), 604-613 (2013).
- Nakamura, M., et al. Anti-CRISPR-mediated control of gene editing and synthetic circuits in eukaryotic cells. Nature Communications. 10 (1), 194 (2019).
- Louvion, J. F., Havaux-Copf, B., Picard, D. Fusion of GAL4-VP16 to a steroid-binding domain provides a tool for gratuitous induction of galactose-responsive genes in yeast. Gene. 131 (1), 129-134 (1993).
- DiCarlo, J. E., et al. Genome engineering in Saccharomyces cerevisiae using CRISPR-Cas systems. Nucleic Acids Research. 41 (7), 4336-4343 (2013).
- Gao, Y., Zhao, Y. Self-processing of ribozyme-flanked RNAs into guide RNAs in vitro and in vivo for CRISPR-mediated genome editing. Journal of Integrative Plant Biology. 56 (4), 343-349 (2014).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Zetsche, B., et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell. 163 (3), 759-771 (2015).
- Fonfara, I., Richter, H., Bratovic, M., Le Rhun, A., Charpentier, E. The CRISPR-associated DNA-cleaving enzyme Cpf1 also processes precursor CRISPR RNA. Nature. 532 (7600), 517-521 (2016).
- Yu, L., Marchisio, M. A. Saccharomyces cerevisiae synthetic transcriptional networks harnessing dCas12a and Type V-A anti-CRISPR proteins. ACS Synthetic Biology. 10 (4), 870-883 (2021).
- Zhang, Y., Marchisio, M. A. Interaction of bare dSpCas9, scaffold gRNA, and type II anti-CRISPR proteins highly favors the control of gene expression in the yeast S. cerevisiae. ACS Synthetic Biology. 11 (1), 176-190 (2022).
- Sheff, M. A., Thorn, K. S. Optimized cassettes for fluorescent protein tagging in Saccharomyces cerevisiae. Yeast. 21 (8), 661-670 (2004).
- Naito, Y., Hino, K., Bono, H., Ui-Tei, K. CRISPRdirect: software for designing CRISPR/Cas guide RNA with reduced off-target sites. Bioinformatics. 31 (7), 1120-1123 (2015).
- Chee, M. K., Haase, S. B. New and redesigned pRS plasmid shuttle vectors for genetic manipulation of Saccharomyces cerevisiae. G3: Genes|Genomes|Genetics. 2 (5), 515-526 (2012).
- Gibson, D. G. Synthesis of DNA fragments in yeast by one-step assembly of overlapping oligonucleotides. Nucleic Acids Research. 37 (20), 6984-6990 (2009).
- Froger, A., Hall, J. E. Transformation of plasmid DNA into E. coli using the heat shock method. Journal of Visualized Experiments. (6), e253 (2007).
- Green, M. R., Sambrook, J. . Molecular Cloning. Fourth edition. , (2012).
- Sanger, F. Determination of nucleotide sequences in DNA. Science. 214 (4526), 1205-1210 (1981).
- Gietz, R. D., Woods, R. A. Transformation of yeast by lithium acetate/single-stranded carrier DNA/polyethylene glycol method. Methods in Enzymology. 350, 87-96 (2002).
- Zalatan, J. G., et al. Engineering complex synthetic transcriptional programs with CRISPR RNA scaffolds. Cell. 160 (1-2), 339-350 (2015).
- Song, W., Li, J., Liang, Q., Marchisio, M. A. Can terminators be used as insulators into yeast synthetic gene circuits. Journal of Biological Engineering. 10, 19 (2016).
- Rauch, B. J., et al. Inhibition of CRISPR-Cas9 with bacteriophage proteins. Cell. 168 (1-2), 150-158 (2017).
- Hynes, A. P., et al. An anti-CRISPR from a virulent streptococcal phage inhibits Streptococcus pyogenes Cas9. Nature Microbiology. 2 (10), 1374-1380 (2017).
- Watters, K. E., Fellmann, C., Bai, H. B., Ren, S. M., Doudna, J. A. Systematic discovery of natural CRISPR-Cas12a inhibitors. Science. 362 (6411), 236-239 (2018).
- Chen, C., et al. Real-time quantification of microRNAs by stem-loop RT-PCR. Nucleic Acids Research. 33 (20), 179 (2005).
- Pfaffl, M. W. A new mathematical model for relative quantification in real-time RT-PCR. Nucleic Acids Research. 29 (9), 45 (2001).
- Hahne, F., et al. flowCore: a Bioconductor package for high throughput flow cytometry. BMC Bioinformatics. 10 (1), 106 (2009).
- Li, J., Xu, Z., Chupalov, A., Marchisio, M. A. Anti-CRISPR-based biosensors in the yeast S. cerevisiae. Journal of Biological Engineering. 12, 11 (2018).
- Dong, L., et al. An anti-CRISPR protein disables type V Cas12a by acetylation. Nature Structural & Molecular Biology. 26 (4), 308-314 (2019).

