Цифровые схемы генов на основе систем CRISPR-Cas и белков анти-CRISPR
Summary
Системы CRISPR-Cas и анти-CRISPR белки были интегрированы в схему булевых ворот у Saccharomyces cerevisiae. Новые небольшие логические схемы показали хорошую производительность и углубили понимание как факторов транскрипции на основе dCas9/dCas12a, так и свойств белков анти-CRISPR.
Abstract
Логические вентили синтетических генов и цифровые схемы имеют широкий спектр применения, от медицинской диагностики до заботы об окружающей среде. Открытие систем CRISPR-Cas и их естественных ингибиторов — белков анти-CRISPR (Acrs) — предоставляет новый инструмент для разработки и реализации цифровых схем генов in vivo . Здесь мы описываем протокол, который следует идее цикла биологической инженерии «Проектирование-Строительство-Тестирование-Обучение» и использует dCas9/dCas12a вместе с соответствующими Acrs для создания небольших транскрипционных сетей, некоторые из которых ведут себя как логические ворота, в Saccharomyces cerevisiae. Эти результаты указывают на свойства dCas9/dCas12a в качестве факторов транскрипции. В частности, для достижения максимальной активации экспрессии генов dSpCas9 должен взаимодействовать с сконструированной каркасной РНК, которая собирает несколько копий домена активации VP64 (AD). Напротив, dCas12a должен быть сплавлен на конце C с сильным VP64-p65-Rta (VPR) AD. Кроме того, активность обоих белков Cas не усиливается за счет увеличения количества сгРНК/крРНК в клетке. В этой статье также объясняется, как построить логические вентили на основе взаимодействия CRISPR-dCas-Acr. Слитый гормон-связывающий домен AcrIIA4 рецептора эстрогена человека является ядром ворот NOT, реагирующих на β-эстрадиол, тогда как AcrVA, синтезированные индуцируемым промотором GAL1 , позволяют имитировать как YES, так и NOT gates с галактозой в качестве входа. В последних схемах AcrVA5 вместе с dLbCas12a показали наилучшее логическое поведение.
Introduction
В 2011 году исследователи предложили вычислительный метод и разработали соответствующее программное обеспечение для автоматического проектирования цифровых синтетических генных схем1. Пользователь должен был указать количество входов (три или четыре) и заполнить таблицу истинности схемы; Это предоставило всю необходимую информацию для получения структуры схемы с использованием методов электроники. Таблица истинности была переведена в две логические формулы с помощью методакарты Карно 2. Каждая булева формула состоит из предложений, описывающих логические операции (сумма или умножение) между (частью) входов схемы и их отрицаний (литералов). Предложения, в свою очередь, либо суммируются (ИЛИ), либо умножаются (И) для вычисления выходного сигнала схемы. Каждая схема может быть реализована в соответствии с любой из двух соответствующих формул: одна записана в форме POS (произведение сумм), а другая – в представлении SOP (сумма произведений). Первый состоит из умножения предложений (т. е. булевых ворот), которые содержат логическую сумму литералов. Последний, напротив, представляет собой сумму предложений, в которых перемножаются литералы.
Электрические цепи могут быть реализованы на макетной плате путем физического соединения различных ворот вместе. Электрический ток позволяет обмениваться сигналами между вентилями, что приводит к вычислению выхода.
В биологии ситуация сложнее. Булевы ворота могут быть реализованы как транскрипционная единица (TU; т.е. последовательность «промотор-кодирующая область-терминатор» внутри эукариотических клеток), где транскрипция или трансляция (или и то, и другое) регулируются. Таким образом, по крайней мере, два вида молекул устанавливают биологическую связь: белки фактора транскрипции и некодирующие, антисмысловые РНК1.
Цифровая схема гена организована в два или три слоя вентилей, а именно: 1) входной слой, который состоит из затворов YES (буфер) и NOT и преобразует входные химические вещества в молекулы проводки; 2) внутренний слой, который состоит из стольких ТУ, сколько предложений в соответствующей булевой формуле. Если схема спроектирована в соответствии с формулой СОП, каждый пункт во внутреннем слое будет производить выходной сигнал схемы (например, флуоресценцию) в так называемой распределенной выходной архитектуре. Если используется формула произведения суммы (POS), то требуется 3) конечный слой, который будет содержать один мультипликативный затвор, собирающий молекулы проводки из внутреннего слоя.
В целом, в синтетической биологии для одной и той же схемы может быть разработано множество различных схем. Они различаются по количеству и виду как TU, так и молекул проводки. Чтобы выбрать самое простое решение, которое будет реализовано в дрожжевых клетках, каждая схема связана с оценкой сложности S, определяемой как
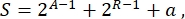
где A — количество активаторов, R — количество репрессоров, a — количество молекул антисмысловой РНК. Если в цепи отсутствуют активаторы или репрессоры, их вклад в S равен нулю. Следовательно, сложнее реализовать схему в лаборатории (высокий S), когда требуется большое количество ортогональных факторов транскрипции. Это означает, что новые активаторы и репрессоры должны быть спроектированы заново, чтобы реализовать полную проводку внутри цифровых цепей. В принципе, новые ДНК-связывающие белки могут быть собраны с использованием белков цинкового пальца3 и эффекторовTAL 4 в качестве шаблонов. Однако этот вариант представляется слишком трудным и трудоемким; поэтому следует полагаться в основном на малые РНК и регуляцию трансляции для завершения сложных генных цепей.
Первоначально этот метод был разработан для изготовления цифровых схем у бактерий. Действительно, в эукариотических клетках вместо антисмысловых РНК более уместно говорить о микроРНК (миРНК) или малых интерферирующих РНК (миРНК)5. Однако путь РНКи отсутствует у дрожжей S. cerevisiae. Следовательно, следует выбирать полностью транскрипционные сети. Предположим, что для цепи требуется пять активаторов и пять репрессоров; его оценка сложности будет S = 32. Сложность схемы может быть уменьшена путем замены 10 факторов транскрипции одним dCas96 (Cas9 с дефицитом нуклеазы), слитым с активационным доменом (AD). Как показано нарисунке 7, dCas9-AD работает как репрессор у дрожжей при связывании промотора между коробкой TATA и TSS (начальным сайтом транскрипции) и как активатор при связывании значительно выше по течению от коробки TATA. Таким образом, можно заменить 10 факторов транскрипции одним слитым белком dCas9-AD и 10 сгРНК (одиночными направляющими РНК) для общей оценки сложности S = 11. Синтезировать десять сгРНК можно быстро и легко, в то время как, как отмечалось ранее, сборка 10 белков потребовала бы гораздо более длительной и сложной работы.
В качестве альтернативы можно использовать два ортогональных белка dCas (например, dCas9 и dCas12a): один для слияния с AD, а другой голый или в сочетании с доменом репрессии. Оценка сложности увеличится всего на одну единицу (S = 12). Следовательно, системы CRISPR-dCas являются ключом к построению очень сложных цифровых схем генов у S. cerevisiae.
В данной работе подробно охарактеризована эффективность репрессоров и активаторов на основе dCas9 и dCas12a у дрожжей. Результаты показывают, что они не требуют большого количества сгРНК для оптимизации своей активности, поэтому эписомальные плазмиды предпочтительно избегают. Более того, активаторы на основе dCas9 гораздо более эффективны при использовании каркасной РНК (скРНК), которая рекрутирует копии VP64 AD. Напротив, dCas12a хорошо работает при непосредственном слиянии с сильным AD VPR. Кроме того, синтетический активированный промотор требует переменного числа целевых сайтов, в зависимости от конфигурации активатора (например, три при использовании dCas12a-VPR, шесть для dCas9-VP64 и только один с dCas9 и скРНК). В качестве репрессора dCas12a кажется более острым при связывании кодирующей области, а не промотора.
Однако недостатком является то, что CRISPR-dCas9/dCas12a не взаимодействуют с химическими веществами напрямую. Поэтому они могут быть бесполезны на входном уровне. По этой причине были исследованы альтернативные конструкции булевых затворов, содержащие белки анти-CRISPR (Acrs). Acrs действуют на (d)Cas белки и ингибируют их работу8. Следовательно, они являются средством модуляции активности систем CRISPR-(d)Cas. В данной работе подробно анализируются взаимодействия между типом II Acrs и (d)Cas9, а также типом V Acrs и (d)Cas12a у S. cerevisiae. Поскольку Acrs намного меньше, чем белки Cas, ворота NOT, реагирующие на эстроген β-эстрадиол, были построены путем слияния гормонсвязывающего домена рецептора эстрогеначеловека 9-HBD (hER) – с AcrIIA4. Кроме того, было реализовано несколько вентилей ДА и НЕ, которые конститутивно экспрессировали dCas12a(-AD) и AcrVA при индукции с галактозой. В настоящее время эти ворота служат только доказательством концепции. Тем не менее, они также представляют собой первый шаг к глубокому переосмыслению алгоритма для выполнения вычислительного автоматического проектирования цифровых схем синтетических генов в дрожжевых клетках.
Protocol
Representative Results
Discussion
Протокол показал возможный полный рабочий процесс для цифровых схем синтетических генов в соответствии с циклом биологической инженерии «Проектирование-Строительство-Тестирование-Обучение» (DBTL) и в отношении экспериментов как в сухой, так и в мокрой лаборатории. Здесь мы сосредоточи…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Мы хотим поблагодарить всех студентов лаборатории синтетической биологии SPST, TJU за их общую помощь, а также Чжи Ли и Сянъян Чжан за помощь в экспериментах FACS.
Materials
| 0.1 mL PCR 8-strip tubes | NEST | 403112 | |
| 0.2 mL PCR tubes | Axygen | PCR-02-C | |
| 1.5 mL Microtubes | Axygen | MCT-150-C | |
| 15 mL Centrifuge tubes | BIOFIL | CFT011150 | |
| 2 mL Microtubes | Axygen | MCT-200-C | |
| 50 mL Centrifuge tubes | BIOFIL | CFT011500 | |
| Agarose-molecular biology grade | Invitrogen | 75510-019 | |
| Ampicillin sodium salt | Solarbio | 69-52-3 | |
| Applied biosystems veriti 96-well thermal cycler | Thermo Fisher Scientific | 4375786 | |
| AxyPrep DNA gel extraction kit | Axygen | AP-GX-250 | |
| BD FACSuite CS&T research beads | BD | 650621 | Fluorescent beads |
| BD FACSVerse flow cytometer | BD | – | |
| Centrifuge | Eppendorf | 5424 | |
| Centrifuge Sorvall ST 16R | Thermo Fisher Scientific | 75004380 | |
| E. coli competent cells (Strain DH5α) | Life Technologies | 18263-012 | |
| ECL select Western Blotting detection reagent | GE Healthcare | RPN2235 | |
| Electrophoresis apparatus | Beijing JUNYI Electrophoresis Co., Ltd | JY300C | |
| Flat 8-strip caps | NEST | 406012 | |
| Gene synthesis company | Azenta Life Sciences | https://web.azenta.com/zh-cn/azenta-life-sciences | |
| Goat anti-Mouse IgG (H+L) cross-adsorbed secondary antibody Alexa Fluor 568 | Invitrogen | A-11004 | |
| HiFiScript cDNA synthesis kit | CWBIO | CW2569M | Kit used in step 6.2.2.1 |
| Lysate solution (Zymolyase) | zymoresearch | E1004-A | |
| Nikon Eclipse 80i fluorescence microscope | Nikon | – | Fluorescence microscope |
| Pipet tips—10 μL | Axygen | T-300-R-S | |
| Pipet tips—1000 μL | Axygen | T-1000-B-R-S | |
| Pipet tips—200 μL | Axygen | T-200-Y-R-S | |
| pRSII403 | Addgene | 35436 | |
| pRSII404 | Addgene | 35438 | |
| pRSII405 | Addgene | 35440 | |
| pRSII406 | Addgene | 35442 | |
| pRSII424 | Addgene | 35466 | |
| pTPGI_dSpCas9_VP64 | Addgene | 49013 | |
| Q5 High-fidelity DNApolymerase | New England Biolabs | M0491 | |
| Restriction enzyme-Acc65I | New England Biolabs | R0599 | |
| Restriction enzyme-BamHI | New England Biolabs | R0136 | |
| Restriction enzyme-SacI-HF | New England Biolabs | R3156 | |
| Restriction enzyme-XhoI | New England Biolabs | R0146 | |
| Roche LightCycler 96 | Roche | – | Real-Time PCR Instrument |
| S. cerevisiae CEN.PK2-1C | – | – | The parent strain. The genotype is: MATa; his3D1; leu2-3_112; ura3-52; trp1-289; MAL2-8c; SUC2 |
| Stem-Loop Kit | SparkJade | AG0502 | Kit used in step 6.2.1.3 |
| T100 Thermal Cycler | BIO-RAD | 186-1096 | |
| T4 DNA ligase | New England Biolabs | M0202 | |
| T5 Exonuclease | New England Biolabs | M0363 | |
| Taq DNA ligase | New England Biolabs | M0208 | |
| Taq DNA polymerase | New England Biolabs | M0495 | |
| TB Green Premix Ex Taq II (Tli RNaseH Plus)(2x) (SYBR Green I dye) | Takara | RR820Q | |
| YeaStar RNA kit | Zymo Research | R1002 | |
| β-estradiol | Sigma-Aldrich | E8875 |
References
- Marchisio, M. A., Stelling, J. Automatic design of digital synthetic gene circuits. PLOS Computational Biology. 7 (2), 1001083 (2011).
- Karnaugh, M. The map method for synthesis of combinational logic circuits. Transactions of the American Institute of Electrical Engineers. 72 (9), 593-599 (1953).
- Mandell, J. G., Barbas, C. F. Zinc finger tools: custom DNA-binding domains for transcription factors and nucleases. Nucleic Acids Research. 34, 516-523 (2006).
- Bogdanove, A. J., Voytas, D. F. TAL effectors: customizable proteins for DNA targeting. Science. 333 (6051), 1843-1846 (2011).
- Drinnenberg, I. A., et al. RNAi in budding yeast. Science. 326 (5952), 544-550 (2009).
- Gander, M. W., Vrana, J. D., Voje, W. E., Carothers, J. M., Klavins, E. Digital logic circuits in yeast with CRISPR-dCas9 NOR gates. Nature Communications. 8, 15459 (2017).
- Farzadfard, F., Perli, S. D., Lu, T. K. Tunable and multifunctional eukaryotic transcription factors based on CRISPR/Cas. ACS Synthetic Biology. 2 (10), 604-613 (2013).
- Nakamura, M., et al. Anti-CRISPR-mediated control of gene editing and synthetic circuits in eukaryotic cells. Nature Communications. 10 (1), 194 (2019).
- Louvion, J. F., Havaux-Copf, B., Picard, D. Fusion of GAL4-VP16 to a steroid-binding domain provides a tool for gratuitous induction of galactose-responsive genes in yeast. Gene. 131 (1), 129-134 (1993).
- DiCarlo, J. E., et al. Genome engineering in Saccharomyces cerevisiae using CRISPR-Cas systems. Nucleic Acids Research. 41 (7), 4336-4343 (2013).
- Gao, Y., Zhao, Y. Self-processing of ribozyme-flanked RNAs into guide RNAs in vitro and in vivo for CRISPR-mediated genome editing. Journal of Integrative Plant Biology. 56 (4), 343-349 (2014).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Zetsche, B., et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell. 163 (3), 759-771 (2015).
- Fonfara, I., Richter, H., Bratovic, M., Le Rhun, A., Charpentier, E. The CRISPR-associated DNA-cleaving enzyme Cpf1 also processes precursor CRISPR RNA. Nature. 532 (7600), 517-521 (2016).
- Yu, L., Marchisio, M. A. Saccharomyces cerevisiae synthetic transcriptional networks harnessing dCas12a and Type V-A anti-CRISPR proteins. ACS Synthetic Biology. 10 (4), 870-883 (2021).
- Zhang, Y., Marchisio, M. A. Interaction of bare dSpCas9, scaffold gRNA, and type II anti-CRISPR proteins highly favors the control of gene expression in the yeast S. cerevisiae. ACS Synthetic Biology. 11 (1), 176-190 (2022).
- Sheff, M. A., Thorn, K. S. Optimized cassettes for fluorescent protein tagging in Saccharomyces cerevisiae. Yeast. 21 (8), 661-670 (2004).
- Naito, Y., Hino, K., Bono, H., Ui-Tei, K. CRISPRdirect: software for designing CRISPR/Cas guide RNA with reduced off-target sites. Bioinformatics. 31 (7), 1120-1123 (2015).
- Chee, M. K., Haase, S. B. New and redesigned pRS plasmid shuttle vectors for genetic manipulation of Saccharomyces cerevisiae. G3: Genes|Genomes|Genetics. 2 (5), 515-526 (2012).
- Gibson, D. G. Synthesis of DNA fragments in yeast by one-step assembly of overlapping oligonucleotides. Nucleic Acids Research. 37 (20), 6984-6990 (2009).
- Froger, A., Hall, J. E. Transformation of plasmid DNA into E. coli using the heat shock method. Journal of Visualized Experiments. (6), e253 (2007).
- Green, M. R., Sambrook, J. . Molecular Cloning. Fourth edition. , (2012).
- Sanger, F. Determination of nucleotide sequences in DNA. Science. 214 (4526), 1205-1210 (1981).
- Gietz, R. D., Woods, R. A. Transformation of yeast by lithium acetate/single-stranded carrier DNA/polyethylene glycol method. Methods in Enzymology. 350, 87-96 (2002).
- Zalatan, J. G., et al. Engineering complex synthetic transcriptional programs with CRISPR RNA scaffolds. Cell. 160 (1-2), 339-350 (2015).
- Song, W., Li, J., Liang, Q., Marchisio, M. A. Can terminators be used as insulators into yeast synthetic gene circuits. Journal of Biological Engineering. 10, 19 (2016).
- Rauch, B. J., et al. Inhibition of CRISPR-Cas9 with bacteriophage proteins. Cell. 168 (1-2), 150-158 (2017).
- Hynes, A. P., et al. An anti-CRISPR from a virulent streptococcal phage inhibits Streptococcus pyogenes Cas9. Nature Microbiology. 2 (10), 1374-1380 (2017).
- Watters, K. E., Fellmann, C., Bai, H. B., Ren, S. M., Doudna, J. A. Systematic discovery of natural CRISPR-Cas12a inhibitors. Science. 362 (6411), 236-239 (2018).
- Chen, C., et al. Real-time quantification of microRNAs by stem-loop RT-PCR. Nucleic Acids Research. 33 (20), 179 (2005).
- Pfaffl, M. W. A new mathematical model for relative quantification in real-time RT-PCR. Nucleic Acids Research. 29 (9), 45 (2001).
- Hahne, F., et al. flowCore: a Bioconductor package for high throughput flow cytometry. BMC Bioinformatics. 10 (1), 106 (2009).
- Li, J., Xu, Z., Chupalov, A., Marchisio, M. A. Anti-CRISPR-based biosensors in the yeast S. cerevisiae. Journal of Biological Engineering. 12, 11 (2018).
- Dong, L., et al. An anti-CRISPR protein disables type V Cas12a by acetylation. Nature Structural & Molecular Biology. 26 (4), 308-314 (2019).

