Imitando la función de las proteínas de señalización: hacia artificial de la señal de transducción Terapia
Summary
We present guidelines for developing synthetic ‘chemical transducers’ that can induce communication between naturally unrelated proteins. In addition, detailed protocols are presented for synthesizing and testing a specific ‘transducer’ that enables a growth factor to activate a detoxifying enzyme and consequently, to regulate the cleavage of an anticancer prodrug.
Abstract
Signal transduction pathways, which control the response of cells to various environmental signals, are mediated by the function of signaling proteins that interact with each other and activate one other with high specificity. Synthetic agents that mimic the function of these proteins might therefore be used to generate unnatural signal transduction steps and consequently, alter the cell’s function. We present guidelines for designing ‘chemical transducers’ that can induce artificial communication between native proteins. In addition, we present detailed protocols for synthesizing and testing a specific ‘transducer’, which can induce communication between two unrelated proteins: platelet-derived growth-factor (PDGF) and glutathione-S-transferase (GST). The way by which this unnatural PDGF-GST communication could be used to control the cleavage of an anticancer prodrug is also presented, indicating the potential for using such systems in ‘artificial signal transduction therapy’. This work is intended to facilitate developing additional ‘transducers’ of this class, which may be used to mediate intracellular protein-protein communication and consequently, to induce artificial cell signaling pathways.
Introduction
Las vías de transducción de señal desempeñan un papel importante en prácticamente todos los procesos celular y permiten a la célula responder rápidamente a las señales ambientales. 1 Estas vías son a menudo desencadenan por la unión de una molécula de señalización a un receptor extracelular, lo que resulta en la activación de enzimas intracelulares. La amplificación y la propagación de esta señal dentro de la célula está mediada por la función de las proteínas que forman una red de interacciones proteína-proteína en el que las enzimas se activan de forma reversible con una alta especificidad de señalización. Debido a la desregulación de estas redes con frecuencia conduce al desarrollo del cáncer, no ha habido mucho interés en el establecimiento de "terapia de transducción de señales de cáncer ', 2 por el que los medicamentos están diseñados para interrumpir las rutas de señalización malignas. Recientemente hemos propuesto un enfoque alternativo a la señal de transducción de la terapia que se basa en la capacidad de los fármacos para generar vías de transducción de señales no naturales. <sup> 3 En particular, creemos que mediante el diseño de agentes sintéticos que imitan la función de las proteínas de señalización, sería posible para modular la función de la célula indirectamente. Por ejemplo, estas redes artificiales pueden permitir biomarcadores de proteínas para activar las enzimas que escinden profármacos. Alternativamente, estos miméticos de proteína de señalización podrían ser capaces de activar las vías de señalización celular no naturales, dando como resultado efectos terapéuticos.
Para demostrar la viabilidad de este enfoque, hemos creado recientemente un "transductor química 'sintética 4 que permite el factor de crecimiento derivado de plaquetas (PDGF) para activar la escisión de un profármaco anticáncer mediante la activación de la glutatión-s-transferasa (GST), que es no su pareja de unión natural. La estructura de este "transductor" consiste en un anti-PDGF aptámero de ADN que se ha modificado con un inhibidor bivalente para GST. Por lo tanto, este agente sintético pertenece a una familia de moléculas con sitios de unión adiferentes proteínas, 5-7, tales como inductores químicos de dimerización (CID) 8-10 y también para el grupo de proteínas aglutinantes sobre la base de los conjugados de molécula de oligonucleótido sintético. 11-21
Los principios generales que subyacen en el diseño de tales sistemas se describe en este documento y se proporcionan protocolos detallados para sintetizar y probar la función de este "transductor" con ensayos enzimáticos convencionales. Este trabajo tiene por objeto facilitar el desarrollo de transductores '' adicionales de esta clase, que se puede utilizar para mediar en la comunicación proteína-proteína intracelular y, por consiguiente, para inducir la vías de señalización celular artificiales.
La Figura 1 describe esquemáticamente los principios de funcionamiento de los transductores '' químicos sintéticos que pueden mediar comunicaciones proteína-proteína no naturales. En esta ilustración, un "transductor química ', que integra aglutinantes sintéticos para proteins I y II (aglutinantes I y II), permite a la proteína II para desencadenar la actividad catalítica de la proteína I, que no es su pareja de unión natural. En la ausencia de la proteína II, el transductor se une al sitio catalítico de la enzima (proteína I) e inhibe su actividad (Figura 1, el estado ii). La unión de la "transductor" a la proteína II, sin embargo, promueve interacciones entre aglutinante I y la superficie de la proteína II (Figura 1, el estado iii), lo que reduce su afinidad hacia la proteína I. Como resultado, la concentración eficaz de la " transductor libre 'en la solución se reduce, lo que conduce a la disociación del transductor-proteína complejo I y para la reactivación de la proteína I (Figura 1, el estado iv). Tomados en conjunto, estos pasos destacar tres principios fundamentales que subyacen en el diseño de transductores '' eficientes: (1) un "transductor" debe tener una carpeta específica para cada una de las proteínas diana, (2) la interacción between ligante II y II de la proteína debe ser más fuerte que la interacción entre la proteína ligante I y I, y (3) ligante debo ser capaz de interactuar con la superficie de la proteína II. Este último principio no requiere necesariamente que ligante yo solo tendría una alta afinidad y selectividad para la proteína II. En su lugar, se basa en nuestros estudios recientes que mostraron que traer una molécula sintética en la proximidad de una proteína puede promover interacciones entre esta molécula y la superficie de la proteína. 19,22,23
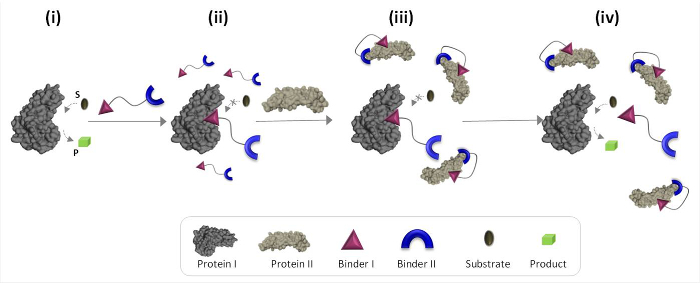
Figura 1:. Operando principios de transductores químicos '' Cuando se añade el 'transductor química' a una proteína activa I (estado i), se une a su sitio activo a través de aglutinante que e inhibe su actividad (estado II). En presencia de la proteína II, sin embargo, el no unido 'química transducer 'interactúa con la proteína II a través de aglutinante II, que promueve las interacciones entre aglutinante I y la superficie de la proteína II. Esta carpeta inducida I-II de la proteína de interacción reduce la concentración efectiva de aglutinante I, que conduce a la disociación de la 'transducer'-proteína complejo I y para la proteína I reactivación (estado IV). Haga clic aquí para ver una versión más grande de esta figura .
Protocol
Representative Results
Discussion
We presented a method for designing and testing of a ‘chemical transducer’ that can induce artificial communication between two naturally unrelated proteins, GST and PDGF, without modifying the native proteins. The unnatural GST-PDGF communications could be detected in real time by using enzymatic assays that follow the changes in the activity of GST in the presence of the ‘chemical transducer’ and increasing the concentrations of PDGF. In addition to detecting the activation of GST by PDGF, these assays were used to fol…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Esta investigación fue apoyada por la Fundación Minerva, la Organización HFSP, y un Consejo de Investigación Europeo de Grant (Starting Grant 338265).
Materials
| 1-chloro-2,4-dinitrobenzene | Sigma-Aldrich | 237329 | |
| Acetic acid | Bio Lab | 01070521 | |
| Acetnitrile | J.T.Baker | 9017-03 | |
| Ascorbic acid | Sigma-Aldrich | A4544 | |
| Copper(II) Sulfate pentahydrate | Merck-Millipore | 102790 | |
| Dimethyl sulfoxide | Merck-Millipore | 802912 | |
| Dulbecco's Phosphate Buffered Saline | Biological Industries | 02-023-5A | |
| Ethacrynic acid | Tokyo Chemical Industry Co. Ltd | E0526 | |
| Glutathione-s-transferase M1-1 | Israel Structural Proteomics Center (Weizmann Institute of Science, Rehovot, Israel) | ||
| JS-K | Sigma-Aldrich | J4137 | |
| L-glutathione reduced | Sigma-Aldrich | G4251 | |
| Magnesium Chloride | J.T.Baker | 0162 | |
| nitrate/nitrite colorimetric assay kit | Cayman Chemical | 780001 | |
| Oligonucleotides | W. M. Keck Foundation Biotechnology at Yale University | custom order | |
| PDGF-BB | Israel Structural Proteomics Center (Weizmann Institute of Science, Rehovot, Israel) | ||
| TBTA | Sigma-Aldrich | 678937 | |
| Triethylamine | Sigma-Aldrich | T0886 | |
| Desalting column | GE Healthcare | illustra MicroSpin G-25 Columns | |
| HPLC | Waters | 2695 separation module | |
| HPLC column | Waters | XBridgeTM OST C18 column (2.5 μM, 4.6 mm × 50 mm) | |
| HPLC column | Waters | XBridgeTM OST C18 column (2.5μM, 10 mm × 50 mm) | |
| Plate reader | BioTek | synergy H4 hybrid |
References
- Hunter, T. Signaling—2000 and Beyond. Cell. 100, 113-127 (2000).
- Levitzki, A., Klein, S. Signal transduction therapy of cancer. Mol Aspects Med. 31, 287-329 (2010).
- Peri-Naor, R., Motiei, L., Margulies, D. Artificial signal transduction therapy: a futuristic approach to disease treatment. Future Med. Chem. 7, 2091-2093 (2015).
- Peri-Naor, R., Ilani, T., Motiei, L., Margulies, D. Protein-Protein Communication and Enzyme Activation Mediated by a Synthetic Chemical Transducer. J. Am. Chem. Soc. 137, 9507-9510 (2015).
- Corson, T. W., Aberle, N., Crews, C. M. Design and Applications of Bifunctional Small Molecules: Why Two Heads Are Better Than One. ACS Chem. Biol. 3, 677-692 (2008).
- Rutkowska, A., Schultz, C. Protein Tango: The Toolbox to Capture Interacting Partners. Angew. Chem. Int. Ed. 51, 8166-8176 (2012).
- Meyer, C., Köhn, M. A Molecular Tête-à-Tête Arranged by a Designed Adaptor Protein. Angew. Chem. Int. Ed. 51, 8160-8162 (2012).
- Klemm, J. D., Schreiber, S. L., Crabtree, G. R. Dimerization as a Regulatory Mechanism in Signal Transduction. Annu. Rev. Immunol. 16, 569-592 (1998).
- DeRose, R., Miyamoto, T., Inoue, T. Manipulating signaling at will: chemically-inducible dimerization (CID) techniques resolve problems in cell biology. Pflugers Arch. 465, 409-417 (2013).
- Gestwicki, J. E., Marinec, P. S. Chemical control over protein-protein interactions: beyond inhibitors. Comb. Chem. High Throughput. Screen. 10, 667-675 (2007).
- Battle, C., Chu, X., Jayawickramarajah, J. Oligonucleotide-based systems for input-controlled and non-covalently regulated protein binding. Supramol. Chem. 25, 848-862 (2013).
- Diezmann, F., Seitz, O. DNA-guided display of proteins and protein ligands for the interrogation of biology. Chem. Soc. Rev. 40, 5789-5801 (2011).
- Röglin, L., Ahmadian, M. R., Seitz, O. DNA-Controlled Reversible Switching of Peptide Conformation and Bioactivity. Angew. Chem. Int. Ed. 46, 2704-2707 (2007).
- Röglin, L., Altenbrunn, F., Seitz, O. DNA and RNA-Controlled Switching of Protein Kinase Activity. ChemBioChem. 10, 758-765 (2009).
- Harris, D. C., Chu, X., Jayawickramarajah, J. DNA-Small Molecule Chimera with Responsive Protein-Binding Ability. J. Am. Chem. Soc. 130, 14950-14951 (2008).
- Harris, D. C., Saks, B. R., Jayawickramarajah, J. Protein-Binding Molecular Switches via Host-Guest Stabilized DNA Hairpins. J. Am. Chem. Soc. 133, 7676-7679 (2011).
- Kim, Y., Cao, Z., Tan, W. Molecular assembly for high-performance bivalent nucleic acid inhibitor. Proc. Nat. Acad. Sci. U.S.A. 105, 5664-5669 (2008).
- Han, D., et al. A Logical Molecular Circuit for Programmable and Autonomous Regulation of Protein Activity Using DNA Aptamer-Protein Interactions. J. Am. Chem. Soc. 134, 20797-20804 (2012).
- Motiei, L., Pode, Z., Koganitsky, A., Margulies, D. Targeted Protein Surface Sensors as a Tool for Analyzing Small Populations of Proteins in Biological Mixtures. Angew. Chem. Int. Ed. 53, 9289-9293 (2014).
- Ranallo, S., Rossetti, M., Plaxco, K. W., Vallée-Bélisle, A., Ricci, F. A Modular, DNA-Based Beacon for Single-Step Fluorescence Detection of Antibodies and Other Proteins. Angew. Chem. Int. Ed. 54, 13214-13218 (2015).
- Franzini, R. M., et al. Identification of Structure-Activity Relationships from Screening a Structurally Compact DNA-Encoded Chemical Library. Angew. Chem. Int. Ed. 54, 3927-3931 (2015).
- Unger-Angel, L., et al. Protein recognition by bivalent, ‘turn-on’ fluorescent molecular probes. Chem. Sci. 6, 5419-5425 (2015).
- Nissinkorn, Y., et al. Sensing Protein Surfaces with Targeted Fluorescent Receptors. Chem. Eur. J. 21, 15981-15987 (2015).
- Huber, C. G., Oefner, P. J., Bonn, G. K. High-Resolution Liquid Chromatography of Oligonucleotides on Nonporous Alkylated Styrene-Divinylbenzene Copolymers. Anal. Biochem. 212, 351-358 (1993).
- Lyon, R. P., Hill, J. J., Atkins, W. M. Novel class of bivalent glutathione S-transferase inhibitors. Biochemistry. 42, 10418-10428 (2003).
- Battle, C., Chu, X., Jayawickramarajah, J. Oligonucleotide-based systems for input-controlled and non-covalently regulated protein binding. Supramol. Chem. , 1-16 (2013).

