Application de la microscopie vitesse haut débit Super-résolution en direct cil primaire
Summary
Récemment, nous avons cartographié les emplacements de spatiales en trois dimensions (3D) de voies de transport pour différentes protéines translocation à l’intérieur des cils primaires dans des cellules vivantes. Ici ce détails papier expérimental, le processus d’échantillons biologiques et les analyses de données pour la fluorescence 3D Super-resolution imaging approche nouvellement appliqués en live cils primaires.
Abstract
Le cil primaire est une saillie axée sur les microtubules sur la surface de nombreuses cellules eucaryotes et contient un complément unique de protéines cette fonction critique dans la motilité cellulaire et signalisation. Étant donné que les cils sont incapables de synthétiser ses propres protéines, près de 200 protéines ciliaires uniques doivent être victimes de la traite entre le cytosol et les cils primaires. Toutefois, il est toujours un défi technique pour mapper des lieux (3D) en trois dimensions des voies de transport pour ces protéines en direct cils primaires en raison des limites existant actuellement techniques. Pour conquérir le défi, récemment nous avons développé et utilisé une microscopie haute vitesse super-résolution 3D virtuel, appelée la microscopie monopoint bord-excitation auxiliaire diffraction (vitesse), pour déterminer l’emplacement dans l’espace 3D de voies de transport pour les deux cytosolique et les protéines membranaires des cils primaires de cellules vivantes. Dans cet article, nous allons démontrer la configuration détaillée de la microscopie de vitesse, la préparation des cellules exprimant les protéines ciliaires fluorescence-protéine-marquées, le suivi en temps réel de la molécule unique de protéines individuelles en direct CIL et la réalisation de densité de probabilité spatiale 3D cartes des itinéraires de transport pour les protéines ciliaires.
Introduction
Puisqu’a déclaré Ernst Abbe en 1873, la résolution de microscopie optique conventionnelle a cru être limitée à environ 200 nm en raison de la diffraction de la lumière de l’objectif1,2. Actuellement, les techniques de microscopie photonique Super-résolution briser cette limitation et permettent la capture d’images dynamiques avec résolution diffraction subsidiaire (< 200 nm). Les techniques généralement entrent dans deux grandes catégories : stimulée par des approches d’émission épuisement (STED) microscopie basée, qui génèrent des diffraction sous illumination volume en raison de la réponse optique non linéaire de fluorophores à échantillons3; et microscopie photonique photoactivation (PALM) et microscopie de reconstruction optique stochastique (tempête)-techniques de Super-résolution, qui utilisent des fonctions mathématiques pour localiser le centre de gravité des fluorophores et ensuite reconstituer ces centroïdes basées sur pour former la Super-résolution images4,5. Actuellement, en raison de la configuration optique relativement uncomplicated, PALM et tempête beaucoup travaillent en activant uniquement un petit sous-ensemble des fluorophores dans chaque image d’une vidéo longue d’une préparation biologique. Cela permet la localisation plus précise en 2D raccord gaussienne de la tache fluorescente, appelé la fonction de point propagation (PSF), de protéines marquées fluorescent dans chaque image de la vidéo. L’emplacement 2D de chaque molécule marquée fluorescent peut ensuite être superposé sur un seul plan d’imagerie pour produire une image de Super-résolution de la préparation biologique1,2. Tandis que ces molécules simples de localisation, Super-résolution approches à la microscopie certainement a révolutionné la mode d’exécution de l’imagerie d’échantillons biologiques, il y a encore des défis à surmonter. Par exemple, STORM et PALM peuvent réaliser leurs meilleures résolutions spatiales après fixation d’échantillons biologiques et ainsi présenter une représentation statique des protéines fluorescent marqué, qui est une restriction semblable de la microscopie électronique. En outre, pour parvenir à une résolution spatiale élevée pour chaque protéine fluorescent marqué dans des cellules vivantes, des échantillons doivent être copiés à des framerates très longs qui sont incapables de saisir la dynamique des protéines. Par conséquent, il est nécessaire pour surmonter ces obstacles techniques principales.
Pour obtenir une haute résolution spatio-temporelle qui est bien adaptée pour détecter les protéines rapides ou ARN dans des cellules vivantes, nous avons développé la microscopie de Super-résolution vitesse dans notre laboratoire (Figure 1)6,,7, 8. plusieurs avancées techniques majeures en microscopie de vitesse précédemment nous ont permis de suivre avec succès les transport nucléocytoplasmique de petites molécules, protéines, ADN messagère et virus par native nucléaire pore complexes (NPC)6, 7 , 8. en bref, les fonctionnalités suivantes de microscopie de vitesse seront utilisées pour suivre les macromolécules rapides par le biais de structures cône d’éclairage symétrique sous micromètre dans des cellules vivantes, telles que les PNJ et les cils primaires : (1) une tendance ou un l’éclairage vertical PSF permet l’excitation des molécules simples dans un volume de la petite limite de diffraction dans le plan focal (Figure 1) ; (2), les fibres discontinues de polyesters incliné peut éviter grandement fluorescence out-of-focus et donc d’améliorer le rapport signal-bruit. (3), la densité optique de 100 à 500 kW / cm2 dans l’illumination PSF permet à des milliers de photons à collecter des fluorophores unique avec des vitesses de détection rapide (> 500 Hz). Vitesse de détection (4), le jeûne réduit aussi considérablement l’erreur de localisation spatiale de molécules simples (< 10 nm) pour déterminer les trajectoires spatiales des molécules fluorescentes en mouvement dans des cellules vivantes, car la diffusion moléculaire est l’un des principaux facteurs imperfections de la localisation de la molécule unique pour le déplacement des molécules à l’origine. Algorithmes de transformation (5) bien établie 2D à la 3D pour nous permettent de fournir des cartes de densité de probabilité spatiale 3D des itinéraires de transport pour les molécules dans le NPC ou cil primaire. Il convient de noter que notre processus de conversion entre le cartésien et le système de coordination cylindrique est utilisé pour générer un suivi de molécules simples densité de probabilité spatiale 3D carte plutôt que 3D (Figure 2). Auparavant, la microscopie électronique données ont révélé que le NPC9,10 et le cil primaire11 tous les deux ont une structure par rotation symétrique. En principe, diffusant au hasard des molécules se déplaçant à travers le NPC ou cil primaire devrait également avoir des distributions de cône d’éclairage symétrique. Comme illustré à la Figure 2, un nombre élevé de diffusion au hasard des molécules à l’intérieur de la bouteille générerait des distributions cône d’éclairage symétrique à la vue de coupe transversale que celle dans le NPC, encore ayant pour résultat une sensiblement uniforme spatiale distribution au sein de chaque sous-région infime entre deux anneaux voisins (Figure 2E). Cette répartition uniforme entraîne que la distribution spatiale le long de la dimension θ dans le système cylindrique est constante. Puis les coordonnées 3D (R, X, θ) peuvent être simplifiées pour les coordonnées 2D (R, X, constante). En fait, notre processus de conversion entre le cartésien et les systèmes cylindriques est de la 2D (X, Y) en 2D (R, X, constante). La constante θ, fait référence à la densité spatiale p dans la Figure 2E, est calculée en utilisant l’équation A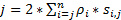 .
.
En fin de compte, suivi de la molécule unique a large application dans la recherche biologique, il est donc naturel qu’une pléthore de techniques est développée pour remplir les niches biologiques spécifiques12,13,14. Tel est le cas avec la microscopie de vitesse. Auparavant, lorsqu’il est couplé avec un algorithme de transformation 3D, cette technique a été développée pour résoudre les voies de transport 3D de molécules à travers les PNJ, une symétrie de révolution et sous-sous-diffraction-taille de la structure biologique6en transit. Dans cet article, cils primaires sont avérés organites excellent modèle aussi bien. Cils primaires sont des organites cylindriques, antenne-like (rayon de ~ 125 nm) qui projettent de la surface des cellules mammifères plus15,16,17. Ils sont responsables de recevoir des signaux externes et de transmettre une réponse intracellulaire, généralement associée à la croissance et le métabolisme15,16. Par conséquent, du flux des protéines structurales, recyclage des récepteurs transmembranaires et la transmission des messagers intracellulaires est des responsabilités vitales des cils primaires. À la jonction entre les cils primaires et le corps cellulaire est une barrière de sélectivité critique, appelée zone de transition ou TZ, grâce auquel tous les transports de cette protéine doivent avoir lieu à11,18,19, 20. En plus de la fonction blocage de la TZ, au moins deux processus de transport, intraflagellaire transport et diffusion passive, sont considérés comme responsables de la circulation des protéines à travers cette région16,,21, 22. du point de vue de la santé humaine, la perte des cils primaires et déréglementation ultérieure de la signalisation en aval est caractéristique de nombreux cancers. En outre, beaucoup de maladies génétiques, comme le syndrome de Bardet-Biedl et maladie polykystique des reins, est associés à la protéine défectueuse transport23. La taille limite de diffraction sous tant le processus complexe de transport des protéines sélectif par le biais de la TZ faire les cils primaires une cible de choix pour cette technique. Dans cet article des méthodes, nous allons démontrer le suivi d’une protéine transmembranaire ciliaire, récepteurs de la somatostatine 3 (SSTR3)24, marquées extérieurement à Alexa Fluor 647 ainsi qu’une composante de l’IFT, IFT2025, marquée avec une molécule GFP fusionnée.
Protocol
Representative Results
Discussion
Ce protocole décrit l’application de la microscopie de vitesse pour le cil primaire, un organite cellulaire de signalisation qui est fortement tributaire de transport des protéines efficaces. Microscopie de vitesse peut fournir des emplacements (< 10 nm) pour des molécules marquées fluorescent haute résolution lorsqu’ils traversent l’éclairage seul point centré sur le TZ. Auparavant, il a été appliqué à l’étude de la protéine le trafic à travers les NPC6,<sup class…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Nous remercions le Dr Kristen Verhey (University of Michigan, Ann Arbor) et Dr Gregory Pazour (University of Massachusetts Medical School) pour fournir certains plasmides. Le projet a été soutenu par des subventions de la National Institutes of Health (NIH GM097037, GM116204 et GM122552 à W.Y.).
Materials
| 25 cm2 tissue culture dish | Corning | VV-01936-00 | |
| Penicillin/streptomycin | ThermoFisher | 15140122 | |
| Fetal bovine serum | ThermoFisher | 10438018 | |
| DMEM | ThermoFisher | 10566-016 | |
| OPTIMEM | ThermoFisher | 31985062 | |
| Trypsin | ThermoFisher | 25300054 | |
| Phosphate buffered saline | Sigma-Aldrich | P3813-1PAK | |
| Transit LT1 | Mirus | MIR 2300 | |
| 35 mm glass bottom dish | MatTek | P35GCOL-0-14-C | |
| AlexaFluor 647-conjugated streptavidin | ThermoFisher | S21374 | |
| Biotin | Sigma-Aldrich | B4501-100MG | |
| 633 nm He-Ne laser | Melles Griot | 25-LHP-928-249 | |
| 561 nm solid state laser | Coherent | OBIS 561-50 LS | |
| 488 nm solid state laser | Coherent | 1185053 | |
| Inverted fluorescence microscope | Olympus | IX81 | |
| 1.4-NA 100× oil-immersion apochromatic objective | Olympus | UPLSAPO 100× | |
| On-chip multiplication gain charge-coupled-device camera | Roper Scientific | Cascade 128+ | |
| Dichroic filter | Semrock | Di01- R405/488/561/635-25×36 | |
| Emission filter | Semrock | NF01-405/488/561/635-25X5.0 | |
| Slidebook 6.0 | Intelligent Imaging Innovations | digital microscopy software |
References
- Huang, B., Bates, M., Zhuang, X. Super-resolution fluorescence microscopy. Annu Rev Biochem. 78, 993-1016 (2009).
- Leung, B. O., Chou, K. C. Review of super-resolution fluorescence microscopy for biology. Appl Spectrosc. 65, 967-980 (2011).
- Willig, K. I., Rizzoli, S. O., Westphal, V., Jahn, R., Hell, S. W. STED microscopy reveals that synaptotagmin remains clustered after synaptic vesicle exocytosis. Nature. 440, 935-939 (2006).
- Betzig, E., et al. Imaging intracellular fluorescent proteins at nanometer resolution. Science. 313, 1642-1645 (2006).
- Rust, M. J., Bates, M., Zhuang, X. Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy (STORM). Nat Meth. 3, 793-796 (2006).
- Ma, J., Yang, W. Three-dimensional distribution of transient interactions in the nuclear pore complex obtained from single-molecule snapshots. Proc Natl Acad Sci USA. 107, 7305-7310 (2010).
- Ma, J., Goryaynov, A., Sarma, A., Yang, W. Self-regulated viscous channel in the nuclear pore complex. Proc Natl Acad Sci USA. 109, 7326-7331 (2012).
- Ma, J., et al. High-resolution three-dimensional mapping of mRNA export through the nuclear pore. Nat Comm. 4, (2013).
- Akey, C. W., Radermacher, M. Architecture of the Xenopus nuclear pore complex revealed by three-dimensional cryo-electron microscopy. J Cell Biol. 122, 1-19 (1993).
- Akey, C. W. Interactions and structure of the nuclear pore complex revealed by cryo-electron microscopy. J Cell Biol. 109, 955-970 (1989).
- Czarnecki, P. G., Shah, J. V. The ciliary transition zone: from morphology and molecules to medicine. Trends Cell Biol. 22, 201-210 (2012).
- Elf, J., Li, G. -. W., Xie, X. S. Probing transcription factor dynamics at the single-molecule level in a living cell. Science. 316, 1191-1194 (2007).
- Anzalone, A., Annibale, P., Gratton, E. 3D orbital tracking in a modified two-photon microscope: an application to the tracking of intracellular vesicles. J Vis Exp. , (2014).
- Ritter, J. G., Veith, R., Veenendaal, A., Siebrasse, J. P., Kubitscheck, U. Light sheet microscopy for single molecule tracking in living tissue. PloS one. 5, 11639 (2010).
- Marshall, W. F., Nonaka, S. Cilia: tuning in to the cell’s antenna. Curr Biol. 16, 604-614 (2006).
- Scholey, J. M., Anderson, K. V. Intraflagellar transport and cilium-based signaling. Cell. 125, 439-442 (2006).
- Yang, T. T., et al. Superresolution pattern recognition reveals the architectural map of the ciliary transition zone. Sci Rep. 5, 14096 (2015).
- Craige, B., et al. CEP290 tethers flagellar transition zone microtubules to the membrane and regulates flagellar protein content. J Cell Biol. 190, 927-940 (2010).
- Kee, H. L., et al. A size-exclusion permeability barrier and nucleoporins characterize a ciliary pore complex that regulates transport into cilia. Nat Cell Biol. 14, 431-437 (2012).
- Najafi, M., Maza, N. A., Calvert, P. D. Steric volume exclusion sets soluble protein concentrations in photoreceptor sensory cilia. Proc Natl Acad Sci USA. 109, 203-208 (2012).
- Nachury, M. V., Seeley, E. S., Jin, H. Trafficking to the ciliary membrane: how to get across the periciliary diffusion barrier. Annu Rev Cell Dev Biol. 26, 59-87 (2010).
- Ye, F., et al. Single molecule imaging reveals a major role for diffusion in the exploration of ciliary space by signaling receptors. Elife. 2, 00654 (2013).
- Ross, A. J., et al. Disruption of Bardet-Biedl syndrome ciliary proteins perturbs planar cell polarity in vertebrates. Nat Genetics. 37, 1135-1140 (2005).
- Handel, M., et al. Selective targeting of somatostatin receptor 3 to neuronal cilia. Neuroscience. 89, 909-926 (1999).
- Follit, J. A., Tuft, R. A., Fogarty, K. E., Pazour, G. J. The intraflagellar transport protein IFT20 is associated with the Golgi complex and is required for cilia assembly. Mol Biol Cell. 17, 3781-3792 (2006).
- Awata, J., et al. NPHP4 controls ciliary trafficking of membrane proteins and large soluble proteins at the transition zone. J Cell Sci. 127, 4714-4727 (2014).
- Howarth, M., Ting, A. Y. Imaging proteins in live mammalian cells with biotin ligase and monovalent streptavidin. Nat Protoc. 3, 534-545 (2008).
- Huang, B., Wang, W., Bates, M., Zhuang, X. Three-dimensional super-resolution imaging by stochastic optical reconstruction microscopy. Science. 319, 810-813 (2008).

